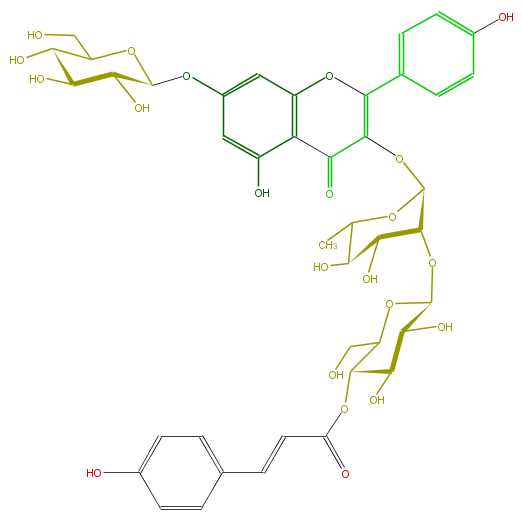
Mol:FL5FAAGS0130
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 64 70 0 0 0 0 0 0 0 0999 V2000 | + | 64 70 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.7917 4.5391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7917 4.5391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7917 3.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7917 3.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5060 3.3016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5060 3.3016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2205 3.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2205 3.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2205 4.5391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2205 4.5391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5060 4.9516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5060 4.9516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0772 3.3016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0772 3.3016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3627 3.7141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3627 3.7141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6483 3.3016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6483 3.3016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6483 2.4766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6483 2.4766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3627 2.0642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3627 2.0642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0772 2.4766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0772 2.4766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0661 3.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0661 3.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7806 3.3016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7806 3.3016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7806 2.4766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7806 2.4766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0661 2.0642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0661 2.0642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3627 1.3459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3627 1.3459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4951 3.7141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4951 3.7141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7548 2.0419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7548 2.0419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8212 4.8859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8212 4.8859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0661 1.3646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0661 1.3646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1950 4.2390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1950 4.2390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7824 3.5243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7824 3.5243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9842 3.7334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9842 3.7334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1907 3.5066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1907 3.5066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6032 4.2213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6032 4.2213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4015 4.0122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4015 4.0122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7428 4.4935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7428 4.4935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4688 4.5217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4688 4.5217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8212 4.0225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8212 4.0225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4154 3.6581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4154 3.6581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4529 3.0715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4529 3.0715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8055 0.7578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8055 0.7578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7286 -0.0638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7286 -0.0638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3552 0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3552 0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1680 0.6161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1680 0.6161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2450 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2450 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6183 0.9007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6183 0.9007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4171 -0.0232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4171 -0.0232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0948 -0.3504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0948 -0.3504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2524 -0.0991 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2524 -0.0991 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2159 0.3314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2159 0.3314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7655 -2.2239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7655 -2.2239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5907 -2.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5907 -2.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8039 -1.4216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8039 -1.4216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3936 -0.8444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3936 -0.8444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5684 -0.8492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5684 -0.8492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3550 -1.6465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3550 -1.6465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7697 -1.7213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7697 -1.7213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4167 -2.2708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4167 -2.2708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2288 -2.7721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2288 -2.7721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6066 -1.3111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6066 -1.3111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6501 -2.9553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6501 -2.9553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2588 -3.5236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2588 -3.5236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6747 -4.2438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6747 -4.2438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4349 -3.5236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4349 -3.5236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0230 -4.2371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0230 -4.2371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8008 -4.2371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8008 -4.2371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2133 -3.5226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2133 -3.5226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0383 -3.5226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0383 -3.5226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4508 -4.2371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4508 -4.2371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0383 -4.9516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0383 -4.9516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2133 -4.9516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2133 -4.9516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2747 -4.2371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2747 -4.2371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | 37 19 1 0 0 0 0 | + | 37 19 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 44 51 1 0 0 0 0 | + | 44 51 1 0 0 0 0 |
| − | 45 52 1 0 0 0 0 | + | 45 52 1 0 0 0 0 |
| − | 46 39 1 0 0 0 0 | + | 46 39 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 54 56 1 0 0 0 0 | + | 54 56 1 0 0 0 0 |
| − | 56 57 2 0 0 0 0 | + | 56 57 2 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 2 0 0 0 0 | + | 58 59 2 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 61 2 0 0 0 0 | + | 60 61 2 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 62 63 2 0 0 0 0 | + | 62 63 2 0 0 0 0 |
| − | 63 58 1 0 0 0 0 | + | 63 58 1 0 0 0 0 |
| − | 61 64 1 0 0 0 0 | + | 61 64 1 0 0 0 0 |
| − | 53 43 1 0 0 0 0 | + | 53 43 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGS0130 | + | ID FL5FAAGS0130 |
| − | FORMULA C42H46O22 | + | FORMULA C42H46O22 |
| − | EXACTMASS 902.248073156 | + | EXACTMASS 902.248073156 |
| − | AVERAGEMASS 902.80144 | + | AVERAGEMASS 902.80144 |
| − | SMILES C(O3)(c(c7)ccc(c7)O)=C(OC(C(OC(C(O)5)OC(C(OC(=O)C=Cc(c6)ccc(O)c6)C5O)CO)4)OC(C(O)C4O)C)C(c(c23)c(O)cc(c2)OC(O1)C(O)C(O)C(C1CO)O)=O | + | SMILES C(O3)(c(c7)ccc(c7)O)=C(OC(C(OC(C(O)5)OC(C(OC(=O)C=Cc(c6)ccc(O)c6)C5O)CO)4)OC(C(O)C4O)C)C(c(c23)c(O)cc(c2)OC(O1)C(O)C(O)C(C1CO)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
64 70 0 0 0 0 0 0 0 0999 V2000
2.7917 4.5391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7917 3.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5060 3.3016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2205 3.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2205 4.5391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5060 4.9516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0772 3.3016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3627 3.7141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6483 3.3016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6483 2.4766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3627 2.0642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0772 2.4766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0661 3.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7806 3.3016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7806 2.4766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0661 2.0642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3627 1.3459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4951 3.7141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7548 2.0419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8212 4.8859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0661 1.3646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1950 4.2390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7824 3.5243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9842 3.7334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1907 3.5066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6032 4.2213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4015 4.0122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7428 4.4935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4688 4.5217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8212 4.0225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4154 3.6581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4529 3.0715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8055 0.7578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7286 -0.0638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3552 0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1680 0.6161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2450 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6183 0.9007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4171 -0.0232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0948 -0.3504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2524 -0.0991 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2159 0.3314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7655 -2.2239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5907 -2.2189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8039 -1.4216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3936 -0.8444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5684 -0.8492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3550 -1.6465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7697 -1.7213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4167 -2.2708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2288 -2.7721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6066 -1.3111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6501 -2.9553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2588 -3.5236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6747 -4.2438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4349 -3.5236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0230 -4.2371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8008 -4.2371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2133 -3.5226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0383 -3.5226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4508 -4.2371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0383 -4.9516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2133 -4.9516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2747 -4.2371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 18 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
36 39 1 0 0 0 0
35 40 1 0 0 0 0
34 41 1 0 0 0 0
33 42 1 0 0 0 0
37 19 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
44 51 1 0 0 0 0
45 52 1 0 0 0 0
46 39 1 0 0 0 0
53 54 1 0 0 0 0
54 55 2 0 0 0 0
54 56 1 0 0 0 0
56 57 2 0 0 0 0
57 58 1 0 0 0 0
58 59 2 0 0 0 0
59 60 1 0 0 0 0
60 61 2 0 0 0 0
61 62 1 0 0 0 0
62 63 2 0 0 0 0
63 58 1 0 0 0 0
61 64 1 0 0 0 0
53 43 1 0 0 0 0
S SKP 5
ID FL5FAAGS0130
FORMULA C42H46O22
EXACTMASS 902.248073156
AVERAGEMASS 902.80144
SMILES C(O3)(c(c7)ccc(c7)O)=C(OC(C(OC(C(O)5)OC(C(OC(=O)C=Cc(c6)ccc(O)c6)C5O)CO)4)OC(C(O)C4O)C)C(c(c23)c(O)cc(c2)OC(O1)C(O)C(O)C(C1CO)O)=O
M END