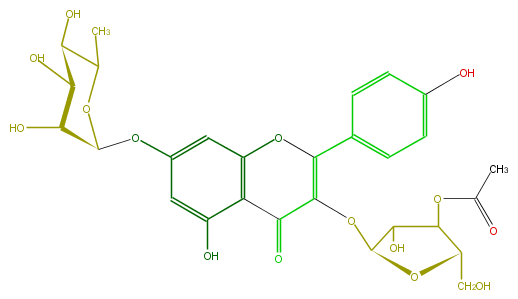
Mol:FL5FAAGS0064
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.7129 0.5383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7129 0.5383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7129 -0.2866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7129 -0.2866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9984 -0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9984 -0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2840 -0.2866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2840 -0.2866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2840 0.5383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2840 0.5383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9984 0.9508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9984 0.9508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4305 -0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4305 -0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1450 -0.2866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1450 -0.2866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1450 0.5383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1450 0.5383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4305 0.9508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4305 0.9508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4305 -1.4761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4305 -1.4761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9002 0.9653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9002 0.9653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6284 0.5448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6284 0.5448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3566 0.9653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3566 0.9653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3566 1.8060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3566 1.8060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6284 2.2265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6284 2.2265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9002 1.8060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9002 1.8060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9984 -1.4095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9984 -1.4095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1360 2.2561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1360 2.2561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4254 0.9427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4254 0.9427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8852 -0.7073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8852 -0.7073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9227 1.1339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9227 1.1339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1807 0.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1807 0.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4161 1.5293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4161 1.5293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1807 2.3582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1807 2.3582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9227 2.7867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9227 2.7867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6873 1.9628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6873 1.9628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7496 3.4325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7496 3.4325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2472 3.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2472 3.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4666 2.5485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4666 2.5485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7295 1.1461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7295 1.1461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6110 -0.8388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6110 -0.8388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7296 -0.8388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7296 -0.8388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2794 -1.3127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2794 -1.3127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1596 -1.8320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1596 -1.8320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0849 -1.3579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0849 -1.3579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7296 -1.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7296 -1.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6177 -0.2742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6177 -0.2742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3602 -0.2742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3602 -0.2742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7295 -0.9137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7295 -0.9137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7110 0.3334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7110 0.3334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0849 -2.0076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0849 -2.0076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6719 -3.4325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6719 -3.4325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 23 20 1 0 0 0 0 | + | 23 20 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 32 1 0 0 0 0 | + | 36 32 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 34 21 1 0 0 0 0 | + | 34 21 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 47 0.0000 0.6497 | + | M SBV 1 47 0.0000 0.6497 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGS0064 | + | ID FL5FAAGS0064 |
| − | FORMULA C28H30O15 | + | FORMULA C28H30O15 |
| − | EXACTMASS 606.15847029 | + | EXACTMASS 606.15847029 |
| − | AVERAGEMASS 606.5288 | + | AVERAGEMASS 606.5288 |
| − | SMILES OC(C1Oc(c2)cc(O)c(C(=O)3)c2OC(c(c5)ccc(c5)O)=C3OC(O4)C(C(OC(C)=O)C4CO)O)C(C(C(O1)C)O)O | + | SMILES OC(C1Oc(c2)cc(O)c(C(=O)3)c2OC(c(c5)ccc(c5)O)=C3OC(O4)C(C(OC(C)=O)C4CO)O)C(C(C(O1)C)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.7129 0.5383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7129 -0.2866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9984 -0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2840 -0.2866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2840 0.5383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9984 0.9508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4305 -0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1450 -0.2866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1450 0.5383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4305 0.9508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4305 -1.4761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9002 0.9653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6284 0.5448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3566 0.9653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3566 1.8060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6284 2.2265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9002 1.8060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9984 -1.4095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1360 2.2561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4254 0.9427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8852 -0.7073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9227 1.1339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1807 0.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4161 1.5293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1807 2.3582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9227 2.7867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6873 1.9628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7496 3.4325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2472 3.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4666 2.5485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7295 1.1461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6110 -0.8388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7296 -0.8388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2794 -1.3127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1596 -1.8320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0849 -1.3579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7296 -1.2436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6177 -0.2742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3602 -0.2742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7295 -0.9137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7110 0.3334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0849 -2.0076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6719 -3.4325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
27 30 1 0 0 0 0
22 31 1 0 0 0 0
23 20 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 32 1 0 0 0 0
33 37 1 0 0 0 0
32 38 1 0 0 0 0
34 21 1 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
39 41 1 0 0 0 0
42 43 1 0 0 0 0
36 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 CH2OH
M SBV 1 47 0.0000 0.6497
S SKP 5
ID FL5FAAGS0064
FORMULA C28H30O15
EXACTMASS 606.15847029
AVERAGEMASS 606.5288
SMILES OC(C1Oc(c2)cc(O)c(C(=O)3)c2OC(c(c5)ccc(c5)O)=C3OC(O4)C(C(OC(C)=O)C4CO)O)C(C(C(O1)C)O)O
M END