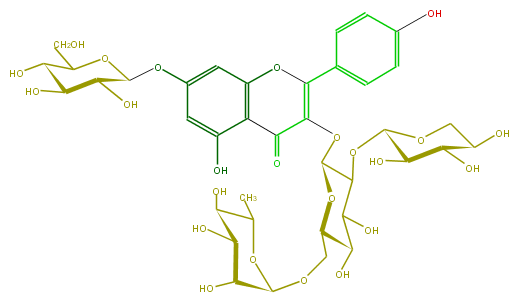
Mol:FL5FAAGL0056
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 62 68 0 0 0 0 0 0 0 0999 V2000 | + | 62 68 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4746 1.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4746 1.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4746 0.8150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4746 0.8150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7601 0.4025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7601 0.4025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0456 0.8150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0456 0.8150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0456 1.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0456 1.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7601 2.0524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7601 2.0524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6688 0.4025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6688 0.4025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3833 0.8150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3833 0.8150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3833 1.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3833 1.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6688 2.0524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6688 2.0524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6688 -0.2408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6688 -0.2408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0975 2.0523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0975 2.0523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8257 1.6318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8257 1.6318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5539 2.0523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5539 2.0523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5539 2.8931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5539 2.8931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8257 3.3135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8257 3.3135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0975 2.8931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0975 2.8931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1888 2.0523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1888 2.0523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1179 0.3688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1179 0.3688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7601 -0.4223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7601 -0.4223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3680 3.3632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3680 3.3632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4943 -0.6870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4943 -0.6870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7458 -0.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7458 -0.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9833 -1.0859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9833 -1.0859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7458 -1.9218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7458 -1.9218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4943 -2.3541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4943 -2.3541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2569 -1.5230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2569 -1.5230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5102 0.0084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5102 0.0084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8496 -1.8652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8496 -1.8652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1667 -2.9215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1667 -2.9215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8530 -2.5219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8530 -2.5219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2452 0.5317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2452 0.5317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8134 -0.2183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8134 -0.2183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6315 0.0998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6315 0.0998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4210 0.1084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4210 0.1084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8473 0.6822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8473 0.6822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1315 0.3044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1315 0.3044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1572 -0.2023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1572 -0.2023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2887 -0.3598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2887 -0.3598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0090 0.4639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0090 0.4639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9288 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9288 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4082 1.4380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4082 1.4380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6583 1.7297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6583 1.7297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8768 1.7211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8768 1.7211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4605 2.2633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4605 2.2633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2264 1.9883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2264 1.9883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5145 1.9146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5145 1.9146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1252 1.4704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1252 1.4704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9385 1.1720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9385 1.1720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3242 -3.1221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3242 -3.1221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5758 -3.3632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5758 -3.3632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0966 -2.5642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0966 -2.5642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1107 -1.6273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1107 -1.6273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7894 -1.3861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7894 -1.3861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3102 -2.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3102 -2.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7913 -0.9116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7913 -0.9116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1681 -1.0665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1681 -1.0665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1015 -1.8154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1015 -1.8154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9339 -3.2494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9339 -3.2494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3209 -3.0622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3209 -3.0622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7559 2.6049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7559 2.6049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.0090 2.8862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.0090 2.8862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 15 1 0 0 0 0 | + | 21 15 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 18 1 0 0 0 0 | + | 44 18 1 0 0 0 0 |
| − | 51 50 1 1 0 0 0 | + | 51 50 1 1 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 1 0 0 0 | + | 54 55 1 1 0 0 0 |
| − | 55 50 1 1 0 0 0 | + | 55 50 1 1 0 0 0 |
| − | 54 56 1 0 0 0 0 | + | 54 56 1 0 0 0 0 |
| − | 53 57 1 0 0 0 0 | + | 53 57 1 0 0 0 0 |
| − | 55 58 1 0 0 0 0 | + | 55 58 1 0 0 0 0 |
| − | 50 59 1 0 0 0 0 | + | 50 59 1 0 0 0 0 |
| − | 51 60 1 0 0 0 0 | + | 51 60 1 0 0 0 0 |
| − | 60 31 1 0 0 0 0 | + | 60 31 1 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 46 61 1 0 0 0 0 | + | 46 61 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 61 62 | + | M SAL 1 2 61 62 |
| − | M SBL 1 1 68 | + | M SBL 1 1 68 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 68 0.5294 -0.6166 | + | M SBV 1 68 0.5294 -0.6166 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGL0056 | + | ID FL5FAAGL0056 |
| − | FORMULA C38H48O24 | + | FORMULA C38H48O24 |
| − | EXACTMASS 888.253552464 | + | EXACTMASS 888.253552464 |
| − | AVERAGEMASS 888.77332 | + | AVERAGEMASS 888.77332 |
| − | SMILES c(O6)(c1C(=O)C(=C6c(c7)ccc(O)c7)OC(O3)C(OC(C(O)5)OCC(C5O)O)C(C(C3COC(C4O)OC(C)C(O)C4O)O)O)cc(OC(O2)C(O)C(O)C(O)C(CO)2)cc(O)1 | + | SMILES c(O6)(c1C(=O)C(=C6c(c7)ccc(O)c7)OC(O3)C(OC(C(O)5)OCC(C5O)O)C(C(C3COC(C4O)OC(C)C(O)C4O)O)O)cc(OC(O2)C(O)C(O)C(O)C(CO)2)cc(O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
62 68 0 0 0 0 0 0 0 0999 V2000
-1.4746 1.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4746 0.8150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7601 0.4025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0456 0.8150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0456 1.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7601 2.0524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6688 0.4025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3833 0.8150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3833 1.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6688 2.0524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6688 -0.2408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0975 2.0523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8257 1.6318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5539 2.0523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5539 2.8931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8257 3.3135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0975 2.8931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1888 2.0523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1179 0.3688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7601 -0.4223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3680 3.3632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4943 -0.6870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7458 -0.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9833 -1.0859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7458 -1.9218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4943 -2.3541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2569 -1.5230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5102 0.0084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8496 -1.8652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1667 -2.9215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8530 -2.5219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2452 0.5317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8134 -0.2183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6315 0.0998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4210 0.1084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8473 0.6822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1315 0.3044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1572 -0.2023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2887 -0.3598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.0090 0.4639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9288 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4082 1.4380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6583 1.7297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8768 1.7211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4605 2.2633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2264 1.9883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5145 1.9146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1252 1.4704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9385 1.1720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3242 -3.1221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5758 -3.3632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0966 -2.5642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1107 -1.6273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7894 -1.3861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3102 -2.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7913 -0.9116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1681 -1.0665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1015 -1.8154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9339 -3.2494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3209 -3.0622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7559 2.6049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.0090 2.8862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 15 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
25 31 1 0 0 0 0
23 19 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
33 38 1 0 0 0 0
34 39 1 0 0 0 0
28 32 1 0 0 0 0
35 40 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 18 1 0 0 0 0
51 50 1 1 0 0 0
51 52 1 0 0 0 0
52 53 1 0 0 0 0
53 54 1 0 0 0 0
54 55 1 1 0 0 0
55 50 1 1 0 0 0
54 56 1 0 0 0 0
53 57 1 0 0 0 0
55 58 1 0 0 0 0
50 59 1 0 0 0 0
51 60 1 0 0 0 0
60 31 1 0 0 0 0
61 62 1 0 0 0 0
46 61 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 61 62
M SBL 1 1 68
M SMT 1 ^ CH2OH
M SBV 1 68 0.5294 -0.6166
S SKP 5
ID FL5FAAGL0056
FORMULA C38H48O24
EXACTMASS 888.253552464
AVERAGEMASS 888.77332
SMILES c(O6)(c1C(=O)C(=C6c(c7)ccc(O)c7)OC(O3)C(OC(C(O)5)OCC(C5O)O)C(C(C3COC(C4O)OC(C)C(O)C4O)O)O)cc(OC(O2)C(O)C(O)C(O)C(CO)2)cc(O)1
M END