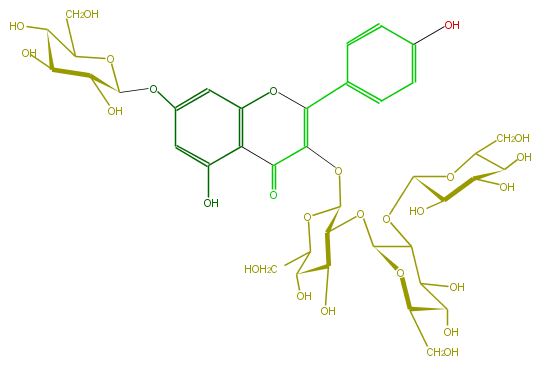
Mol:FL5FAAGL0054
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 65 71 0 0 0 0 0 0 0 0999 V2000 | + | 65 71 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0858 1.1129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0858 1.1129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0858 0.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0858 0.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3713 -0.1246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3713 -0.1246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6568 0.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6568 0.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6568 1.1129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6568 1.1129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3713 1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3713 1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0576 -0.1246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0576 -0.1246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7721 0.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7721 0.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7721 1.1129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7721 1.1129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0576 1.5254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0576 1.5254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0576 -0.7677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0576 -0.7677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6715 1.6842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6715 1.6842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3997 1.2638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3997 1.2638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1279 1.6842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1279 1.6842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1279 2.5250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1279 2.5250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3997 2.9453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3997 2.9453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6715 2.5250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6715 2.5250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3713 -0.9492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3713 -0.9492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9072 2.9748 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9072 2.9748 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4953 -0.2380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4953 -0.2380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5708 1.5600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5708 1.5600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9133 2.8603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9133 2.8603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8018 1.9713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8018 1.9713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9756 1.8442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9756 1.8442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3205 1.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3205 1.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5205 2.2216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5205 2.2216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2905 2.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2905 2.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4846 2.9427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4846 2.9427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3657 2.3560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3657 2.3560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4783 1.0856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4783 1.0856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5535 -2.5199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5535 -2.5199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2670 -2.3359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2670 -2.3359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2330 -1.6069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2330 -1.6069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5222 -1.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5222 -1.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8066 -1.2540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8066 -1.2540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8757 -2.0137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8757 -2.0137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6594 -2.9785 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6594 -2.9785 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1873 -3.3070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1873 -3.3070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9832 -1.1888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9832 -1.1888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0785 -1.9102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0785 -1.9102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2580 -1.9102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2580 -1.9102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8478 -2.4806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8478 -2.4806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0494 -3.2807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0494 -3.2807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8698 -3.2807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8698 -3.2807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2802 -2.7103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2802 -2.7103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5428 -1.2919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5428 -1.2919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0146 -2.7877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0146 -2.7877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8753 -3.7624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8753 -3.7624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1226 -0.3705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1226 -0.3705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7972 -0.8947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7972 -0.8947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4400 -0.4234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4400 -0.4234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1303 -0.2304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1303 -0.2304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4923 0.1380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4923 0.1380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9534 -0.3615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9534 -0.3615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3014 -1.1228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3014 -1.1228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1145 -0.5850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1145 -0.5850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4846 0.0602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4846 0.0602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0380 0.4815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0380 0.4815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2943 1.5284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2943 1.5284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5226 3.2050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5226 3.2050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1602 4.2007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1602 4.2007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4897 -4.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4897 -4.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7406 -3.4516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7406 -3.4516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2009 -2.3905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2009 -2.3905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9821 -2.8902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9821 -2.8902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 21 25 1 0 0 0 0 | + | 21 25 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 20 1 0 0 0 0 | + | 34 20 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 40 1 0 0 0 0 | + | 45 40 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 44 48 1 0 0 0 0 | + | 44 48 1 0 0 0 0 |
| − | 41 39 1 0 0 0 0 | + | 41 39 1 0 0 0 0 |
| − | 49 50 1 1 0 0 0 | + | 49 50 1 1 0 0 0 |
| − | 50 51 1 1 0 0 0 | + | 50 51 1 1 0 0 0 |
| − | 52 51 1 1 0 0 0 | + | 52 51 1 1 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 49 1 0 0 0 0 | + | 54 49 1 0 0 0 0 |
| − | 50 55 1 0 0 0 0 | + | 50 55 1 0 0 0 0 |
| − | 51 56 1 0 0 0 0 | + | 51 56 1 0 0 0 0 |
| − | 46 49 1 0 0 0 0 | + | 46 49 1 0 0 0 0 |
| − | 52 57 1 0 0 0 0 | + | 52 57 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 53 58 1 0 0 0 0 | + | 53 58 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 27 60 1 0 0 0 0 | + | 27 60 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 43 62 1 0 0 0 0 | + | 43 62 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 36 64 1 0 0 0 0 | + | 36 64 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 58 59 | + | M SAL 1 2 58 59 |
| − | M SBL 1 1 65 | + | M SBL 1 1 65 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 65 -0.5457 -0.3435 | + | M SBV 1 65 -0.5457 -0.3435 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 60 61 | + | M SAL 2 2 60 61 |
| − | M SBL 2 1 67 | + | M SBL 2 1 67 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 67 0.2321 -0.9542 | + | M SBV 2 67 0.2321 -0.9542 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 62 63 | + | M SAL 3 2 62 63 |
| − | M SBL 3 1 69 | + | M SBL 3 1 69 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SBV 3 69 -0.4403 0.9199 | + | M SBV 3 69 -0.4403 0.9199 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 64 65 | + | M SAL 4 2 64 65 |
| − | M SBL 4 1 71 | + | M SBL 4 1 71 |
| − | M SMT 4 ^ CH2OH | + | M SMT 4 ^ CH2OH |
| − | M SBV 4 71 0.6748 0.3769 | + | M SBV 4 71 0.6748 0.3769 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGL0054 | + | ID FL5FAAGL0054 |
| − | FORMULA C39H50O26 | + | FORMULA C39H50O26 |
| − | EXACTMASS 934.2590317720001 | + | EXACTMASS 934.2590317720001 |
| − | AVERAGEMASS 934.7987 | + | AVERAGEMASS 934.7987 |
| − | SMILES O(C(O7)C(C(C(C7CO)O)O)O)C(C1O)C(OC(C2OC(C(=O)3)=C(c(c6)ccc(c6)O)Oc(c4)c3c(O)cc(OC(O5)C(O)C(C(O)C(CO)5)O)4)C(C(O)C(O2)CO)O)OC(CO)C1O | + | SMILES O(C(O7)C(C(C(C7CO)O)O)O)C(C1O)C(OC(C2OC(C(=O)3)=C(c(c6)ccc(c6)O)Oc(c4)c3c(O)cc(OC(O5)C(O)C(C(O)C(CO)5)O)4)C(C(O)C(O2)CO)O)OC(CO)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
65 71 0 0 0 0 0 0 0 0999 V2000
-2.0858 1.1129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0858 0.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3713 -0.1246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6568 0.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6568 1.1129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3713 1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0576 -0.1246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7721 0.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7721 1.1129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0576 1.5254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0576 -0.7677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6715 1.6842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3997 1.2638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1279 1.6842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1279 2.5250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3997 2.9453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6715 2.5250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3713 -0.9492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9072 2.9748 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4953 -0.2380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5708 1.5600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9133 2.8603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8018 1.9713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9756 1.8442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3205 1.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5205 2.2216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2905 2.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4846 2.9427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3657 2.3560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4783 1.0856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5535 -2.5199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2670 -2.3359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2330 -1.6069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5222 -1.0263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8066 -1.2540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8757 -2.0137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6594 -2.9785 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1873 -3.3070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9832 -1.1888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0785 -1.9102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2580 -1.9102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8478 -2.4806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0494 -3.2807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8698 -3.2807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2802 -2.7103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5428 -1.2919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0146 -2.7877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8753 -3.7624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1226 -0.3705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7972 -0.8947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4400 -0.4234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1303 -0.2304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4923 0.1380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9534 -0.3615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3014 -1.1228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1145 -0.5850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4846 0.0602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0380 0.4815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2943 1.5284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5226 3.2050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1602 4.2007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4897 -4.2007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7406 -3.4516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2009 -2.3905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9821 -2.8902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
1 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
21 25 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 20 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 40 1 0 0 0 0
40 46 1 0 0 0 0
45 47 1 0 0 0 0
44 48 1 0 0 0 0
41 39 1 0 0 0 0
49 50 1 1 0 0 0
50 51 1 1 0 0 0
52 51 1 1 0 0 0
52 53 1 0 0 0 0
53 54 1 0 0 0 0
54 49 1 0 0 0 0
50 55 1 0 0 0 0
51 56 1 0 0 0 0
46 49 1 0 0 0 0
52 57 1 0 0 0 0
58 59 1 0 0 0 0
53 58 1 0 0 0 0
60 61 1 0 0 0 0
27 60 1 0 0 0 0
62 63 1 0 0 0 0
43 62 1 0 0 0 0
64 65 1 0 0 0 0
36 64 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 58 59
M SBL 1 1 65
M SMT 1 CH2OH
M SBV 1 65 -0.5457 -0.3435
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 60 61
M SBL 2 1 67
M SMT 2 ^ CH2OH
M SBV 2 67 0.2321 -0.9542
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 62 63
M SBL 3 1 69
M SMT 3 CH2OH
M SBV 3 69 -0.4403 0.9199
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 64 65
M SBL 4 1 71
M SMT 4 ^ CH2OH
M SBV 4 71 0.6748 0.3769
S SKP 5
ID FL5FAAGL0054
FORMULA C39H50O26
EXACTMASS 934.2590317720001
AVERAGEMASS 934.7987
SMILES O(C(O7)C(C(C(C7CO)O)O)O)C(C1O)C(OC(C2OC(C(=O)3)=C(c(c6)ccc(c6)O)Oc(c4)c3c(O)cc(OC(O5)C(O)C(C(O)C(CO)5)O)4)C(C(O)C(O2)CO)O)OC(CO)C1O
M END