Mol:FL5FAAGI0014
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 69 75 0 0 0 0 0 0 0 0999 V2000 | + | 69 75 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8710 1.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8710 1.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8710 0.2519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8710 0.2519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1527 -0.1628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1527 -0.1628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4344 0.2519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4344 0.2519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4344 1.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4344 1.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1527 1.4961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1527 1.4961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2839 -0.1628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2839 -0.1628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0022 0.2519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0022 0.2519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0022 1.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0022 1.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2839 1.4961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2839 1.4961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2858 -0.9826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2858 -0.9826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7203 1.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7203 1.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4524 1.0733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4524 1.0733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1846 1.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1846 1.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1846 2.3413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1846 2.3413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4524 2.7640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4524 2.7640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7203 2.3413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7203 2.3413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5891 1.4959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5891 1.4959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9164 2.7639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9164 2.7639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8632 -0.2310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8632 -0.2310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1527 -0.9919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1527 -0.9919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1527 2.3252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1527 2.3252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8708 2.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8708 2.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8708 3.5690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8708 3.5690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1527 3.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1527 3.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5888 3.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5888 3.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0853 1.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0853 1.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6060 0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6060 0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9158 1.0328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9158 1.0328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2499 1.0401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2499 1.0401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7339 1.5240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7339 1.5240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3377 1.2054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3377 1.2054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7233 1.1737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7233 1.1737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3047 0.8481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3047 0.8481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2716 0.5120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2716 0.5120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8126 -1.8369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8126 -1.8369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2175 -2.3362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2175 -2.3362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9925 -2.3911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9925 -2.3911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6255 -2.8487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6255 -2.8487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2207 -2.3494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2207 -2.3494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4458 -2.2945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4458 -2.2945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2262 -1.7648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2262 -1.7648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0395 -1.9696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0395 -1.9696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7233 -2.6092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7233 -2.6092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2682 -1.3674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2682 -1.3674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2682 -2.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2682 -2.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7276 -1.5534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7276 -1.5534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3719 -1.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3719 -1.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3721 -0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3721 -0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9125 -1.2051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9125 -1.2051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6899 -2.2726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6899 -2.2726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7626 -2.0977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7626 -2.0977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8525 -1.7464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8525 -1.7464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7589 -1.9955 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7589 -1.9955 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8309 -0.5598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8309 -0.5598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5453 -0.1472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5453 -0.1472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3186 -0.9404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3186 -0.9404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5453 -1.7384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5453 -1.7384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8309 -2.1509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8309 -2.1509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0575 -1.3577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0575 -1.3577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9110 0.0193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9110 0.0193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5500 -1.8079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5500 -1.8079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0133 -2.8749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0133 -2.8749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8386 1.7600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8386 1.7600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2030 2.7609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2030 2.7609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3703 -2.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3703 -2.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0059 -3.9835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0059 -3.9835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5942 -2.4319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5942 -2.4319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1184 -3.6120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1184 -3.6120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 6 22 1 0 0 0 0 | + | 6 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 27 1 0 0 0 0 | + | 32 27 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 18 1 0 0 0 0 | + | 30 18 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 1 0 0 0 | + | 37 38 1 1 0 0 0 |
| − | 38 39 1 1 0 0 0 | + | 38 39 1 1 0 0 0 |
| − | 40 39 1 1 0 0 0 | + | 40 39 1 1 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 40 44 1 0 0 0 0 | + | 40 44 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 47 48 1 1 0 0 0 | + | 47 48 1 1 0 0 0 |
| − | 49 48 1 1 0 0 0 | + | 49 48 1 1 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 47 51 1 0 0 0 0 | + | 47 51 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 48 54 1 0 0 0 0 | + | 48 54 1 0 0 0 0 |
| − | 49 20 1 0 0 0 0 | + | 49 20 1 0 0 0 0 |
| − | 37 54 1 0 0 0 0 | + | 37 54 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 1 1 0 0 0 | + | 56 57 1 1 0 0 0 |
| − | 57 58 1 1 0 0 0 | + | 57 58 1 1 0 0 0 |
| − | 59 58 1 1 0 0 0 | + | 59 58 1 1 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 55 1 0 0 0 0 | + | 60 55 1 0 0 0 0 |
| − | 55 61 1 0 0 0 0 | + | 55 61 1 0 0 0 0 |
| − | 60 62 1 0 0 0 0 | + | 60 62 1 0 0 0 0 |
| − | 59 63 1 0 0 0 0 | + | 59 63 1 0 0 0 0 |
| − | 56 35 1 0 0 0 0 | + | 56 35 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 32 64 1 0 0 0 0 | + | 32 64 1 0 0 0 0 |
| − | 66 67 1 0 0 0 0 | + | 66 67 1 0 0 0 0 |
| − | 39 66 1 0 0 0 0 | + | 39 66 1 0 0 0 0 |
| − | 68 69 1 0 0 0 0 | + | 68 69 1 0 0 0 0 |
| − | 58 68 1 0 0 0 0 | + | 58 68 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 64 65 | + | M SAL 1 2 64 65 |
| − | M SBL 1 1 71 | + | M SBL 1 1 71 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 71 0.5009 -0.5546 | + | M SBV 1 71 0.5009 -0.5546 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 66 67 | + | M SAL 2 2 66 67 |
| − | M SBL 2 1 73 | + | M SBL 2 1 73 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 73 -0.7448 0.1336 | + | M SBV 2 73 -0.7448 0.1336 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 68 69 | + | M SAL 3 2 68 69 |
| − | M SBL 3 1 75 | + | M SBL 3 1 75 |
| − | M SMT 3 ^ CH2OH | + | M SMT 3 ^ CH2OH |
| − | M SBV 3 75 0.0489 0.6935 | + | M SBV 3 75 0.0489 0.6935 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGI0014 | + | ID FL5FAAGI0014 |
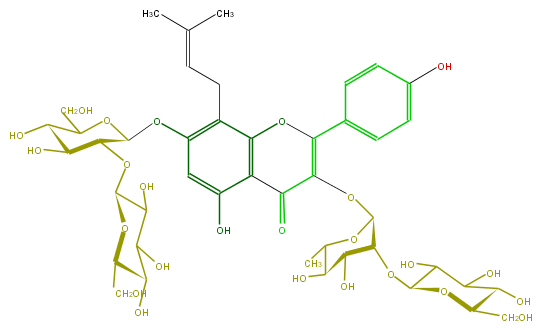
| − | FORMULA C44H58O25 | + | FORMULA C44H58O25 |
| − | EXACTMASS 986.326717406 | + | EXACTMASS 986.326717406 |
| − | AVERAGEMASS 986.91632 | + | AVERAGEMASS 986.91632 |
| − | SMILES Oc(c5)c(c3c(c5OC(O6)C(OC(O7)C(C(O)C(C7CO)O)O)C(O)C(C(CO)6)O)CC=C(C)C)C(=O)C(=C(c(c4)ccc(c4)O)O3)OC(O2)C(C(O)C(C(C)2)O)OC(C(O)1)OC(CO)C(O)C1O | + | SMILES Oc(c5)c(c3c(c5OC(O6)C(OC(O7)C(C(O)C(C7CO)O)O)C(O)C(C(CO)6)O)CC=C(C)C)C(=O)C(=C(c(c4)ccc(c4)O)O3)OC(O2)C(C(O)C(C(C)2)O)OC(C(O)1)OC(CO)C(O)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
69 75 0 0 0 0 0 0 0 0999 V2000
-1.8710 1.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8710 0.2519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1527 -0.1628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4344 0.2519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4344 1.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1527 1.4961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2839 -0.1628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0022 0.2519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0022 1.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2839 1.4961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2858 -0.9826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7203 1.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4524 1.0733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1846 1.4959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1846 2.3413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4524 2.7640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7203 2.3413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5891 1.4959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9164 2.7639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8632 -0.2310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1527 -0.9919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1527 2.3252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8708 2.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8708 3.5690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1527 3.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5888 3.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0853 1.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6060 0.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9158 1.0328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2499 1.0401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7339 1.5240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3377 1.2054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7233 1.1737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3047 0.8481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2716 0.5120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8126 -1.8369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2175 -2.3362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9925 -2.3911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6255 -2.8487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2207 -2.3494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4458 -2.2945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2262 -1.7648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0395 -1.9696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7233 -2.6092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2682 -1.3674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2682 -2.0283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7276 -1.5534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3719 -1.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3721 -0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9125 -1.2051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6899 -2.2726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7626 -2.0977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8525 -1.7464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7589 -1.9955 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8309 -0.5598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5453 -0.1472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3186 -0.9404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5453 -1.7384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8309 -2.1509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0575 -1.3577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9110 0.0193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5500 -1.8079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0133 -2.8749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8386 1.7600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2030 2.7609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3703 -2.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0059 -3.9835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5942 -2.4319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1184 -3.6120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
3 21 1 0 0 0 0
6 22 1 0 0 0 0
22 23 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
24 26 1 0 0 0 0
27 28 1 1 0 0 0
28 29 1 1 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 27 1 0 0 0 0
27 33 1 0 0 0 0
28 34 1 0 0 0 0
29 35 1 0 0 0 0
30 18 1 0 0 0 0
36 37 1 0 0 0 0
37 38 1 1 0 0 0
38 39 1 1 0 0 0
40 39 1 1 0 0 0
40 41 1 0 0 0 0
41 36 1 0 0 0 0
36 42 1 0 0 0 0
41 43 1 0 0 0 0
40 44 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 1 0 0 0
47 48 1 1 0 0 0
49 48 1 1 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
47 51 1 0 0 0 0
46 52 1 0 0 0 0
45 53 1 0 0 0 0
48 54 1 0 0 0 0
49 20 1 0 0 0 0
37 54 1 0 0 0 0
55 56 1 0 0 0 0
56 57 1 1 0 0 0
57 58 1 1 0 0 0
59 58 1 1 0 0 0
59 60 1 0 0 0 0
60 55 1 0 0 0 0
55 61 1 0 0 0 0
60 62 1 0 0 0 0
59 63 1 0 0 0 0
56 35 1 0 0 0 0
64 65 1 0 0 0 0
32 64 1 0 0 0 0
66 67 1 0 0 0 0
39 66 1 0 0 0 0
68 69 1 0 0 0 0
58 68 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 64 65
M SBL 1 1 71
M SMT 1 ^ CH2OH
M SBV 1 71 0.5009 -0.5546
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 66 67
M SBL 2 1 73
M SMT 2 CH2OH
M SBV 2 73 -0.7448 0.1336
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 68 69
M SBL 3 1 75
M SMT 3 ^ CH2OH
M SBV 3 75 0.0489 0.6935
S SKP 5
ID FL5FAAGI0014
FORMULA C44H58O25
EXACTMASS 986.326717406
AVERAGEMASS 986.91632
SMILES Oc(c5)c(c3c(c5OC(O6)C(OC(O7)C(C(O)C(C7CO)O)O)C(O)C(C(CO)6)O)CC=C(C)C)C(=O)C(=C(c(c4)ccc(c4)O)O3)OC(O2)C(C(O)C(C(C)2)O)OC(C(O)1)OC(CO)C(O)C1O
M END