Mol:FL4DACGS0010
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.1116 0.3486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1116 0.3486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5908 0.0479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5908 0.0479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0699 0.3486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0699 0.3486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0699 0.9501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0699 0.9501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5908 1.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5908 1.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1116 0.9501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1116 0.9501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5490 0.0479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5490 0.0479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0281 0.3486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0281 0.3486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0281 0.9501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0281 0.9501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5490 1.2508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5490 1.2508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5490 -0.4760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5490 -0.4760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4484 1.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4484 1.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9197 0.9443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9197 0.9443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3910 1.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3910 1.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3910 1.8600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3910 1.8600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9197 2.1653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9197 2.1653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4484 1.8600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4484 1.8600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5623 1.2373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5623 1.2373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5599 -0.2281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5599 -0.2281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1377 2.1653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1377 2.1653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5908 -0.5530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5908 -0.5530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3351 0.1114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3351 0.1114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6360 -0.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6360 -0.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0575 -0.2443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0575 -0.2443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5245 -0.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5245 -0.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8254 0.1114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8254 0.1114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2469 -0.0538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2469 -0.0538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8938 -0.0383 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8938 -0.0383 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4850 0.3587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4850 0.3587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2594 -0.1392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2594 -0.1392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4822 -0.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4822 -0.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0371 -0.9293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0371 -0.9293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1121 -1.7563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1121 -1.7563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7964 -1.7563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7964 -1.7563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0907 -2.2661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0907 -2.2661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6795 -2.2661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6795 -2.2661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9738 -1.7563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9738 -1.7563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6795 -1.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6795 -1.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0907 -1.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0907 -1.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9531 -0.6749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9531 -0.6749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5623 -1.7563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5623 -1.7563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9737 -2.7758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9737 -2.7758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8346 -2.2370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8346 -2.2370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9197 2.7758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9197 2.7758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 6 0 0 0 | + | 9 12 1 6 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 8 19 1 1 0 0 0 | + | 8 19 1 1 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 2 21 1 0 0 0 0 | + | 2 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 43 2 0 0 0 0 | + | 33 43 2 0 0 0 0 |
| − | 16 44 1 0 0 0 0 | + | 16 44 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL4DACGS0010 | + | ID FL4DACGS0010 |
| − | KNApSAcK_ID C00008702 | + | KNApSAcK_ID C00008702 |
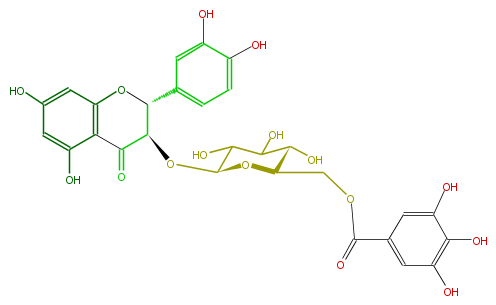
| − | NAME Taxillusin | + | NAME Taxillusin |
| − | CAS_RN 66656-93-7 | + | CAS_RN 66656-93-7 |
| − | FORMULA C28H26O16 | + | FORMULA C28H26O16 |
| − | EXACTMASS 618.122084784 | + | EXACTMASS 618.122084784 |
| − | AVERAGEMASS 618.49644 | + | AVERAGEMASS 618.49644 |
| − | SMILES c(c1O)cc(C(C(OC(C5O)OC(C(O)C5O)COC(=O)c(c4)cc(O)c(c(O)4)O)3)Oc(c2)c(C3=O)c(cc(O)2)O)cc1O | + | SMILES c(c1O)cc(C(C(OC(C5O)OC(C(O)C5O)COC(=O)c(c4)cc(O)c(c(O)4)O)3)Oc(c2)c(C3=O)c(cc(O)2)O)cc1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-4.1116 0.3486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5908 0.0479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0699 0.3486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0699 0.9501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5908 1.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1116 0.9501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5490 0.0479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0281 0.3486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0281 0.9501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5490 1.2508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5490 -0.4760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4484 1.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9197 0.9443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3910 1.2495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3910 1.8600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9197 2.1653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4484 1.8600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5623 1.2373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5599 -0.2281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1377 2.1653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5908 -0.5530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3351 0.1114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6360 -0.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0575 -0.2443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5245 -0.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8254 0.1114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2469 -0.0538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8938 -0.0383 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4850 0.3587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2594 -0.1392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4822 -0.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0371 -0.9293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1121 -1.7563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7964 -1.7563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0907 -2.2661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6795 -2.2661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9738 -1.7563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6795 -1.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0907 -1.2464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9531 -0.6749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5623 -1.7563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9737 -2.7758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8346 -2.2370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9197 2.7758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 6 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
6 18 1 0 0 0 0
8 19 1 1 0 0 0
15 20 1 0 0 0 0
2 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
25 31 1 0 0 0 0
31 32 1 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 34 1 0 0 0 0
38 40 1 0 0 0 0
37 41 1 0 0 0 0
36 42 1 0 0 0 0
32 33 1 0 0 0 0
33 43 2 0 0 0 0
16 44 1 0 0 0 0
23 19 1 0 0 0 0
S SKP 8
ID FL4DACGS0010
KNApSAcK_ID C00008702
NAME Taxillusin
CAS_RN 66656-93-7
FORMULA C28H26O16
EXACTMASS 618.122084784
AVERAGEMASS 618.49644
SMILES c(c1O)cc(C(C(OC(C5O)OC(C(O)C5O)COC(=O)c(c4)cc(O)c(c(O)4)O)3)Oc(c2)c(C3=O)c(cc(O)2)O)cc1O
M END