Mol:FL3FF8GS0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 37 40 0 0 0 0 0 0 0 0999 V2000 | + | 37 40 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0377 -1.7749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0377 -1.7749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0377 -2.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0377 -2.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3365 -3.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3365 -3.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6356 -2.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6356 -2.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6356 -1.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6356 -1.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3365 -1.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3365 -1.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0655 -3.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0655 -3.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7666 -2.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7666 -2.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7666 -1.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7666 -1.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0655 -1.4342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0655 -1.4342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0655 -3.7576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0655 -3.7576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4674 -1.4343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4674 -1.4343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1819 -1.8468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1819 -1.8468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8964 -1.4343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8964 -1.4343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8964 -0.6094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8964 -0.6094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1819 -0.1969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1819 -0.1969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4674 -0.6094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4674 -0.6094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3365 -3.7193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3365 -3.7193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1977 1.2331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1977 1.2331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9788 0.7822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9788 0.7822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7310 1.6494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7310 1.6494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9788 2.5219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9788 2.5219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1977 2.9730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1977 2.9730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4455 2.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4455 2.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4813 3.7576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4813 3.7576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9932 3.3382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9932 3.3382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3212 1.3086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3212 1.3086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7905 -0.1893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7905 -0.1893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1819 -2.6231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1819 -2.6231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3869 -2.1846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3869 -2.1846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6098 -1.4446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6098 -1.4446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3869 -0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3869 -0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3365 -0.6571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3365 -0.6571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7178 0.7687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7178 0.7687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2287 2.6241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2287 2.6241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2287 3.6499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2287 3.6499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1170 2.1112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1170 2.1112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 20 19 1 1 0 0 0 | + | 20 19 1 1 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 19 1 1 0 0 0 | + | 24 19 1 1 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 22 26 1 0 0 0 0 | + | 22 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 17 28 1 0 0 0 0 | + | 17 28 1 0 0 0 0 |
| − | 28 20 1 0 0 0 0 | + | 28 20 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 13 29 1 0 0 0 0 | + | 13 29 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 1 31 1 0 0 0 0 | + | 1 31 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 6 33 1 0 0 0 0 | + | 6 33 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 24 35 1 0 0 0 0 | + | 24 35 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 29 30 | + | M SAL 1 2 29 30 |
| − | M SBL 1 1 33 | + | M SBL 1 1 33 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 33 0.0000 0.7762 | + | M SBV 1 33 0.0000 0.7762 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 31 32 | + | M SAL 2 2 31 32 |
| − | M SBL 2 1 35 | + | M SBL 2 1 35 |
| − | M SMT 2 ^ OCH3 | + | M SMT 2 ^ OCH3 |
| − | M SBV 2 35 0.5721 -0.3303 | + | M SBV 2 35 0.5721 -0.3303 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 33 34 | + | M SAL 3 2 33 34 |
| − | M SBL 3 1 37 | + | M SBL 3 1 37 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 37 0.0000 -0.7771 | + | M SBV 3 37 0.0000 -0.7771 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 3 35 36 37 | + | M SAL 4 3 35 36 37 |
| − | M SBL 4 1 40 | + | M SBL 4 1 40 |
| − | M SMT 4 ^ COOH | + | M SMT 4 ^ COOH |
| − | M SBV 4 40 0.6742 -0.5184 | + | M SBV 4 40 0.6742 -0.5184 |
| − | S SKP 5 | + | S SKP 5 |
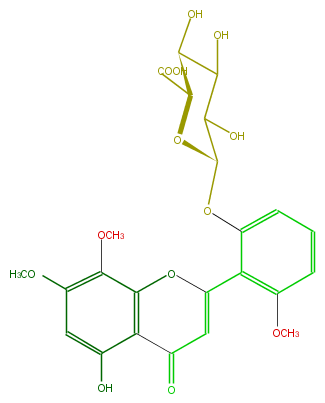
| − | ID FL3FF8GS0008 | + | ID FL3FF8GS0008 |
| − | FORMULA C24H24O13 | + | FORMULA C24H24O13 |
| − | EXACTMASS 520.121690854 | + | EXACTMASS 520.121690854 |
| − | AVERAGEMASS 520.43956 | + | AVERAGEMASS 520.43956 |
| − | SMILES C(=C2)(c(c(OC)3)c(OC(O4)C(O)C(O)C(C(C(O)=O)4)O)ccc3)Oc(c(C2=O)1)c(OC)c(cc1O)OC | + | SMILES C(=C2)(c(c(OC)3)c(OC(O4)C(O)C(O)C(C(C(O)=O)4)O)ccc3)Oc(c(C2=O)1)c(OC)c(cc1O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
37 40 0 0 0 0 0 0 0 0999 V2000
-2.0377 -1.7749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0377 -2.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3365 -3.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6356 -2.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6356 -1.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3365 -1.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0655 -3.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7666 -2.6485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7666 -1.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0655 -1.4342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0655 -3.7576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4674 -1.4343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1819 -1.8468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8964 -1.4343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8964 -0.6094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1819 -0.1969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4674 -0.6094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3365 -3.7193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1977 1.2331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9788 0.7822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7310 1.6494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9788 2.5219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1977 2.9730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4455 2.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4813 3.7576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9932 3.3382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3212 1.3086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7905 -0.1893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1819 -2.6231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3869 -2.1846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6098 -1.4446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3869 -0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3365 -0.6571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7178 0.7687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2287 2.6241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2287 3.6499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1170 2.1112 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
20 19 1 1 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 19 1 1 0 0 0
23 25 1 0 0 0 0
22 26 1 0 0 0 0
21 27 1 0 0 0 0
17 28 1 0 0 0 0
28 20 1 0 0 0 0
29 30 1 0 0 0 0
13 29 1 0 0 0 0
31 32 1 0 0 0 0
1 31 1 0 0 0 0
33 34 1 0 0 0 0
6 33 1 0 0 0 0
35 36 2 0 0 0 0
35 37 1 0 0 0 0
24 35 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 29 30
M SBL 1 1 33
M SMT 1 OCH3
M SBV 1 33 0.0000 0.7762
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 31 32
M SBL 2 1 35
M SMT 2 ^ OCH3
M SBV 2 35 0.5721 -0.3303
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 33 34
M SBL 3 1 37
M SMT 3 OCH3
M SBV 3 37 0.0000 -0.7771
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 3 35 36 37
M SBL 4 1 40
M SMT 4 ^ COOH
M SBV 4 40 0.6742 -0.5184
S SKP 5
ID FL3FF8GS0008
FORMULA C24H24O13
EXACTMASS 520.121690854
AVERAGEMASS 520.43956
SMILES C(=C2)(c(c(OC)3)c(OC(O4)C(O)C(O)C(C(C(O)=O)4)O)ccc3)Oc(c(C2=O)1)c(OC)c(cc1O)OC
M END