Mol:FL3FADGS0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.0502 -0.1504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0502 -0.1504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0502 -0.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0502 -0.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5013 -0.9316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5013 -0.9316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9523 -0.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9523 -0.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9523 -0.1504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9523 -0.1504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5013 0.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5013 0.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4034 -0.9316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4034 -0.9316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8545 -0.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8545 -0.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8545 -0.1504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8545 -0.1504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4034 0.1100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4034 0.1100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5751 -1.2995 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5751 -1.2995 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3054 0.1099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3054 0.1099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7651 -0.1555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7651 -0.1555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2248 0.1099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2248 0.1099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2248 0.6408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2248 0.6408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7651 0.9062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7651 0.9062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3054 0.6408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3054 0.6408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5013 -1.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5013 -1.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2248 0.6408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2248 0.6408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3999 0.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3999 0.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1012 -0.1815 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.1012 -0.1815 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.5856 -0.8621 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.5856 -0.8621 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.8431 -0.5734 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.8431 -0.5734 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.1266 -0.5656 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.1266 -0.5656 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.6473 -0.0449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6473 -0.0449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2968 -0.3877 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.2968 -0.3877 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.8150 -0.5936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8150 -0.5936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3894 -0.9012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3894 -0.9012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4177 -1.2876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4177 -1.2876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9949 1.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9949 1.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3833 2.3729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3833 2.3729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5009 0.5187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5009 0.5187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4556 0.8162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4556 0.8162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 16 30 1 0 0 0 0 | + | 16 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 32 33 | + | M SAL 2 2 32 33 |
| − | M SBL 2 1 35 | + | M SBL 2 1 35 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 35 -2.5009 0.5187 | + | M SVB 2 35 -2.5009 0.5187 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 30 31 | + | M SAL 1 2 30 31 |
| − | M SBL 1 1 33 | + | M SBL 1 1 33 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 33 2.9949 1.4514 | + | M SVB 1 33 2.9949 1.4514 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FADGS0004 | + | ID FL3FADGS0004 |
| − | KNApSAcK_ID C00004338 | + | KNApSAcK_ID C00004338 |
| − | NAME Luteolin 3'-methyl ether 7-glucoside | + | NAME Luteolin 3'-methyl ether 7-glucoside |
| − | CAS_RN 19993-32-9 | + | CAS_RN 19993-32-9 |
| − | FORMULA C22H22O11 | + | FORMULA C22H22O11 |
| − | EXACTMASS 462.116211546 | + | EXACTMASS 462.116211546 |
| − | AVERAGEMASS 462.40348000000006 | + | AVERAGEMASS 462.40348000000006 |
| − | SMILES [C@H]([C@@H]1O)([C@H](C(O[C@H]1Oc(c4)cc(c(c4O)2)OC(c(c3)cc(c(c3)O)OC)=CC2=O)CO)O)O | + | SMILES [C@H]([C@@H]1O)([C@H](C(O[C@H]1Oc(c4)cc(c(c4O)2)OC(c(c3)cc(c(c3)O)OC)=CC2=O)CO)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
0.0502 -0.1504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0502 -0.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5013 -0.9316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9523 -0.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9523 -0.1504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5013 0.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4034 -0.9316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8545 -0.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8545 -0.1504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4034 0.1100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5751 -1.2995 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3054 0.1099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7651 -0.1555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2248 0.1099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2248 0.6408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7651 0.9062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3054 0.6408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5013 -1.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2248 0.6408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3999 0.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1012 -0.1815 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.5856 -0.8621 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.8431 -0.5734 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.1266 -0.5656 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.6473 -0.0449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2968 -0.3877 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.8150 -0.5936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3894 -0.9012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4177 -1.2876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9949 1.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3833 2.3729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5009 0.5187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4556 0.8162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
1 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 20 1 0 0 0 0
16 30 1 0 0 0 0
30 31 1 0 0 0 0
26 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 32 33
M SBL 2 1 35
M SMT 2 CH2OH
M SVB 2 35 -2.5009 0.5187
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 30 31
M SBL 1 1 33
M SMT 1 OCH3
M SVB 1 33 2.9949 1.4514
S SKP 8
ID FL3FADGS0004
KNApSAcK_ID C00004338
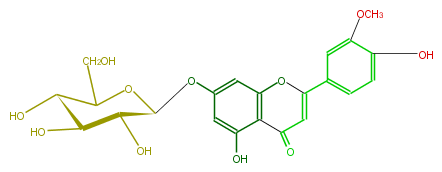
NAME Luteolin 3'-methyl ether 7-glucoside
CAS_RN 19993-32-9
FORMULA C22H22O11
EXACTMASS 462.116211546
AVERAGEMASS 462.40348000000006
SMILES [C@H]([C@@H]1O)([C@H](C(O[C@H]1Oc(c4)cc(c(c4O)2)OC(c(c3)cc(c(c3)O)OC)=CC2=O)CO)O)O
M END