Mol:FL3FACGS0068
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.3118 0.1327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3118 0.1327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3118 -0.7409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3118 -0.7409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6107 -1.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6107 -1.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9096 -0.7409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9096 -0.7409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9096 0.0685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9096 0.0685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6107 0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6107 0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2086 -1.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2086 -1.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5075 -0.7409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5075 -0.7409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5075 0.0685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5075 0.0685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2086 0.4733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2086 0.4733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2086 -1.9062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2086 -1.9062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8067 0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8067 0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0923 0.0606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0923 0.0606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6222 0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6222 0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6222 1.2982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6222 1.2982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0923 1.7107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0923 1.7107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8067 1.2982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8067 1.2982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6107 -1.9535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6107 -1.9535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0286 0.5466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0286 0.5466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3367 1.7107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3367 1.7107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5588 0.5244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5588 0.5244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1370 -0.2061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1370 -0.2061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9480 0.0257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9480 0.0257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7639 -0.2061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7639 -0.2061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1858 0.5244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1858 0.5244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3747 0.2927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3747 0.2927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8246 0.4405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8246 0.4405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9516 0.9026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9516 0.9026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8988 0.3573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8988 0.3573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4354 0.0157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4354 0.0157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9516 1.6245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9516 1.6245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3817 1.9535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3817 1.9535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4770 1.9278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4770 1.9278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5798 -0.0631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5798 -0.0631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0286 0.7143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0286 0.7143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0286 -0.8404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0286 -0.8404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 1 1 0 0 0 0 | + | 19 1 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 14 30 1 0 0 0 0 | + | 14 30 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 24 34 1 0 0 0 0 | + | 24 34 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 34 35 36 | + | M SAL 1 3 34 35 36 |
| − | M SBL 1 1 39 | + | M SBL 1 1 39 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SBV 1 39 -0.8159 -0.1430 | + | M SBV 1 39 -0.8159 -0.1430 |
| − | S SKP 5 | + | S SKP 5 |
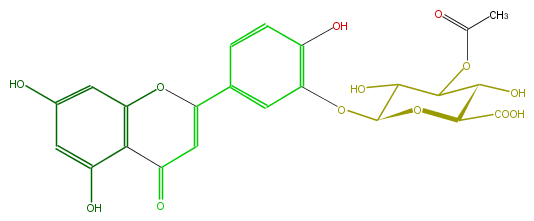
| − | ID FL3FACGS0068 | + | ID FL3FACGS0068 |
| − | FORMULA C23H20O13 | + | FORMULA C23H20O13 |
| − | EXACTMASS 504.090390726 | + | EXACTMASS 504.090390726 |
| − | AVERAGEMASS 504.3971 | + | AVERAGEMASS 504.3971 |
| − | SMILES C(O)(C(Oc(c4O)cc(cc4)C(O3)=CC(c(c32)c(O)cc(O)c2)=O)1)C(OC(C)=O)C(O)C(C(O)=O)O1 | + | SMILES C(O)(C(Oc(c4O)cc(cc4)C(O3)=CC(c(c32)c(O)cc(O)c2)=O)1)C(OC(C)=O)C(O)C(C(O)=O)O1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
-4.3118 0.1327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3118 -0.7409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6107 -1.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9096 -0.7409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9096 0.0685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6107 0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2086 -1.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5075 -0.7409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5075 0.0685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2086 0.4733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2086 -1.9062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8067 0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0923 0.0606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6222 0.4733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6222 1.2982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0923 1.7107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8067 1.2982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6107 -1.9535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0286 0.5466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3367 1.7107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5588 0.5244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1370 -0.2061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9480 0.0257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7639 -0.2061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1858 0.5244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3747 0.2927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8246 0.4405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9516 0.9026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8988 0.3573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4354 0.0157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9516 1.6245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3817 1.9535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4770 1.9278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5798 -0.0631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0286 0.7143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0286 -0.8404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 1 1 0 0 0 0
15 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
14 30 1 0 0 0 0
22 30 1 0 0 0 0
28 31 1 0 0 0 0
31 32 2 0 0 0 0
31 33 1 0 0 0 0
34 35 2 0 0 0 0
34 36 1 0 0 0 0
24 34 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 34 35 36
M SBL 1 1 39
M SMT 1 COOH
M SBV 1 39 -0.8159 -0.1430
S SKP 5
ID FL3FACGS0068
FORMULA C23H20O13
EXACTMASS 504.090390726
AVERAGEMASS 504.3971
SMILES C(O)(C(Oc(c4O)cc(cc4)C(O3)=CC(c(c32)c(O)cc(O)c2)=O)1)C(OC(C)=O)C(O)C(C(O)=O)O1
M END