Mol:FL3FACCS0045
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.7134 -1.0072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7134 -1.0072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7134 -1.8322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7134 -1.8322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4279 -2.2447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4279 -2.2447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1424 -1.8322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1424 -1.8322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1424 -1.0072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1424 -1.0072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4279 -0.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4279 -0.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8568 -2.2447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8568 -2.2447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5713 -1.8322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5713 -1.8322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5713 -1.0072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5713 -1.0072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8568 -0.5948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8568 -0.5948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8568 -2.8879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8568 -2.8879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0008 -0.5949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0008 -0.5949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6339 0.2969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6339 0.2969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4253 0.0088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4253 0.0088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0568 0.7661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0568 0.7661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1432 1.6086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1432 1.6086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3518 1.8967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3518 1.8967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7203 1.1393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7203 1.1393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5688 2.7066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5688 2.7066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9728 2.2444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9728 2.2444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0422 1.5387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0422 1.5387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7747 0.3425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7747 0.3425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6248 1.1190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6248 1.1190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4279 -3.0694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4279 -3.0694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1795 -0.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1795 -0.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9064 -0.9879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9064 -0.9879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6334 -0.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6334 -0.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6334 0.2711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6334 0.2711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9064 0.6908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9064 0.6908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1795 0.2711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1795 0.2711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3598 -0.9876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3598 -0.9876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3598 0.6906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3598 0.6906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4996 1.6132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4996 1.6132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4994 2.5154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4994 2.5154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2809 1.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2809 1.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0622 1.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0622 1.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8434 1.1623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8434 1.1623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5512 1.5710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5512 1.5710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2590 1.1623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2590 1.1623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2590 0.3451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2590 0.3451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5512 -0.0636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5512 -0.0636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8434 0.3451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8434 0.3451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9655 -0.0628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9655 -0.0628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3378 1.4586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3378 1.4586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5378 0.7122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5378 0.7122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9129 1.3877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9129 1.3877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5917 1.7661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5917 1.7661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3917 2.5125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3917 2.5125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0164 1.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0164 1.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9000 1.0208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9000 1.0208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0164 2.5137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0164 2.5137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0450 2.6876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0450 2.6876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7177 1.9570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7177 1.9570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.3598 3.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.3598 3.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 14 13 1 1 0 0 0 | + | 14 13 1 1 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 1 0 0 0 | + | 17 18 1 1 0 0 0 |
| − | 18 13 1 1 0 0 0 | + | 18 13 1 1 0 0 0 |
| − | 17 19 1 0 0 0 0 | + | 17 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 18 21 1 0 0 0 0 | + | 18 21 1 0 0 0 0 |
| − | 15 22 1 0 0 0 0 | + | 15 22 1 0 0 0 0 |
| − | 6 14 1 0 0 0 0 | + | 6 14 1 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 3 24 1 0 0 0 0 | + | 3 24 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 30 25 1 0 0 0 0 | + | 30 25 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 25 9 1 0 0 0 0 | + | 25 9 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 23 33 1 0 0 0 0 | + | 23 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 49 51 1 0 0 0 0 | + | 49 51 1 0 0 0 0 |
| − | 48 52 1 0 0 0 0 | + | 48 52 1 0 0 0 0 |
| − | 45 22 1 0 0 0 0 | + | 45 22 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 39 53 1 0 0 0 0 | + | 39 53 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 59 | + | M SBL 1 1 59 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 59 0.4588 -0.7947 | + | M SBV 1 59 0.4588 -0.7947 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FACCS0045 | + | ID FL3FACCS0045 |
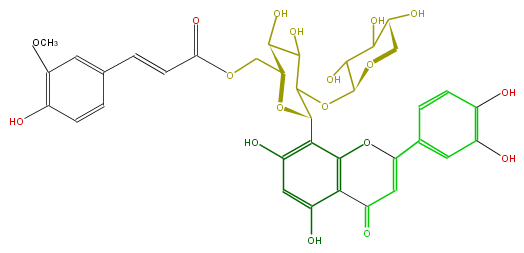
| − | FORMULA C36H36O18 | + | FORMULA C36H36O18 |
| − | EXACTMASS 756.190164348 | + | EXACTMASS 756.190164348 |
| − | AVERAGEMASS 756.6602399999999 | + | AVERAGEMASS 756.6602399999999 |
| − | SMILES COc(c(O)1)cc(C=CC(OCC(O3)C(O)C(O)C(C3c(c45)c(cc(O)c4C(=O)C=C(c(c6)ccc(c(O)6)O)O5)O)OC(O2)C(O)C(O)C(O)C2)=O)cc1 | + | SMILES COc(c(O)1)cc(C=CC(OCC(O3)C(O)C(O)C(C3c(c45)c(cc(O)c4C(=O)C=C(c(c6)ccc(c(O)6)O)O5)O)OC(O2)C(O)C(O)C(O)C2)=O)cc1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
0.7134 -1.0072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7134 -1.8322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4279 -2.2447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1424 -1.8322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1424 -1.0072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4279 -0.5948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8568 -2.2447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5713 -1.8322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5713 -1.0072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8568 -0.5948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8568 -2.8879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0008 -0.5949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6339 0.2969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4253 0.0088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0568 0.7661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1432 1.6086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3518 1.8967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7203 1.1393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5688 2.7066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9728 2.2444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0422 1.5387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7747 0.3425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6248 1.1190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4279 -3.0694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1795 -0.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9064 -0.9879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6334 -0.5682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6334 0.2711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9064 0.6908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1795 0.2711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3598 -0.9876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.3598 0.6906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4996 1.6132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4994 2.5154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2809 1.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0622 1.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8434 1.1623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5512 1.5710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2590 1.1623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2590 0.3451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5512 -0.0636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8434 0.3451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9655 -0.0628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3378 1.4586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5378 0.7122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9129 1.3877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5917 1.7661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3917 2.5125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0164 1.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9000 1.0208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0164 2.5137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0450 2.6876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7177 1.9570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.3598 3.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
14 13 1 1 0 0 0
14 15 1 0 0 0 0
15 16 1 0 0 0 0
16 17 1 0 0 0 0
17 18 1 1 0 0 0
18 13 1 1 0 0 0
17 19 1 0 0 0 0
16 20 1 0 0 0 0
18 21 1 0 0 0 0
15 22 1 0 0 0 0
6 14 1 0 0 0 0
21 23 1 0 0 0 0
3 24 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 29 1 0 0 0 0
29 30 2 0 0 0 0
30 25 1 0 0 0 0
27 31 1 0 0 0 0
25 9 1 0 0 0 0
28 32 1 0 0 0 0
23 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
44 45 1 0 0 0 0
45 46 1 1 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
48 49 1 0 0 0 0
49 44 1 0 0 0 0
44 50 1 0 0 0 0
49 51 1 0 0 0 0
48 52 1 0 0 0 0
45 22 1 0 0 0 0
53 54 1 0 0 0 0
39 53 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 59
M SMT 1 ^ OCH3
M SBV 1 59 0.4588 -0.7947
S SKP 5
ID FL3FACCS0045
FORMULA C36H36O18
EXACTMASS 756.190164348
AVERAGEMASS 756.6602399999999
SMILES COc(c(O)1)cc(C=CC(OCC(O3)C(O)C(O)C(C3c(c45)c(cc(O)c4C(=O)C=C(c(c6)ccc(c(O)6)O)O5)O)OC(O2)C(O)C(O)C(O)C2)=O)cc1
M END