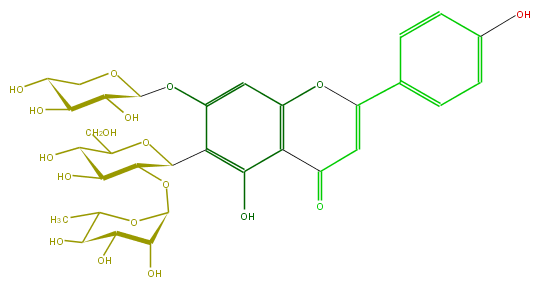
Mol:FL3FAADS0012
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 50 55 0 0 0 0 0 0 0 0999 V2000 | + | 50 55 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4267 0.8345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4267 0.8345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4267 -0.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4267 -0.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5784 -0.6346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5784 -0.6346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2697 -0.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2697 -0.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2697 0.8345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2697 0.8345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5784 1.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5784 1.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1179 -0.6346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1179 -0.6346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9660 -0.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9660 -0.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9660 0.8345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9660 0.8345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1179 1.3242 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1179 1.3242 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1179 -1.3981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1179 -1.3981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2398 1.2912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2398 1.2912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5784 -1.6135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5784 -1.6135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9081 1.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9081 1.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8019 0.8395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8019 0.8395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6957 1.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6957 1.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6957 2.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6957 2.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8019 2.9036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8019 2.9036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9081 2.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9081 2.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5890 2.9032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5890 2.9032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2643 -0.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2643 -0.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7363 -0.7980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7363 -0.7980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9761 -0.5023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9761 -0.5023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1839 -0.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1839 -0.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7757 0.0387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7757 0.0387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5521 -0.2402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5521 -0.2402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9150 -0.2990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9150 -0.2990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5196 -0.7169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5196 -0.7169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3328 -0.9031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3328 -0.9031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8028 -1.5464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8028 -1.5464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1997 -2.2338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1997 -2.2338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4366 -2.0157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4366 -2.0157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6688 -2.2338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6688 -2.2338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2718 -1.5464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2718 -1.5464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0351 -1.7645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0351 -1.7645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5798 -1.6833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5798 -1.6833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6976 -2.1733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6976 -2.1733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7781 -2.6071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7781 -2.6071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6688 -2.9036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6688 -2.9036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9809 1.4406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9809 1.4406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4531 0.7438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4531 0.7438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6928 1.0395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6928 1.0395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9005 1.0306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9005 1.0306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4924 1.5804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4924 1.5804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2688 1.3015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2688 1.3015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5890 1.2208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5890 1.2208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1422 0.7438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1422 0.7438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1783 0.6024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1783 0.6024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0580 0.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0580 0.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2730 0.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2730 0.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 3 13 1 0 0 0 0 | + | 3 13 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 14 9 1 0 0 0 0 | + | 14 9 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 24 2 1 0 0 0 0 | + | 24 2 1 0 0 0 0 |
| − | 40 41 1 1 0 0 0 | + | 40 41 1 1 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 43 42 1 1 0 0 0 | + | 43 42 1 1 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 40 1 0 0 0 0 | + | 45 40 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 12 1 0 0 0 0 | + | 43 12 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 26 49 1 0 0 0 0 | + | 26 49 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 49 50 | + | M SAL 1 2 49 50 |
| − | M SBL 1 1 55 | + | M SBL 1 1 55 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 55 0.5058 -0.5058 | + | M SBV 1 55 0.5058 -0.5058 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAADS0012 | + | ID FL3FAADS0012 |
| − | FORMULA C32H38O18 | + | FORMULA C32H38O18 |
| − | EXACTMASS 710.205814412 | + | EXACTMASS 710.205814412 |
| − | AVERAGEMASS 710.6333199999999 | + | AVERAGEMASS 710.6333199999999 |
| − | SMILES C(C(O)2)(C(c(c5OC(C6O)OCC(O)C6O)c(O)c(c(c5)3)C(C=C(c(c4)ccc(O)c4)O3)=O)OC(CO)C2O)OC(C1O)OC(C)C(C(O)1)O | + | SMILES C(C(O)2)(C(c(c5OC(C6O)OCC(O)C6O)c(O)c(c(c5)3)C(C=C(c(c4)ccc(O)c4)O3)=O)OC(CO)C2O)OC(C1O)OC(C)C(C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
50 55 0 0 0 0 0 0 0 0999 V2000
-1.4267 0.8345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4267 -0.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5784 -0.6346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2697 -0.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2697 0.8345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5784 1.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1179 -0.6346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9660 -0.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9660 0.8345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1179 1.3242 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1179 -1.3981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2398 1.2912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5784 -1.6135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9081 1.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8019 0.8395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6957 1.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6957 2.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8019 2.9036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9081 2.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5890 2.9032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2643 -0.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7363 -0.7980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9761 -0.5023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1839 -0.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7757 0.0387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5521 -0.2402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9150 -0.2990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5196 -0.7169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3328 -0.9031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8028 -1.5464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1997 -2.2338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4366 -2.0157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6688 -2.2338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2718 -1.5464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0351 -1.7645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5798 -1.6833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6976 -2.1733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7781 -2.6071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6688 -2.9036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9809 1.4406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4531 0.7438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6928 1.0395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9005 1.0306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4924 1.5804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2688 1.3015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5890 1.2208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1422 0.7438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1783 0.6024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0580 0.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2730 0.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
3 13 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
14 9 1 0 0 0 0
17 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 29 1 0 0 0 0
24 2 1 0 0 0 0
40 41 1 1 0 0 0
41 42 1 1 0 0 0
43 42 1 1 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 40 1 0 0 0 0
40 46 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 12 1 0 0 0 0
49 50 1 0 0 0 0
26 49 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 49 50
M SBL 1 1 55
M SMT 1 ^CH2OH
M SBV 1 55 0.5058 -0.5058
S SKP 5
ID FL3FAADS0012
FORMULA C32H38O18
EXACTMASS 710.205814412
AVERAGEMASS 710.6333199999999
SMILES C(C(O)2)(C(c(c5OC(C6O)OCC(O)C6O)c(O)c(c(c5)3)C(C=C(c(c4)ccc(O)c4)O3)=O)OC(CO)C2O)OC(C1O)OC(C)C(C(O)1)O
M END