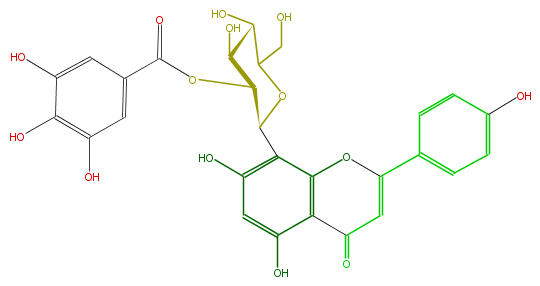
Mol:FL3FAACS0087
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.2877 -0.6948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2877 -0.6948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5732 -0.2823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5732 -0.2823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8587 -0.6948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8587 -0.6948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8587 -1.5198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8587 -1.5198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5732 -1.9323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5732 -1.9323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2877 -1.5198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2877 -1.5198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1443 -0.2823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1443 -0.2823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5702 -0.6948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5702 -0.6948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5702 -1.5198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5702 -1.5198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1443 -1.9323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1443 -1.9323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5732 -2.5043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5732 -2.5043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0891 -0.2321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0891 -0.2321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8035 -0.6446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8035 -0.6446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5180 -0.2321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5180 -0.2321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5180 0.5929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5180 0.5929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8035 1.0054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8035 1.0054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0891 0.5929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0891 0.5929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2015 0.9875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2015 0.9875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1443 -2.7023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1443 -2.7023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2607 -0.2961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2607 -0.2961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3692 2.4540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3692 2.4540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8577 1.7889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8577 1.7889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3453 1.1420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3453 1.1420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2341 0.3243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2341 0.3243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2545 0.9893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2545 0.9893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2578 1.6364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2578 1.6364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2155 2.0156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2155 2.0156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0059 2.7023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0059 2.7023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8577 2.4137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8577 2.4137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2155 2.6279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2155 2.6279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0528 0.5218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0528 0.5218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0399 1.3467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0399 1.3467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7478 1.7704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7478 1.7704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4686 1.3692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4686 1.3692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4816 0.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4816 0.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7737 0.1206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7737 0.1206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7866 -0.7032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7866 -0.7032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2015 0.1436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2015 0.1436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1756 1.7923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1756 1.7923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3200 1.7474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3200 1.7474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3070 2.5712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3070 2.5712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6130 1.3243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6130 1.3243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 5 11 2 0 0 0 0 | + | 5 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 10 19 1 0 0 0 0 | + | 10 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 7 24 1 0 0 0 0 | + | 7 24 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 42 23 1 0 0 0 0 | + | 42 23 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAACS0087 | + | ID FL3FAACS0087 |
| − | KNApSAcK_ID C00014088 | + | KNApSAcK_ID C00014088 |
| − | NAME Vitexin 2''-O-gallate | + | NAME Vitexin 2''-O-gallate |
| − | CAS_RN 267883-59-0 | + | CAS_RN 267883-59-0 |
| − | FORMULA C28H24O14 | + | FORMULA C28H24O14 |
| − | EXACTMASS 584.116605476 | + | EXACTMASS 584.116605476 |
| − | AVERAGEMASS 584.48176 | + | AVERAGEMASS 584.48176 |
| − | SMILES C(C(c(c(O)5)c(O3)c(c(c5)O)C(=O)C=C3c(c4)ccc(c4)O)2)(C(O)C(O)C(CO)O2)OC(=O)c(c1)cc(O)c(O)c1O | + | SMILES C(C(c(c(O)5)c(O3)c(c(c5)O)C(=O)C=C3c(c4)ccc(c4)O)2)(C(O)C(O)C(CO)O2)OC(=O)c(c1)cc(O)c(O)c1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
2.2877 -0.6948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5732 -0.2823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8587 -0.6948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8587 -1.5198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5732 -1.9323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2877 -1.5198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1443 -0.2823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5702 -0.6948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5702 -1.5198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1443 -1.9323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5732 -2.5043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0891 -0.2321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8035 -0.6446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5180 -0.2321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5180 0.5929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8035 1.0054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0891 0.5929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2015 0.9875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1443 -2.7023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2607 -0.2961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3692 2.4540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8577 1.7889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3453 1.1420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2341 0.3243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2545 0.9893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2578 1.6364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2155 2.0156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0059 2.7023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8577 2.4137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2155 2.6279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0528 0.5218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0399 1.3467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7478 1.7704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4686 1.3692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4816 0.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7737 0.1206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7866 -0.7032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2015 0.1436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1756 1.7923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3200 1.7474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3070 2.5712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6130 1.3243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
5 11 2 0 0 0 0
1 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
10 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
27 30 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
7 24 1 0 0 0 0
31 32 2 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
34 35 1 0 0 0 0
35 36 2 0 0 0 0
36 31 1 0 0 0 0
36 37 1 0 0 0 0
35 38 1 0 0 0 0
34 39 1 0 0 0 0
32 40 1 0 0 0 0
40 41 2 0 0 0 0
40 42 1 0 0 0 0
42 23 1 0 0 0 0
S SKP 8
ID FL3FAACS0087
KNApSAcK_ID C00014088
NAME Vitexin 2''-O-gallate
CAS_RN 267883-59-0
FORMULA C28H24O14
EXACTMASS 584.116605476
AVERAGEMASS 584.48176
SMILES C(C(c(c(O)5)c(O3)c(c(c5)O)C(=O)C=C3c(c4)ccc(c4)O)2)(C(O)C(O)C(CO)O2)OC(=O)c(c1)cc(O)c(O)c1O
M END