Mol:FL3FAACS0029
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.2205 -0.6952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2205 -0.6952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2205 -1.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2205 -1.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6642 -1.6587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6642 -1.6587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1079 -1.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1079 -1.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1079 -0.6952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1079 -0.6952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6642 -0.3740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6642 -0.3740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4484 -1.6587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4484 -1.6587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0047 -1.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0047 -1.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0047 -0.6952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0047 -0.6952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4484 -0.3740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4484 -0.3740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4484 -2.1596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4484 -2.1596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7766 -0.3741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7766 -0.3741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6227 -0.3535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6227 -0.3535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2089 -0.6919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2089 -0.6919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7951 -0.3535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7951 -0.3535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7951 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7951 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2089 0.6619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2089 0.6619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6227 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6227 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3810 0.6617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3810 0.6617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4394 2.1042 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.4394 2.1042 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.0450 1.6454 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.0450 1.6454 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.7881 0.9848 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.7881 0.9848 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.7812 0.3473 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.7812 0.3473 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.3179 0.8106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3179 0.8106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6229 1.3885 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.6229 1.3885 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.1213 2.1042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1213 2.1042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0798 2.3606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0798 2.3606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4235 0.6063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4235 0.6063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6642 -2.3008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6642 -2.3008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1736 -1.5644 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.1736 -1.5644 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.8024 -2.0544 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.8024 -2.0544 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.2679 -1.8465 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.2679 -1.8465 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.7521 -1.8409 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.7521 -1.8409 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.1269 -1.4661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1269 -1.4661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5945 -1.7129 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.5945 -1.7129 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.8321 -0.9730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8321 -0.9730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3810 -2.0825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3810 -2.0825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9616 -2.3606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9616 -2.3606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3413 -1.1542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3413 -1.1542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9237 -0.3413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9237 -0.3413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1076 1.6861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1076 1.6861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6069 1.2736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6069 1.2736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 9 1 0 0 0 0 | + | 13 9 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 2 1 0 0 0 0 | + | 33 2 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 25 41 1 0 0 0 0 | + | 25 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 45 -0.1076 1.6861 | + | M SVB 2 45 -0.1076 1.6861 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 43 -2.3413 -1.1542 | + | M SVB 1 43 -2.3413 -1.1542 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAACS0029 | + | ID FL3FAACS0029 |
| − | KNApSAcK_ID C00006234 | + | KNApSAcK_ID C00006234 |
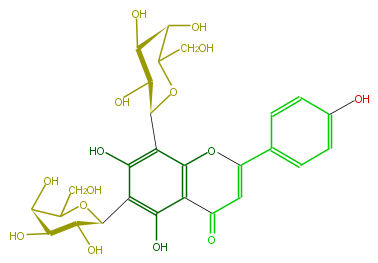
| − | NAME 6,8-Di-C-galactopyranosylapigenin | + | NAME 6,8-Di-C-galactopyranosylapigenin |
| − | CAS_RN 82043-11-6 | + | CAS_RN 82043-11-6 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES C(=C4c(c5)ccc(O)c5)C(c(c(O4)2)c(O)c(c(O)c2[C@H](O3)[C@H]([C@H]([C@H](C3CO)O)O)O)[C@H](O1)[C@H]([C@H]([C@H](C(CO)1)O)O)O)=O | + | SMILES C(=C4c(c5)ccc(O)c5)C(c(c(O4)2)c(O)c(c(O)c2[C@H](O3)[C@H]([C@H]([C@H](C3CO)O)O)O)[C@H](O1)[C@H]([C@H]([C@H](C(CO)1)O)O)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-1.2205 -0.6952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2205 -1.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6642 -1.6587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1079 -1.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1079 -0.6952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6642 -0.3740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4484 -1.6587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0047 -1.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0047 -0.6952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4484 -0.3740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4484 -2.1596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7766 -0.3741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6227 -0.3535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2089 -0.6919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7951 -0.3535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7951 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2089 0.6619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6227 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3810 0.6617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4394 2.1042 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.0450 1.6454 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.7881 0.9848 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.7812 0.3473 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.3179 0.8106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6229 1.3885 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.1213 2.1042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0798 2.3606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4235 0.6063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6642 -2.3008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1736 -1.5644 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.8024 -2.0544 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.2679 -1.8465 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.7521 -1.8409 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.1269 -1.4661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5945 -1.7129 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.8321 -0.9730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3810 -2.0825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9616 -2.3606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3413 -1.1542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9237 -0.3413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1076 1.6861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6069 1.2736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
13 9 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
6 23 1 0 0 0 0
3 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 2 1 0 0 0 0
35 39 1 0 0 0 0
39 40 1 0 0 0 0
25 41 1 0 0 0 0
41 42 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 45
M SMT 2 CH2OH
M SVB 2 45 -0.1076 1.6861
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 43
M SMT 1 CH2OH
M SVB 1 43 -2.3413 -1.1542
S SKP 8
ID FL3FAACS0029
KNApSAcK_ID C00006234
NAME 6,8-Di-C-galactopyranosylapigenin
CAS_RN 82043-11-6
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES C(=C4c(c5)ccc(O)c5)C(c(c(O4)2)c(O)c(c(O)c2[C@H](O3)[C@H]([C@H]([C@H](C3CO)O)O)O)[C@H](O1)[C@H]([C@H]([C@H](C(CO)1)O)O)O)=O
M END