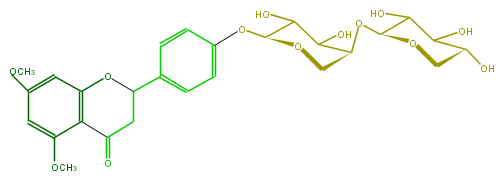
Mol:FL2FDAGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 40 44 0 0 0 0 0 0 0 0999 V2000 | + | 40 44 0 0 0 0 0 0 0 0999 V2000 |
| − | -5.4610 0.2153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4610 0.2153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4610 -0.5941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4610 -0.5941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7600 -0.9989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7600 -0.9989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0590 -0.5941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0590 -0.5941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0590 0.2153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0590 0.2153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7600 0.6200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7600 0.6200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3580 -0.9989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3580 -0.9989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6570 -0.5941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6570 -0.5941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6570 0.2153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6570 0.2153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3580 0.6200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3580 0.6200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9562 0.6199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9562 0.6199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2417 0.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2417 0.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5272 0.6199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5272 0.6199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5272 1.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5272 1.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2417 1.8573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2417 1.8573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9562 1.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9562 1.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3580 -1.6841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3580 -1.6841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2150 1.8734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2150 1.8734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3432 0.7959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3432 0.7959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1139 1.2409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1139 1.2409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2506 1.4570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2506 1.4570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6178 2.0828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6178 2.0828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8470 1.6379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8470 1.6379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7105 1.4217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7105 1.4217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3921 0.8940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3921 0.8940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1627 1.3390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1627 1.3390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2994 1.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2994 1.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6667 2.1809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6667 2.1809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8959 1.7360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8959 1.7360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7592 1.5198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7592 1.5198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3323 2.0341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3323 2.0341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8444 2.2753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8444 2.2753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8364 1.7470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8364 1.7470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8953 2.2956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8953 2.2956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0578 1.8163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0578 1.8163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6396 0.8415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6396 0.8415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.0096 0.7639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.0096 0.7639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.6396 1.8553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.6396 1.8553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7600 -1.7758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7600 -1.7758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8598 -2.2956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8598 -2.2956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 20 19 1 1 0 0 0 | + | 20 19 1 1 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 19 1 1 0 0 0 | + | 24 19 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 25 1 1 0 0 0 | + | 30 25 1 1 0 0 0 |
| − | 20 31 1 0 0 0 0 | + | 20 31 1 0 0 0 0 |
| − | 31 29 1 0 0 0 0 | + | 31 29 1 0 0 0 0 |
| − | 18 23 1 0 0 0 0 | + | 18 23 1 0 0 0 0 |
| − | 22 32 1 0 0 0 0 | + | 22 32 1 0 0 0 0 |
| − | 21 33 1 0 0 0 0 | + | 21 33 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 27 35 1 0 0 0 0 | + | 27 35 1 0 0 0 0 |
| − | 26 36 1 0 0 0 0 | + | 26 36 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 1 37 1 0 0 0 0 | + | 1 37 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 3 39 1 0 0 0 0 | + | 3 39 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 37 38 | + | M SAL 1 2 37 38 |
| − | M SBL 1 1 42 | + | M SBL 1 1 42 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 42 0.5486 -0.5486 | + | M SBV 1 42 0.5486 -0.5486 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 39 40 | + | M SAL 2 2 39 40 |
| − | M SBL 2 1 44 | + | M SBL 2 1 44 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 44 0.0000 0.7770 | + | M SBV 2 44 0.0000 0.7770 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FDAGS0001 | + | ID FL2FDAGS0001 |
| − | FORMULA C27H32O13 | + | FORMULA C27H32O13 |
| − | EXACTMASS 564.18429111 | + | EXACTMASS 564.18429111 |
| − | AVERAGEMASS 564.53518 | + | AVERAGEMASS 564.53518 |
| − | SMILES O(C(C5O)OCC(C5O)O)C(C4)C(O)C(O)C(O4)Oc(c1)ccc(C(C2)Oc(c3)c(c(OC)cc3OC)C2=O)c1 | + | SMILES O(C(C5O)OCC(C5O)O)C(C4)C(O)C(O)C(O4)Oc(c1)ccc(C(C2)Oc(c3)c(c(OC)cc3OC)C2=O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
40 44 0 0 0 0 0 0 0 0999 V2000
-5.4610 0.2153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4610 -0.5941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7600 -0.9989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0590 -0.5941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0590 0.2153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7600 0.6200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3580 -0.9989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6570 -0.5941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6570 0.2153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3580 0.6200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9562 0.6199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2417 0.2073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5272 0.6199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5272 1.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2417 1.8573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9562 1.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3580 -1.6841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2150 1.8734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3432 0.7959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1139 1.2409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2506 1.4570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6178 2.0828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8470 1.6379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7105 1.4217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3921 0.8940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1627 1.3390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2994 1.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6667 2.1809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8959 1.7360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7592 1.5198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3323 2.0341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8444 2.2753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8364 1.7470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8953 2.2956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.0578 1.8163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.6396 0.8415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.0096 0.7639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.6396 1.8553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7600 -1.7758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8598 -2.2956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
14 18 1 0 0 0 0
20 19 1 1 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 19 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 1 0 0 0
30 25 1 1 0 0 0
20 31 1 0 0 0 0
31 29 1 0 0 0 0
18 23 1 0 0 0 0
22 32 1 0 0 0 0
21 33 1 0 0 0 0
28 34 1 0 0 0 0
27 35 1 0 0 0 0
26 36 1 0 0 0 0
37 38 1 0 0 0 0
1 37 1 0 0 0 0
39 40 1 0 0 0 0
3 39 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 37 38
M SBL 1 1 42
M SMT 1 ^ OCH3
M SBV 1 42 0.5486 -0.5486
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 39 40
M SBL 2 1 44
M SMT 2 OCH3
M SBV 2 44 0.0000 0.7770
S SKP 5
ID FL2FDAGS0001
FORMULA C27H32O13
EXACTMASS 564.18429111
AVERAGEMASS 564.53518
SMILES O(C(C5O)OCC(C5O)O)C(C4)C(O)C(O)C(O4)Oc(c1)ccc(C(C2)Oc(c3)c(c(OC)cc3OC)C2=O)c1
M END