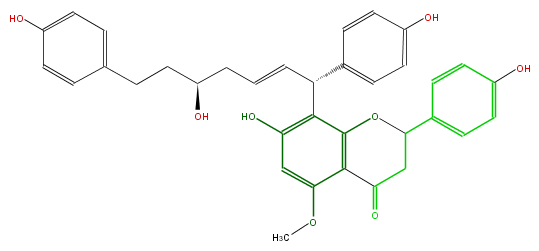
Mol:FL2FBANC0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.2961 -0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2961 -0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0103 0.2067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0103 0.2067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2961 -1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2961 -1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0113 -1.4433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0113 -1.4433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7255 -1.0304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7255 -1.0304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7250 -0.2054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7250 -0.2054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4402 -1.4425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4402 -1.4425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1544 -1.0296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1544 -1.0296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1540 -0.2046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1540 -0.2046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4393 0.2075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4393 0.2075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7829 0.1590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7829 0.1590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4976 -0.2531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4976 -0.2531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2118 0.1598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2118 0.1598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2113 0.9848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2113 0.9848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4966 1.3969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4966 1.3969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7824 0.9840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7824 0.9840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4406 -2.0954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4406 -2.0954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0117 -2.2551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0117 -2.2551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4186 0.2059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4186 0.2059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8112 1.3311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8112 1.3311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3929 -2.6124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3929 -2.6124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0103 0.9774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0103 0.9774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6988 1.3749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6988 1.3749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4133 0.9624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4133 0.9624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1277 1.3749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1277 1.3749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1277 2.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1277 2.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4133 2.6124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4133 2.6124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6988 2.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6988 2.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6770 2.5170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6770 2.5170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3551 1.3557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3551 1.3557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3484 0.9496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3484 0.9496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0056 1.3290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0056 1.3290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6801 0.9396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6801 0.9396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6801 0.2176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6801 0.2176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3847 1.3464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3847 1.3464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1093 0.9281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1093 0.9281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8436 1.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8436 1.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8436 2.1770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8436 2.1770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5581 2.5895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5581 2.5895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2725 2.1770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2725 2.1770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2725 1.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2725 1.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5581 0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5581 0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8112 2.4880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8112 2.4880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 4 18 1 0 0 0 0 | + | 4 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 18 21 1 0 0 0 0 | + | 18 21 1 0 0 0 0 |
| − | 2 22 1 0 0 0 0 | + | 2 22 1 0 0 0 0 |
| − | 22 23 1 6 0 0 0 | + | 22 23 1 6 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FBANC0008 | + | ID FL2FBANC0008 |
| − | KNApSAcK_ID C00014292 | + | KNApSAcK_ID C00014292 |
| − | NAME Epicalyxin D | + | NAME Epicalyxin D |
| − | CAS_RN 193816-93-2 | + | CAS_RN 193816-93-2 |
| − | FORMULA C35H34O8 | + | FORMULA C35H34O8 |
| − | EXACTMASS 582.225368064 | + | EXACTMASS 582.225368064 |
| − | AVERAGEMASS 582.63966 | + | AVERAGEMASS 582.63966 |
| − | SMILES c(O)(c1)ccc(C(C=CCC(O)CCc(c5)ccc(c5)O)c(c42)c(O)cc(OC)c(C(=O)CC(O4)c(c3)ccc(O)c3)2)c1 | + | SMILES c(O)(c1)ccc(C(C=CCC(O)CCc(c5)ccc(c5)O)c(c42)c(O)cc(OC)c(C(=O)CC(O4)c(c3)ccc(O)c3)2)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
0.2961 -0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0103 0.2067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2961 -1.0312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0113 -1.4433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7255 -1.0304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7250 -0.2054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4402 -1.4425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1544 -1.0296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1540 -0.2046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4393 0.2075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7829 0.1590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4976 -0.2531 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2118 0.1598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2113 0.9848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4966 1.3969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7824 0.9840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4406 -2.0954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0117 -2.2551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4186 0.2059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8112 1.3311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3929 -2.6124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0103 0.9774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6988 1.3749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4133 0.9624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1277 1.3749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1277 2.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4133 2.6124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6988 2.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6770 2.5170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3551 1.3557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3484 0.9496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0056 1.3290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6801 0.9396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6801 0.2176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3847 1.3464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1093 0.9281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8436 1.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8436 2.1770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5581 2.5895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2725 2.1770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2725 1.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5581 0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8112 2.4880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
4 18 1 0 0 0 0
1 19 1 0 0 0 0
14 20 1 0 0 0 0
18 21 1 0 0 0 0
2 22 1 0 0 0 0
22 23 1 6 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
26 29 1 0 0 0 0
22 30 1 0 0 0 0
30 31 2 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
33 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
S SKP 8
ID FL2FBANC0008
KNApSAcK_ID C00014292
NAME Epicalyxin D
CAS_RN 193816-93-2
FORMULA C35H34O8
EXACTMASS 582.225368064
AVERAGEMASS 582.63966
SMILES c(O)(c1)ccc(C(C=CCC(O)CCc(c5)ccc(c5)O)c(c42)c(O)cc(OC)c(C(=O)CC(O4)c(c3)ccc(O)c3)2)c1
M END