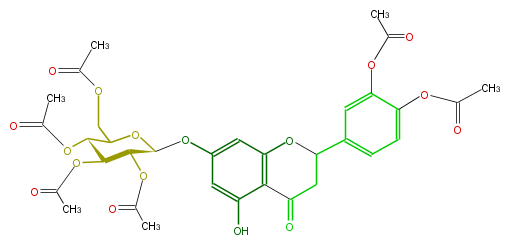
Mol:FL2FACGS0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 50 53 0 0 0 0 0 0 0 0999 V2000 | + | 50 53 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.7528 -1.2667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7528 -1.2667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2319 -1.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2319 -1.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2890 -1.2667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2890 -1.2667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2890 -0.6652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2890 -0.6652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2319 -0.3645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2319 -0.3645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7528 -0.6652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7528 -0.6652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8098 -1.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8098 -1.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3307 -1.2667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3307 -1.2667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3307 -0.6652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3307 -0.6652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8098 -0.3645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8098 -0.3645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8098 -2.0913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8098 -2.0913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2319 -2.1683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2319 -2.1683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9104 -0.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9104 -0.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4391 -0.6710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4391 -0.6710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9679 -0.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9679 -0.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9679 0.2448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9679 0.2448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4391 0.5500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4391 0.5500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9104 0.2448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9104 0.2448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2555 -0.3382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2555 -0.3382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9092 -0.7953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9092 -0.7953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4106 -0.6014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4106 -0.6014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8910 -0.6071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8910 -0.6071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2791 -0.2465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2791 -0.2465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7884 -0.4294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7884 -0.4294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6426 -0.5618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6426 -0.5618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4490 -0.8216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4490 -0.8216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1250 -1.0810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1250 -1.0810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0239 -0.1109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0239 -0.1109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2860 -0.3574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2860 -0.3574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2968 -1.7222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2968 -1.7222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7386 -1.7222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7386 -1.7222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0965 -2.0690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0965 -2.0690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8643 -1.3922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8643 -1.3922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3061 -1.3922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3061 -1.3922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6640 -1.7390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6640 -1.7390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0239 0.6358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0239 0.6358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4080 1.0200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4080 1.0200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9353 1.0200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9353 1.0200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1065 1.5423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1065 1.5423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1399 -0.0645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1399 -0.0645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9944 0.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9944 0.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7186 -0.0645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7186 -0.0645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4391 1.1473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4391 1.1473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7627 1.7077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7627 1.7077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4968 2.1683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4968 2.1683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2015 1.7077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2015 1.7077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4955 0.5494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4955 0.5494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1586 0.3717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1586 0.3717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1586 -0.1529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1586 -0.1529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7186 0.6950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7186 0.6950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 2 12 1 0 0 0 0 | + | 2 12 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 6 29 1 0 0 0 0 | + | 6 29 1 0 0 0 0 |
| − | 29 22 1 0 0 0 0 | + | 29 22 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 26 33 1 0 0 0 0 | + | 26 33 1 0 0 0 0 |
| − | 28 36 1 0 0 0 0 | + | 28 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 25 40 1 0 0 0 0 | + | 25 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 40 42 2 0 0 0 0 | + | 40 42 2 0 0 0 0 |
| − | 17 43 1 0 0 0 0 | + | 17 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 44 46 2 0 0 0 0 | + | 44 46 2 0 0 0 0 |
| − | 16 47 1 0 0 0 0 | + | 16 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 48 50 1 0 0 0 0 | + | 48 50 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FACGS0011 | + | ID FL2FACGS0011 |
| − | KNApSAcK_ID C00008526 | + | KNApSAcK_ID C00008526 |
| − | NAME Hexaacetylpyracanthoside | + | NAME Hexaacetylpyracanthoside |
| − | CAS_RN 138989-34-1 | + | CAS_RN 138989-34-1 |
| − | FORMULA C33H34O17 | + | FORMULA C33H34O17 |
| − | EXACTMASS 702.179599662 | + | EXACTMASS 702.179599662 |
| − | AVERAGEMASS 702.61286 | + | AVERAGEMASS 702.61286 |
| − | SMILES C(C1OC(C)=O)(COC(C)=O)OC(Oc(c4)cc(c3c4O)OC(CC(=O)3)c(c2)cc(c(c2)OC(C)=O)OC(C)=O)C(OC(C)=O)C1OC(C)=O | + | SMILES C(C1OC(C)=O)(COC(C)=O)OC(Oc(c4)cc(c3c4O)OC(CC(=O)3)c(c2)cc(c(c2)OC(C)=O)OC(C)=O)C(OC(C)=O)C1OC(C)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
50 53 0 0 0 0 0 0 0 0999 V2000
-0.7528 -1.2667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2319 -1.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2890 -1.2667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2890 -0.6652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2319 -0.3645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7528 -0.6652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8098 -1.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3307 -1.2667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3307 -0.6652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8098 -0.3645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8098 -2.0913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2319 -2.1683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9104 -0.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4391 -0.6710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9679 -0.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9679 0.2448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4391 0.5500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9104 0.2448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2555 -0.3382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9092 -0.7953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4106 -0.6014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8910 -0.6071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2791 -0.2465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7884 -0.4294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6426 -0.5618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4490 -0.8216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1250 -1.0810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0239 -0.1109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2860 -0.3574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2968 -1.7222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7386 -1.7222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0965 -2.0690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8643 -1.3922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3061 -1.3922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6640 -1.7390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0239 0.6358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4080 1.0200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9353 1.0200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1065 1.5423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1399 -0.0645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9944 0.4786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7186 -0.0645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4391 1.1473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7627 1.7077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4968 2.1683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2015 1.7077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4955 0.5494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1586 0.3717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1586 -0.1529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7186 0.6950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
2 12 1 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
24 28 1 0 0 0 0
6 29 1 0 0 0 0
29 22 1 0 0 0 0
27 30 1 0 0 0 0
30 31 2 0 0 0 0
30 32 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
26 33 1 0 0 0 0
28 36 1 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
37 39 1 0 0 0 0
25 40 1 0 0 0 0
40 41 1 0 0 0 0
40 42 2 0 0 0 0
17 43 1 0 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
44 46 2 0 0 0 0
16 47 1 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
48 50 1 0 0 0 0
S SKP 8
ID FL2FACGS0011
KNApSAcK_ID C00008526
NAME Hexaacetylpyracanthoside
CAS_RN 138989-34-1
FORMULA C33H34O17
EXACTMASS 702.179599662
AVERAGEMASS 702.61286
SMILES C(C1OC(C)=O)(COC(C)=O)OC(Oc(c4)cc(c3c4O)OC(CC(=O)3)c(c2)cc(c(c2)OC(C)=O)OC(C)=O)C(OC(C)=O)C1OC(C)=O
M END