Mol:FL2FACGM0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.3001 -1.9222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3001 -1.9222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5856 -2.3347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5856 -2.3347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8711 -1.9222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8711 -1.9222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8711 -1.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8711 -1.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5856 -0.6847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5856 -0.6847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3001 -1.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3001 -1.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1566 -2.3347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1566 -2.3347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5579 -1.9222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5579 -1.9222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5579 -1.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5579 -1.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1566 -0.6847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1566 -0.6847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1566 -3.0533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1566 -3.0533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5856 -3.1589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5856 -3.1589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9951 -0.6958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9951 -0.6958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3531 -0.6865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3531 -0.6865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0783 -1.1051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0783 -1.1051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8034 -0.6865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8034 -0.6865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8034 0.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8034 0.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0783 0.5697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0783 0.5697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3531 0.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3531 0.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2646 1.9965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2646 1.9965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7897 1.3696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7897 1.3696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1058 1.6356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1058 1.6356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3930 1.6278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3930 1.6278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9255 2.1224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9255 2.1224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6240 1.8715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6240 1.8715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2013 2.3899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2013 2.3899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5286 1.3941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5286 1.3941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4238 1.2239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4238 1.2239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0785 2.4184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0785 2.4184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4831 1.6771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4831 1.6771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2918 1.9916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2918 1.9916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0720 2.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0720 2.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5051 2.5672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5051 2.5672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7976 2.1938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7976 2.1938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6880 2.0493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6880 2.0493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2452 1.6731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2452 1.6731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4446 3.1589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4446 3.1589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3440 2.7456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3440 2.7456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0763 1.2858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0763 1.2858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5286 0.5697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5286 0.5697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5286 -1.1051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5286 -1.1051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7883 2.8181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7883 2.8181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8083 2.2522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8083 2.2522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 2 12 1 0 0 0 0 | + | 2 12 1 0 0 0 0 |
| − | 6 13 1 0 0 0 0 | + | 6 13 1 0 0 0 0 |
| − | 9 14 1 0 0 0 0 | + | 9 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 39 18 1 0 0 0 0 | + | 39 18 1 0 0 0 0 |
| − | 35 23 1 0 0 0 0 | + | 35 23 1 0 0 0 0 |
| − | 17 40 1 0 0 0 0 | + | 17 40 1 0 0 0 0 |
| − | 16 41 1 0 0 0 0 | + | 16 41 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 25 42 1 0 0 0 0 | + | 25 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 47 0.1642 -0.9465 | + | M SBV 1 47 0.1642 -0.9465 |
| − | S SKP 5 | + | S SKP 5 |
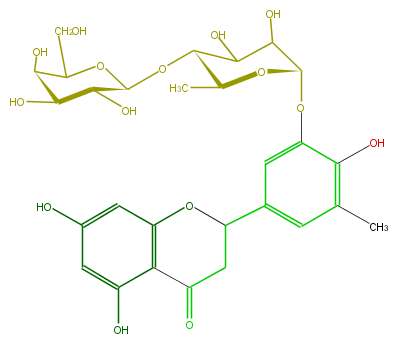
| − | ID FL2FACGM0002 | + | ID FL2FACGM0002 |
| − | FORMULA C28H34O15 | + | FORMULA C28H34O15 |
| − | EXACTMASS 610.189770418 | + | EXACTMASS 610.189770418 |
| − | AVERAGEMASS 610.56056 | + | AVERAGEMASS 610.56056 |
| − | SMILES C(O)C(C(O)5)OC(C(O)C(O)5)OC(C(C)4)C(O)C(C(O4)Oc(c1)c(c(C)cc1C(C3)Oc(c2C3=O)cc(O)cc(O)2)O)O | + | SMILES C(O)C(C(O)5)OC(C(O)C(O)5)OC(C(C)4)C(O)C(C(O4)Oc(c1)c(c(C)cc1C(C3)Oc(c2C3=O)cc(O)cc(O)2)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.3001 -1.9222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5856 -2.3347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8711 -1.9222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8711 -1.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5856 -0.6847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3001 -1.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1566 -2.3347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5579 -1.9222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5579 -1.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1566 -0.6847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1566 -3.0533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5856 -3.1589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9951 -0.6958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3531 -0.6865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0783 -1.1051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8034 -0.6865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8034 0.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0783 0.5697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3531 0.1510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2646 1.9965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7897 1.3696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1058 1.6356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3930 1.6278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9255 2.1224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6240 1.8715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2013 2.3899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5286 1.3941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4238 1.2239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0785 2.4184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4831 1.6771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2918 1.9916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0720 2.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5051 2.5672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7976 2.1938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6880 2.0493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2452 1.6731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4446 3.1589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3440 2.7456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0763 1.2858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5286 0.5697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5286 -1.1051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7883 2.8181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8083 2.2522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
2 12 1 0 0 0 0
6 13 1 0 0 0 0
9 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
33 37 1 0 0 0 0
34 38 1 0 0 0 0
32 39 1 0 0 0 0
39 18 1 0 0 0 0
35 23 1 0 0 0 0
17 40 1 0 0 0 0
16 41 1 0 0 0 0
42 43 1 0 0 0 0
25 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 ^CH2OH
M SBV 1 47 0.1642 -0.9465
S SKP 5
ID FL2FACGM0002
FORMULA C28H34O15
EXACTMASS 610.189770418
AVERAGEMASS 610.56056
SMILES C(O)C(C(O)5)OC(C(O)C(O)5)OC(C(C)4)C(O)C(C(O4)Oc(c1)c(c(C)cc1C(C3)Oc(c2C3=O)cc(O)cc(O)2)O)O
M END