Mol:FL1CG9NS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 26 27 0 0 0 0 0 0 0 0999 V2000 | + | 26 27 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8725 0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8725 0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8725 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8725 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3162 -0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3162 -0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7599 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7599 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7599 0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7599 0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3162 0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3162 0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2036 -0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2036 -0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3527 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3527 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9088 -0.6422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9088 -0.6422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4649 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4649 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0259 -0.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0259 -0.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5869 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5869 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5869 0.3266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5869 0.3266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0259 0.6505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0259 0.6505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4649 0.3266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4649 0.3266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2036 -1.1437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2036 -1.1437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0381 1.2154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0381 1.2154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6015 2.1150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6015 2.1150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3162 -1.6424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3162 -1.6424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3162 -2.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3162 -2.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1061 0.8212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1061 0.8212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9721 1.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9721 1.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2297 0.9399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2297 0.9399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7297 1.8059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7297 1.8059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5869 -0.2297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5869 -0.2297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5788 -0.1027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5788 -0.1027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 6 17 1 0 0 0 0 | + | 6 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 1 23 1 0 0 0 0 | + | 1 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 2 25 1 0 0 0 0 | + | 2 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 25 26 | + | M SAL 5 2 25 26 |
| − | M SBL 5 1 26 | + | M SBL 5 1 26 |
| − | M SMT 5 OCH3 | + | M SMT 5 OCH3 |
| − | M SVB 5 26 -2.5869 -0.2297 | + | M SVB 5 26 -2.5869 -0.2297 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 23 24 | + | M SAL 4 2 23 24 |
| − | M SBL 4 1 24 | + | M SBL 4 1 24 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 24 -2.2297 0.9399 | + | M SVB 4 24 -2.2297 0.9399 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 21 22 | + | M SAL 3 2 21 22 |
| − | M SBL 3 1 22 | + | M SBL 3 1 22 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 22 -0.2038 0.6422 | + | M SVB 3 22 -0.2038 0.6422 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 19 20 | + | M SAL 2 2 19 20 |
| − | M SBL 2 1 20 | + | M SBL 2 1 20 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 20 -1.7526 -0.8029 | + | M SVB 2 20 -1.7526 -0.8029 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 17 18 | + | M SAL 1 2 17 18 |
| − | M SBL 1 1 18 | + | M SBL 1 1 18 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 18 -1.0381 1.2154 | + | M SVB 1 18 -1.0381 1.2154 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1CG9NS0005 | + | ID FL1CG9NS0005 |
| − | KNApSAcK_ID C00006986 | + | KNApSAcK_ID C00006986 |
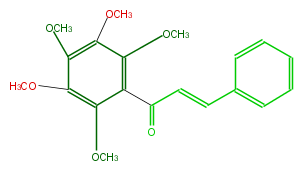
| − | NAME Pedicellin | + | NAME Pedicellin |
| − | CAS_RN 518-58-1 | + | CAS_RN 518-58-1 |
| − | FORMULA C20H22O6 | + | FORMULA C20H22O6 |
| − | EXACTMASS 358.141638436 | + | EXACTMASS 358.141638436 |
| − | AVERAGEMASS 358.38508 | + | AVERAGEMASS 358.38508 |
| − | SMILES O(C)c(c2OC)c(c(OC)c(c2OC)OC)C(=O)C=Cc(c1)cccc1 | + | SMILES O(C)c(c2OC)c(c(OC)c(c2OC)OC)C(=O)C=Cc(c1)cccc1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
26 27 0 0 0 0 0 0 0 0999 V2000
-1.8725 0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8725 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3162 -0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7599 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7599 0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3162 0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2036 -0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3527 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9088 -0.6422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4649 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0259 -0.6451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5869 -0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5869 0.3266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0259 0.6505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4649 0.3266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2036 -1.1437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0381 1.2154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6015 2.1150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3162 -1.6424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3162 -2.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1061 0.8212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9721 1.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2297 0.9399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7297 1.8059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5869 -0.2297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5788 -0.1027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
6 17 1 0 0 0 0
17 18 1 0 0 0 0
3 19 1 0 0 0 0
19 20 1 0 0 0 0
5 21 1 0 0 0 0
21 22 1 0 0 0 0
1 23 1 0 0 0 0
23 24 1 0 0 0 0
2 25 1 0 0 0 0
25 26 1 0 0 0 0
M STY 1 5 SUP
M SLB 1 5 5
M SAL 5 2 25 26
M SBL 5 1 26
M SMT 5 OCH3
M SVB 5 26 -2.5869 -0.2297
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 23 24
M SBL 4 1 24
M SMT 4 OCH3
M SVB 4 24 -2.2297 0.9399
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 21 22
M SBL 3 1 22
M SMT 3 OCH3
M SVB 3 22 -0.2038 0.6422
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 19 20
M SBL 2 1 20
M SMT 2 OCH3
M SVB 2 20 -1.7526 -0.8029
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 17 18
M SBL 1 1 18
M SMT 1 OCH3
M SVB 1 18 -1.0381 1.2154
S SKP 8
ID FL1CG9NS0005
KNApSAcK_ID C00006986
NAME Pedicellin
CAS_RN 518-58-1
FORMULA C20H22O6
EXACTMASS 358.141638436
AVERAGEMASS 358.38508
SMILES O(C)c(c2OC)c(c(OC)c(c2OC)OC)C(=O)C=Cc(c1)cccc1
M END