Mol:FL1C3CGS0023
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 59 0 0 0 0 0 0 0 0999 V2000 | + | 55 59 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.3343 -0.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3343 -0.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3343 -0.9023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3343 -0.9023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0488 -1.3148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0488 -1.3148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7632 -0.9023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7632 -0.9023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7632 -0.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7632 -0.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0488 0.3352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0488 0.3352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4777 -1.3148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4777 -1.3148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1922 -0.9023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1922 -0.9023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1922 -0.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1922 -0.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9067 0.3352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9067 0.3352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6211 -0.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6211 -0.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3356 0.3352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3356 0.3352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3356 1.1602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3356 1.1602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6211 1.5727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6211 1.5727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9067 1.1602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9067 1.1602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4777 -2.1398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4777 -2.1398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0381 0.5012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0381 0.5012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5600 -0.1715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5600 -0.1715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7849 0.1118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7849 0.1118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0264 -0.0394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0264 -0.0394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4516 0.6334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4516 0.6334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2268 0.3501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2268 0.3501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4382 0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4382 0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4737 -0.0253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4737 -0.0253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0311 0.0442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0311 0.0442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8954 -0.5414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8954 -0.5414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0488 -2.0261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0488 -2.0261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6663 0.3083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6663 0.3083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0168 1.5535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0168 1.5535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5968 -1.2323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5968 -1.2323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6211 2.2987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6211 2.2987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0713 1.2944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0713 1.2944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4832 2.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4832 2.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0713 2.7214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0713 2.7214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3071 2.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3071 2.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7191 1.2944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7191 1.2944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5429 1.2944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5429 1.2944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9554 2.0089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9554 2.0089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7804 2.0089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7804 2.0089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1929 1.2944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1929 1.2944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7804 0.5800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7804 0.5800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9554 0.5800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9554 0.5800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.0168 1.2944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.0168 1.2944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3131 -1.2935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3131 -1.2935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8950 -2.0178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8950 -2.0178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1370 -1.2935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1370 -1.2935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5490 -2.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5490 -2.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3728 -2.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3728 -2.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7853 -1.2925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7853 -1.2925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6103 -1.2925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6103 -1.2925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0228 -2.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0228 -2.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6103 -2.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6103 -2.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7853 -2.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7853 -2.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8467 -2.0070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8467 -2.0070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0223 -0.5790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0223 -0.5790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 17 18 1 1 0 0 0 | + | 17 18 1 1 0 0 0 |
| − | 18 19 1 1 0 0 0 | + | 18 19 1 1 0 0 0 |
| − | 20 19 1 1 0 0 0 | + | 20 19 1 1 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 17 1 0 0 0 0 | + | 22 17 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 17 24 1 0 0 0 0 | + | 17 24 1 0 0 0 0 |
| − | 18 25 1 0 0 0 0 | + | 18 25 1 0 0 0 0 |
| − | 19 26 1 0 0 0 0 | + | 19 26 1 0 0 0 0 |
| − | 3 27 1 0 0 0 0 | + | 3 27 1 0 0 0 0 |
| − | 1 28 1 0 0 0 0 | + | 1 28 1 0 0 0 0 |
| − | 13 29 1 0 0 0 0 | + | 13 29 1 0 0 0 0 |
| − | 2 30 1 0 0 0 0 | + | 2 30 1 0 0 0 0 |
| − | 14 31 1 0 0 0 0 | + | 14 31 1 0 0 0 0 |
| − | 20 28 1 0 0 0 0 | + | 20 28 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 32 23 1 0 0 0 0 | + | 32 23 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 53 48 1 0 0 0 0 | + | 53 48 1 0 0 0 0 |
| − | 51 54 1 0 0 0 0 | + | 51 54 1 0 0 0 0 |
| − | 50 55 1 0 0 0 0 | + | 50 55 1 0 0 0 0 |
| − | 26 44 1 0 0 0 0 | + | 26 44 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1C3CGS0023 | + | ID FL1C3CGS0023 |
| − | KNApSAcK_ID C00014511 | + | KNApSAcK_ID C00014511 |
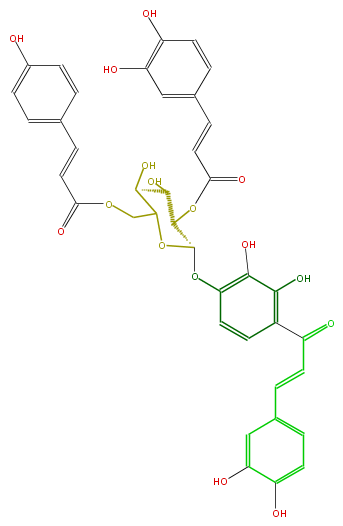
| − | NAME Okanin 4'-O-(2''-O-caffeoyl-6''-O-p-coumaroylglucoside);3,4,2',3',4'-Pentahydroxychalcone 4'-O-(2''-O-caffeoyl-6''-O-p-coumaroylglucoside) | + | NAME Okanin 4'-O-(2''-O-caffeoyl-6''-O-p-coumaroylglucoside);3,4,2',3',4'-Pentahydroxychalcone 4'-O-(2''-O-caffeoyl-6''-O-p-coumaroylglucoside) |
| − | CAS_RN 142628-31-7 | + | CAS_RN 142628-31-7 |
| − | FORMULA C39H34O16 | + | FORMULA C39H34O16 |
| − | EXACTMASS 758.18468504 | + | EXACTMASS 758.18468504 |
| − | AVERAGEMASS 758.6776600000001 | + | AVERAGEMASS 758.6776600000001 |
| − | SMILES Oc(c5)ccc(c5)C=CC(OCC(C4O)OC(C(C4O)OC(C=Cc(c3)cc(O)c(O)c3)=O)Oc(c(O)2)ccc(c2O)C(=O)C=Cc(c1)ccc(c1O)O)=O | + | SMILES Oc(c5)ccc(c5)C=CC(OCC(C4O)OC(C(C4O)OC(C=Cc(c3)cc(O)c(O)c3)=O)Oc(c(O)2)ccc(c2O)C(=O)C=Cc(c1)ccc(c1O)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 59 0 0 0 0 0 0 0 0999 V2000
1.3343 -0.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3343 -0.9023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0488 -1.3148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7632 -0.9023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7632 -0.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0488 0.3352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4777 -1.3148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1922 -0.9023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1922 -0.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9067 0.3352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6211 -0.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3356 0.3352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3356 1.1602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6211 1.5727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9067 1.1602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4777 -2.1398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0381 0.5012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5600 -0.1715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7849 0.1118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0264 -0.0394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4516 0.6334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2268 0.3501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4382 0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4737 -0.0253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0311 0.0442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8954 -0.5414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0488 -2.0261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6663 0.3083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.0168 1.5535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5968 -1.2323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.6211 2.2987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0713 1.2944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4832 2.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0713 2.7214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3071 2.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7191 1.2944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5429 1.2944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9554 2.0089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7804 2.0089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.1929 1.2944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7804 0.5800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9554 0.5800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.0168 1.2944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3131 -1.2935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8950 -2.0178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1370 -1.2935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5490 -2.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3728 -2.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7853 -1.2925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6103 -1.2925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0228 -2.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6103 -2.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7853 -2.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8467 -2.0070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0223 -0.5790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
17 18 1 1 0 0 0
18 19 1 1 0 0 0
20 19 1 1 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
22 17 1 0 0 0 0
22 23 1 0 0 0 0
17 24 1 0 0 0 0
18 25 1 0 0 0 0
19 26 1 0 0 0 0
3 27 1 0 0 0 0
1 28 1 0 0 0 0
13 29 1 0 0 0 0
2 30 1 0 0 0 0
14 31 1 0 0 0 0
20 28 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
32 23 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 52 1 0 0 0 0
52 53 2 0 0 0 0
53 48 1 0 0 0 0
51 54 1 0 0 0 0
50 55 1 0 0 0 0
26 44 1 0 0 0 0
S SKP 8
ID FL1C3CGS0023
KNApSAcK_ID C00014511
NAME Okanin 4'-O-(2''-O-caffeoyl-6''-O-p-coumaroylglucoside);3,4,2',3',4'-Pentahydroxychalcone 4'-O-(2''-O-caffeoyl-6''-O-p-coumaroylglucoside)
CAS_RN 142628-31-7
FORMULA C39H34O16
EXACTMASS 758.18468504
AVERAGEMASS 758.6776600000001
SMILES Oc(c5)ccc(c5)C=CC(OCC(C4O)OC(C(C4O)OC(C=Cc(c3)cc(O)c(O)c3)=O)Oc(c(O)2)ccc(c2O)C(=O)C=Cc(c1)ccc(c1O)O)=O
M END