Mol:BMCCPTPTe009
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 58 0 0 1 0 0 0 0 0999 V2000 | + | 55 58 0 0 1 0 0 0 0 0999 V2000 |
| − | 3.7321 1.9333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 1.9333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 1.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 1.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 0.4333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 0.4333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 -0.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 -0.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4641 -0.0667 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4641 -0.0667 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3301 0.4333 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 6.3301 0.4333 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 6.3301 1.4333 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 6.3301 1.4333 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 5.4641 1.9333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4641 1.9333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1962 -0.0667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 7.1962 -0.0667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 8.0622 0.4333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0622 0.4333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 1.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 1.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 0.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 0.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 1.9333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 1.9333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4641 -1.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4641 -1.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1962 1.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1962 1.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1962 -1.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1962 -1.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9282 -0.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9282 -0.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9282 -1.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9282 -1.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7942 -1.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7942 -1.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6603 -1.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6603 -1.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6603 -0.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6603 -0.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7942 0.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7942 0.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.5263 -1.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.5263 -1.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.3923 -1.0667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 12.3923 -1.0667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 13.2583 -1.5667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 13.2583 -1.5667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 14.1244 -1.0667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 14.1244 -1.0667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 14.9904 -1.5667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 14.9904 -1.5667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 16.7224 -1.5667 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 16.7224 -1.5667 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 16.8270 -2.5612 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 16.8270 -2.5612 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 17.8051 -2.7691 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 17.8051 -2.7691 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 18.3051 -1.9031 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 18.3051 -1.9031 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 19.2996 -1.7986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.2996 -1.7986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.0967 0.3421 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.0967 0.3421 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.1021 0.2376 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 22.1021 0.2376 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 21.5144 1.0466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.5144 1.0466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.9211 1.9601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.9211 1.9601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.3333 2.7691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.3333 2.7691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 -1.0667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 -1.0667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.3923 -0.0667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.3923 -0.0667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.2583 -2.5667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.2583 -2.5667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.1244 -0.0667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.1244 -0.0667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.9904 -2.5667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.9904 -2.5667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.8564 -1.0667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.8564 -1.0667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.0838 -3.2304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.0838 -3.2304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.2118 -3.6827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.2118 -3.6827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.6360 -1.1600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.6360 -1.1600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.7064 -0.8850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.7064 -0.8850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.7009 -0.7805 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.7009 -0.7805 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.8054 -1.7750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.8054 -1.7750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.5964 0.2140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.5964 0.2140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.6954 -0.6760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.6954 -0.6760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.6845 -0.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.6845 -0.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.5034 1.2556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.5034 1.2556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.3388 2.6646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.3388 2.6646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.7400 3.6827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.7400 3.6827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 29 28 1 0 0 0 0 | + | 29 28 1 0 0 0 0 |
| − | 28 46 1 6 0 0 0 | + | 28 46 1 6 0 0 0 |
| − | 31 46 1 6 0 0 0 | + | 31 46 1 6 0 0 0 |
| − | 31 30 1 0 0 0 0 | + | 31 30 1 0 0 0 0 |
| − | 30 29 1 0 0 0 0 | + | 30 29 1 0 0 0 0 |
| − | 28 43 1 0 0 0 0 | + | 28 43 1 0 0 0 0 |
| − | 29 44 1 1 0 0 0 | + | 29 44 1 1 0 0 0 |
| − | 30 45 1 1 0 0 0 | + | 30 45 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 47 1 0 0 0 0 | + | 32 47 1 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 3 2 1 0 0 0 0 | + | 3 2 1 0 0 0 0 |
| − | 2 1 2 0 0 0 0 | + | 2 1 2 0 0 0 0 |
| − | 1 11 1 0 0 0 0 | + | 1 11 1 0 0 0 0 |
| − | 12 4 1 0 0 0 0 | + | 12 4 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 11 8 1 0 0 0 0 | + | 11 8 1 0 0 0 0 |
| − | 8 7 1 0 0 0 0 | + | 8 7 1 0 0 0 0 |
| − | 6 7 1 4 0 0 0 | + | 6 7 1 4 0 0 0 |
| − | 6 5 1 0 0 0 0 | + | 6 5 1 0 0 0 0 |
| − | 5 12 1 0 0 0 0 | + | 5 12 1 0 0 0 0 |
| − | 4 38 2 0 0 0 0 | + | 4 38 2 0 0 0 0 |
| − | 2 13 1 0 0 0 0 | + | 2 13 1 0 0 0 0 |
| − | 6 9 1 0 0 0 0 | + | 6 9 1 0 0 0 0 |
| − | 7 15 1 4 0 0 0 | + | 7 15 1 4 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 9 16 1 4 0 0 0 | + | 9 16 1 4 0 0 0 |
| − | 10 17 1 0 0 0 0 | + | 10 17 1 0 0 0 0 |
| − | 22 21 2 0 0 0 0 | + | 22 21 2 0 0 0 0 |
| − | 22 17 1 0 0 0 0 | + | 22 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 2 0 0 0 0 | + | 19 20 2 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 5 14 1 0 0 0 0 | + | 5 14 1 0 0 0 0 |
| − | 20 23 1 0 0 0 0 | + | 20 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 24 39 1 4 0 0 0 | + | 24 39 1 4 0 0 0 |
| − | 25 40 1 4 0 0 0 | + | 25 40 1 4 0 0 0 |
| − | 26 41 1 4 0 0 0 | + | 26 41 1 4 0 0 0 |
| − | 27 43 1 0 0 0 0 | + | 27 43 1 0 0 0 0 |
| − | 27 42 1 4 0 0 0 | + | 27 42 1 4 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 48 50 2 0 0 0 0 | + | 48 50 2 0 0 0 0 |
| − | 34 51 1 4 0 0 0 | + | 34 51 1 4 0 0 0 |
| − | 33 52 1 0 0 0 0 | + | 33 52 1 0 0 0 0 |
| − | 33 53 2 0 0 0 0 | + | 33 53 2 0 0 0 0 |
| − | 34 33 1 0 0 0 0 | + | 34 33 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 55 1 0 0 0 0 | + | 37 55 1 0 0 0 0 |
| − | 37 54 2 0 0 0 0 | + | 37 54 2 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMCCPTPTe009 | + | ID BMCCPTPTe009 |
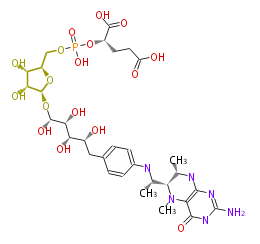
| − | NAME 5-Methyl-5,6,7,8-tetrahydro-methanopterin | + | NAME 5-Methyl-5,6,7,8-tetrahydro-methanopterin |
| − | FORMULA C31H47N6O17P | + | FORMULA C31H47N6O17P |
| − | EXACTMASS 806.2735 | + | EXACTMASS 806.2735 |
| − | AVERAGEMASS 806.7088 | + | AVERAGEMASS 806.7088 |
| − | SMILES c(c3)(ccc(CC(O)C(O)C(O)C(O[C@H](O4)[C@H](O)[C@@H]([C@@H](COP(O)(=O)OC(CCC(O)=O)C(O)=O)4)O)O)c3)NC(C)C(C(C)1)N(C)C(C(=O)2)=C(N=C(N)N2)N1 | + | SMILES c(c3)(ccc(CC(O)C(O)C(O)C(O[C@H](O4)[C@H](O)[C@@H]([C@@H](COP(O)(=O)OC(CCC(O)=O)C(O)=O)4)O)O)c3)NC(C)C(C(C)1)N(C)C(C(=O)2)=C(N=C(N)N2)N1 |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C04488 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C04488 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 58 0 0 1 0 0 0 0 0999 V2000
3.7321 1.9333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 1.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 0.4333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 -0.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4641 -0.0667 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.3301 0.4333 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
6.3301 1.4333 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
5.4641 1.9333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
7.1962 -0.0667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
8.0622 0.4333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 1.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 0.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 1.9333 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
5.4641 -1.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1962 1.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1962 -1.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.9282 -0.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.9282 -1.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.7942 -1.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.6603 -1.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.6603 -0.0667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.7942 0.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.5263 -1.5667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.3923 -1.0667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
13.2583 -1.5667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
14.1244 -1.0667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
14.9904 -1.5667 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
16.7224 -1.5667 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
16.8270 -2.5612 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
17.8051 -2.7691 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
18.3051 -1.9031 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
19.2996 -1.7986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
23.0967 0.3421 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
22.1021 0.2376 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
21.5144 1.0466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
21.9211 1.9601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
21.3333 2.7691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 -1.0667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.3923 -0.0667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.2583 -2.5667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.1244 -0.0667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.9904 -2.5667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
15.8564 -1.0667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
16.0838 -3.2304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.2118 -3.6827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.6360 -1.1600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.7064 -0.8850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.7009 -0.7805 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
20.8054 -1.7750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.5964 0.2140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.6954 -0.6760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.6845 -0.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.5034 1.2556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.3388 2.6646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.7400 3.6827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
29 28 1 0 0 0 0
28 46 1 6 0 0 0
31 46 1 6 0 0 0
31 30 1 0 0 0 0
30 29 1 0 0 0 0
28 43 1 0 0 0 0
29 44 1 1 0 0 0
30 45 1 1 0 0 0
31 32 1 0 0 0 0
32 47 1 0 0 0 0
3 4 1 0 0 0 0
3 2 1 0 0 0 0
2 1 2 0 0 0 0
1 11 1 0 0 0 0
12 4 1 0 0 0 0
11 12 2 0 0 0 0
11 8 1 0 0 0 0
8 7 1 0 0 0 0
6 7 1 4 0 0 0
6 5 1 0 0 0 0
5 12 1 0 0 0 0
4 38 2 0 0 0 0
2 13 1 0 0 0 0
6 9 1 0 0 0 0
7 15 1 4 0 0 0
9 10 1 0 0 0 0
9 16 1 4 0 0 0
10 17 1 0 0 0 0
22 21 2 0 0 0 0
22 17 1 0 0 0 0
17 18 2 0 0 0 0
18 19 1 0 0 0 0
19 20 2 0 0 0 0
20 21 1 0 0 0 0
5 14 1 0 0 0 0
20 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
24 39 1 4 0 0 0
25 40 1 4 0 0 0
26 41 1 4 0 0 0
27 43 1 0 0 0 0
27 42 1 4 0 0 0
47 48 1 0 0 0 0
48 51 1 0 0 0 0
48 49 1 0 0 0 0
48 50 2 0 0 0 0
34 51 1 4 0 0 0
33 52 1 0 0 0 0
33 53 2 0 0 0 0
34 33 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 55 1 0 0 0 0
37 54 2 0 0 0 0
S SKP 7
ID BMCCPTPTe009
NAME 5-Methyl-5,6,7,8-tetrahydro-methanopterin
FORMULA C31H47N6O17P
EXACTMASS 806.2735
AVERAGEMASS 806.7088
SMILES c(c3)(ccc(CC(O)C(O)C(O)C(O[C@H](O4)[C@H](O)[C@@H]([C@@H](COP(O)(=O)OC(CCC(O)=O)C(O)=O)4)O)O)c3)NC(C)C(C(C)1)N(C)C(C(=O)2)=C(N=C(N)N2)N1
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C04488
M END