Mol:BMCCPPPR0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 50 0 0 0 0 0 0 0 0999 V2000 | + | 43 50 0 0 0 0 0 0 0 0999 V2000 |
| − | 7.1238 2.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1238 2.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3444 3.1173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3444 3.1173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0580 3.1173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0580 3.1173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2785 2.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2785 2.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0672 2.2273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0672 2.2273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2785 1.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2785 1.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9573 1.2181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9573 1.2181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9573 0.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9573 0.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2785 0.2839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2785 0.2839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.0672 -0.5047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.0672 -0.5047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2785 -0.7161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2785 -0.7161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0580 -1.3948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0580 -1.3948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3444 -1.3948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3444 -1.3948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1238 -0.7161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1238 -0.7161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3352 -0.5047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3352 -0.5047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1238 0.2839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1238 0.2839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4451 0.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4451 0.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4451 1.2181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4451 1.2181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1238 1.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1238 1.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3352 2.2273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3352 2.2273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7012 1.8613 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7012 1.8613 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7012 0.8613 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7012 0.8613 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7012 -0.1387 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.7012 -0.1387 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7012 0.8613 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7012 0.8613 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7566 3.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7566 3.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6458 3.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6458 3.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2391 4.8399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2391 4.8399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.7663 1.8059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.7663 1.8059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.7663 -0.0833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.7663 -0.0833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.6798 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.6798 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.6458 -2.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.6458 -2.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7566 -2.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7566 -2.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.1633 -3.1173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.1633 -3.1173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5755 -3.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5755 -3.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6361 -0.0833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6361 -0.0833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7226 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7226 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9135 -0.2644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9135 -0.2644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6361 1.8059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6361 1.8059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.7012 0.8613 0.0000 Mg 0 2 0 0 0 0 0 0 0 0 0 0 | + | 7.7012 0.8613 0.0000 Mg 0 2 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5810 -3.8218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5810 -3.8218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9823 -4.8399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9823 -4.8399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0181 -1.2589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0181 -1.2589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 0.1424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 0.1424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 4 21 1 0 0 0 0 | + | 4 21 1 0 0 0 0 |
| − | 22 6 1 0 0 0 0 | + | 22 6 1 0 0 0 0 |
| − | 6 5 1 0 0 0 0 | + | 6 5 1 0 0 0 0 |
| − | 22 9 1 0 0 0 0 | + | 22 9 1 0 0 0 0 |
| − | 23 11 1 0 0 0 0 | + | 23 11 1 0 0 0 0 |
| − | 11 10 1 0 0 0 0 | + | 11 10 1 0 0 0 0 |
| − | 10 9 1 0 0 0 0 | + | 10 9 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 20 19 1 0 0 0 0 | + | 20 19 1 0 0 0 0 |
| − | 19 24 1 0 0 0 0 | + | 19 24 1 0 0 0 0 |
| − | 24 16 1 0 0 0 0 | + | 24 16 1 0 0 0 0 |
| − | 16 15 1 0 0 0 0 | + | 16 15 1 0 0 0 0 |
| − | 15 14 1 0 0 0 0 | + | 15 14 1 0 0 0 0 |
| − | 14 23 1 0 0 0 0 | + | 14 23 1 0 0 0 0 |
| − | 6 7 2 0 0 0 0 | + | 6 7 2 0 0 0 0 |
| − | 9 8 2 0 0 0 0 | + | 9 8 2 0 0 0 0 |
| − | 8 7 1 0 0 0 0 | + | 8 7 1 0 0 0 0 |
| − | 4 3 2 0 0 0 0 | + | 4 3 2 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 19 18 2 0 0 0 0 | + | 19 18 2 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 18 17 1 0 0 0 0 | + | 18 17 1 0 0 0 0 |
| − | 14 13 2 0 0 0 0 | + | 14 13 2 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 7 28 1 0 0 0 0 | + | 7 28 1 0 0 0 0 |
| − | 8 29 1 0 0 0 0 | + | 8 29 1 0 0 0 0 |
| − | 3 26 1 0 0 0 0 | + | 3 26 1 0 0 0 0 |
| − | 2 25 1 0 0 0 0 | + | 2 25 1 0 0 0 0 |
| − | 18 38 1 0 0 0 0 | + | 18 38 1 0 0 0 0 |
| − | 17 35 1 0 0 0 0 | + | 17 35 1 0 0 0 0 |
| − | 13 32 1 0 0 0 0 | + | 13 32 1 0 0 0 0 |
| − | 12 31 1 0 0 0 0 | + | 12 31 1 0 0 0 0 |
| − | 21 39 1 0 0 0 0 | + | 21 39 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 23 39 1 0 0 0 0 | + | 23 39 1 0 0 0 0 |
| − | 24 39 1 0 0 0 0 | + | 24 39 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 34 41 2 0 0 0 0 | + | 34 41 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 37 42 1 0 0 0 0 | + | 37 42 1 0 0 0 0 |
| − | 37 43 2 0 0 0 0 | + | 37 43 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMCCPPPR0003 | + | ID BMCCPPPR0003 |
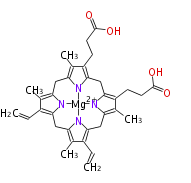
| − | NAME Magnesium protoporphyrin IX | + | NAME Magnesium protoporphyrin IX |
| − | FORMULA C34H36MgN4O4 | + | FORMULA C34H36MgN4O4 |
| − | EXACTMASS 588.2586 | + | EXACTMASS 588.2586 |
| − | AVERAGEMASS 588.9792 | + | AVERAGEMASS 588.9792 |
| − | SMILES n([Mg+2]623)(c15)c(Cc(c8C)n2c(c(CCC(O)=O)8)Cc(c7CCC(O)=O)n3c(c7C)Cc(n64)c(C=C)c(c4C5)C)c(c(C=C)1)C | + | SMILES n([Mg+2]623)(c15)c(Cc(c8C)n2c(c(CCC(O)=O)8)Cc(c7CCC(O)=O)n3c(c7C)Cc(n64)c(C=C)c(c4C5)C)c(c(C=C)1)C |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C03516 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C03516 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 50 0 0 0 0 0 0 0 0999 V2000
7.1238 2.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3444 3.1173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0580 3.1173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.2785 2.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.0672 2.2273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.2785 1.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.9573 1.2181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.9573 0.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.2785 0.2839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.0672 -0.5047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.2785 -0.7161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0580 -1.3948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3444 -1.3948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1238 -0.7161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3352 -0.5047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1238 0.2839 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4451 0.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4451 1.2181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1238 1.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3352 2.2273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.7012 1.8613 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
8.7012 0.8613 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
7.7012 -0.1387 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.7012 0.8613 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
6.7566 3.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.6458 3.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.2391 4.8399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.7663 1.8059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.7663 -0.0833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.6798 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.6458 -2.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.7566 -2.2038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.1633 -3.1173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5755 -3.9264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6361 -0.0833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7226 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9135 -0.2644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6361 1.8059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.7012 0.8613 0.0000 Mg 0 2 0 0 0 0 0 0 0 0 0 0
5.5810 -3.8218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.9823 -4.8399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0181 -1.2589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 0.1424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4 5 1 0 0 0 0
4 21 1 0 0 0 0
22 6 1 0 0 0 0
6 5 1 0 0 0 0
22 9 1 0 0 0 0
23 11 1 0 0 0 0
11 10 1 0 0 0 0
10 9 1 0 0 0 0
1 20 1 0 0 0 0
20 19 1 0 0 0 0
19 24 1 0 0 0 0
24 16 1 0 0 0 0
16 15 1 0 0 0 0
15 14 1 0 0 0 0
14 23 1 0 0 0 0
6 7 2 0 0 0 0
9 8 2 0 0 0 0
8 7 1 0 0 0 0
4 3 2 0 0 0 0
1 21 1 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
19 18 2 0 0 0 0
16 17 2 0 0 0 0
18 17 1 0 0 0 0
14 13 2 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
7 28 1 0 0 0 0
8 29 1 0 0 0 0
3 26 1 0 0 0 0
2 25 1 0 0 0 0
18 38 1 0 0 0 0
17 35 1 0 0 0 0
13 32 1 0 0 0 0
12 31 1 0 0 0 0
21 39 1 0 0 0 0
22 39 1 0 0 0 0
23 39 1 0 0 0 0
24 39 1 0 0 0 0
26 27 2 0 0 0 0
29 30 2 0 0 0 0
35 36 1 0 0 0 0
32 33 1 0 0 0 0
34 40 1 0 0 0 0
34 41 2 0 0 0 0
33 34 1 0 0 0 0
37 42 1 0 0 0 0
37 43 2 0 0 0 0
36 37 1 0 0 0 0
S SKP 7
ID BMCCPPPR0003
NAME Magnesium protoporphyrin IX
FORMULA C34H36MgN4O4
EXACTMASS 588.2586
AVERAGEMASS 588.9792
SMILES n([Mg+2]623)(c15)c(Cc(c8C)n2c(c(CCC(O)=O)8)Cc(c7CCC(O)=O)n3c(c7C)Cc(n64)c(C=C)c(c4C5)C)c(c(C=C)1)C
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C03516
M END