Mol:PR101053
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | ACD/Labs04270909262D | + | ACD/Labs04270909262D |
| − | + | ||
| − | 43 47 0 0 1 0 0 0 0 0 1 V2000 | + | 43 47 0 0 1 0 0 0 0 0 1 V2000 |
| − | 5.9472 -10.8829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9472 -10.8829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1146 -11.3636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1146 -11.3636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1146 -12.3250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1146 -12.3250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9472 -12.8058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9472 -12.8058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7799 -12.3250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7799 -12.3250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7799 -11.3636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7799 -11.3636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2820 -10.8829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2820 -10.8829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9472 -13.7672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9472 -13.7672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2820 -12.8058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2820 -12.8058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8850 -11.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8850 -11.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8850 -12.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8850 -12.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7176 -12.7924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7176 -12.7924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5503 -12.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5503 -12.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5503 -11.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5503 -11.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7176 -10.8695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7176 -10.8695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.3829 -12.7924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.3829 -12.7924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.2155 -12.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.2155 -12.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.2155 -11.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.2155 -11.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.3829 -10.8695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.3829 -10.8695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.0482 -10.8695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.0482 -10.8695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.8808 -11.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.8808 -11.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.7134 -10.8695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.7134 -10.8695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.7134 -9.9081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.7134 -9.9081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.8808 -9.4273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.8808 -9.4273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.0482 -9.9081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.0482 -9.9081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.5461 -9.4273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.5461 -9.4273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.3044 -13.0276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.3044 -13.0276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.3829 -13.7538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.3829 -13.7538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.7176 -13.7538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.7176 -13.7538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8167 -10.8495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8167 -10.8495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9472 -9.9214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9472 -9.9214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.8808 -8.4659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.8808 -8.4659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.4040 -14.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.4040 -14.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.4040 -15.9567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.4040 -15.9567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2366 -16.4374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.2366 -16.4374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.0692 -15.9567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.0692 -15.9567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.0692 -14.9952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.0692 -14.9952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2366 -14.5145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.2366 -14.5145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5713 -16.4374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5713 -16.4374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.9979 -16.2055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.9979 -16.2055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5713 -14.5145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5713 -14.5145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2366 -17.3989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.2366 -17.3989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.6778 -15.5257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.6778 -15.5257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 1 0 0 0 | + | 2 7 1 1 0 0 0 |
| − | 4 8 1 1 0 0 0 | + | 4 8 1 1 0 0 0 |
| − | 3 9 1 6 0 0 0 | + | 3 9 1 6 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 13 16 1 0 0 0 0 | + | 13 16 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 18 20 1 0 0 0 0 | + | 18 20 1 0 0 0 0 |
| − | 20 21 2 0 0 0 0 | + | 20 21 2 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 23 26 1 0 0 0 0 | + | 23 26 1 0 0 0 0 |
| − | 17 27 1 0 0 0 0 | + | 17 27 1 0 0 0 0 |
| − | 16 28 2 0 0 0 0 | + | 16 28 2 0 0 0 0 |
| − | 12 29 1 0 0 0 0 | + | 12 29 1 0 0 0 0 |
| − | 10 30 1 0 0 0 0 | + | 10 30 1 0 0 0 0 |
| − | 30 6 1 6 0 0 0 | + | 30 6 1 6 0 0 0 |
| − | 1 31 1 1 0 0 0 | + | 1 31 1 1 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 34 39 1 6 0 0 0 | + | 34 39 1 6 0 0 0 |
| − | 36 40 1 6 0 0 0 | + | 36 40 1 6 0 0 0 |
| − | 33 41 1 1 0 0 0 | + | 33 41 1 1 0 0 0 |
| − | 35 42 1 1 0 0 0 | + | 35 42 1 1 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 27 38 1 6 0 0 0 | + | 27 38 1 6 0 0 0 |
| − | S SKP | + | S SKP 7 |
| − | CAS_RN 18016-58-5 | + | CAS_RN 18016-58-5 |
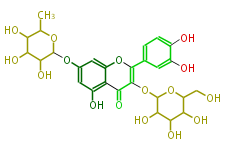
| − | NAME Quercetin-3-O-beta-glucopyranosyl-7-O-alpha-rhamnopyranoside | + | NAME Quercetin-3-O-beta-glucopyranosyl-7-O-alpha-rhamnopyranoside |
| + | ID PR101053 | ||
| + | FORMULA C27H30O16 | ||
| + | EXACTMASS 610.153384912 | ||
| + | AVERAGEMASS 610.5175 | ||
| + | SMILES C(C1O)(C)OC(Oc(c5)cc(O2)c(c5O)C(C(OC(O4)C(C(C(C4CO)O)O)O)=C(c(c3)ccc(O)c3O)2)=O)C(C1O)O | ||
M END | M END | ||
Latest revision as of 10:37, 23 December 2009
ACD/Labs04270909262D
43 47 0 0 1 0 0 0 0 0 1 V2000
5.9472 -10.8829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1146 -11.3636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1146 -12.3250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9472 -12.8058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.7799 -12.3250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.7799 -11.3636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2820 -10.8829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9472 -13.7672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2820 -12.8058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.8850 -11.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8850 -12.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.7176 -12.7924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.5503 -12.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.5503 -11.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.7176 -10.8695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.3829 -12.7924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.2155 -12.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.2155 -11.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.3829 -10.8695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.0482 -10.8695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.8808 -11.3502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.7134 -10.8695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.7134 -9.9081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.8808 -9.4273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.0482 -9.9081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
15.5461 -9.4273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.3044 -13.0276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.3829 -13.7538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.7176 -13.7538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.8167 -10.8495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9472 -9.9214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.8808 -8.4659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.4040 -14.9952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.4040 -15.9567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.2366 -16.4374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
15.0692 -15.9567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
15.0692 -14.9952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.2366 -14.5145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.5713 -16.4374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
15.9979 -16.2055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.5713 -14.5145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.2366 -17.3989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
16.6778 -15.5257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 1 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 1 0 0 0 0
2 7 1 1 0 0 0
4 8 1 1 0 0 0
3 9 1 6 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
13 16 1 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 19 1 0 0 0 0
19 14 1 0 0 0 0
18 20 1 0 0 0 0
20 21 2 0 0 0 0
21 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 20 1 0 0 0 0
23 26 1 0 0 0 0
17 27 1 0 0 0 0
16 28 2 0 0 0 0
12 29 1 0 0 0 0
10 30 1 0 0 0 0
30 6 1 6 0 0 0
1 31 1 1 0 0 0
24 32 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
34 39 1 6 0 0 0
36 40 1 6 0 0 0
33 41 1 1 0 0 0
35 42 1 1 0 0 0
40 43 1 0 0 0 0
27 38 1 6 0 0 0
S SKP 7
CAS_RN 18016-58-5
NAME Quercetin-3-O-beta-glucopyranosyl-7-O-alpha-rhamnopyranoside
ID PR101053
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES C(C1O)(C)OC(Oc(c5)cc(O2)c(c5O)C(C(OC(O4)C(C(C(C4CO)O)O)O)=C(c(c3)ccc(O)c3O)2)=O)C(C1O)O
M END