Mol:PR100461
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | ACD/Labs08070915142D | + | ACD/Labs08070915142D |
| − | + | ||
| − | 43 47 0 0 1 0 0 0 0 0 2 V2000 | + | 43 47 0 0 1 0 0 0 0 0 2 V2000 |
| − | 13.5283 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.5283 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.5283 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.5283 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.3609 -7.7477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.3609 -7.7477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.1936 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.1936 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.1936 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.1936 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.3609 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.3609 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.0262 -7.7477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.0262 -7.7477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.8589 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.8589 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.8589 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.8589 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.0262 -5.8248 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 16.0262 -5.8248 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 21.7572 -8.9405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.7572 -8.9405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.5899 -8.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.5899 -8.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.5899 -7.4983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.5899 -7.4983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.7572 -7.0176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.7572 -7.0176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.9246 -7.4983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.9246 -7.4983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.9246 -8.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.9246 -8.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.7863 -8.4522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.7863 -8.4522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.7572 -6.0562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.7572 -6.0562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.3609 -8.7091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.3609 -8.7091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.6957 -5.8248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.6957 -5.8248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.6915 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.6915 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.5241 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.5241 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.3568 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.3568 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.3568 -4.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.3568 -4.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.5241 -4.3826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.5241 -4.3826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.6915 -4.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.6915 -4.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.5241 -3.4212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.5241 -3.4212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.1894 -4.3826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.1894 -4.3826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.4225 -7.0176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.4225 -7.0176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.7878 -10.6867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.7878 -10.6867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.5185 -8.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.5185 -8.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.1894 -6.3055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.1894 -6.3055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.6859 -5.8073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.6859 -5.8073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.0556 -10.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.0556 -10.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.0556 -11.6815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.0556 -11.6815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.8883 -12.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.8883 -12.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.7209 -11.6815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.7209 -11.6815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.7209 -10.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.7209 -10.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.8883 -10.2393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.8883 -10.2393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.0556 -9.7586 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.0556 -9.7586 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.8883 -9.2779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.8883 -9.2779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.5536 -12.1622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.5536 -12.1622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.5536 -10.2393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.5536 -10.2393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 18 1 6 0 0 0 | + | 14 18 1 6 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 9 21 1 0 0 0 0 | + | 9 21 1 0 0 0 0 |
| − | 21 22 2 0 0 0 0 | + | 21 22 2 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 17 8 1 0 0 0 0 | + | 17 8 1 0 0 0 0 |
| − | 13 29 1 1 0 0 0 | + | 13 29 1 1 0 0 0 |
| − | 11 30 1 1 0 0 0 | + | 11 30 1 1 0 0 0 |
| − | 12 31 1 6 0 0 0 | + | 12 31 1 6 0 0 0 |
| − | 23 32 1 0 0 0 0 | + | 23 32 1 0 0 0 0 |
| − | 18 33 1 0 0 0 0 | + | 18 33 1 0 0 0 0 |
| − | 17 16 1 6 0 0 0 | + | 17 16 1 6 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 34 40 1 6 0 0 0 | + | 34 40 1 6 0 0 0 |
| − | 39 41 1 6 0 0 0 | + | 39 41 1 6 0 0 0 |
| − | 37 42 1 6 0 0 0 | + | 37 42 1 6 0 0 0 |
| − | 38 43 1 1 0 0 0 | + | 38 43 1 1 0 0 0 |
| − | 34 30 1 0 0 0 0 | + | 34 30 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | S SKP | + | S SKP 7 |
| − | CAS_RN 178275-92-8 | + | CAS_RN 178275-92-8 |
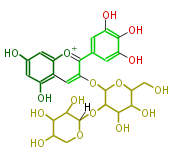
| − | NAME Delphinidin-3-O-(2''-O-beta-xylopyranosyl-beta-glucopyranoside) | + | NAME Delphinidin-3-O-(2''-O-beta-xylopyranosyl-beta-glucopyranoside) |
| + | ID PR100461 | ||
| + | FORMULA C26H29O16 | ||
| + | EXACTMASS 597.1455598800001 | ||
| + | AVERAGEMASS 597.4988599999999 | ||
| + | SMILES C(C5O)(O)COC(C5O)(OC(C4O)C(OC(C4O)CO)Oc(c2)c([o+1]c(c3)c(c(O)cc3O)2)c(c1)cc(c(c1O)O)O)[H] | ||
M END | M END | ||
Latest revision as of 16:44, 14 January 2011
ACD/Labs08070915142D 43 47 0 0 1 0 0 0 0 0 2 V2000 13.5283 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 13.5283 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 14.3609 -7.7477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 15.1936 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 15.1936 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 14.3609 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.0262 -7.7477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.8589 -7.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.8589 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 16.0262 -5.8248 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 21.7572 -8.9405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.5899 -8.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 22.5899 -7.4983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 21.7572 -7.0176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 20.9246 -7.4983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 20.9246 -8.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.7863 -8.4522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.7572 -6.0562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 14.3609 -8.7091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 12.6957 -5.8248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 17.6915 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.5241 -6.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.3568 -5.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 19.3568 -4.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.5241 -4.3826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 17.6915 -4.8633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 18.5241 -3.4212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 20.1894 -4.3826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.4225 -7.0176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 21.7878 -10.6867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 23.5185 -8.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 20.1894 -6.3055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 22.6859 -5.8073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 24.0556 -10.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 24.0556 -11.6815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 24.8883 -12.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 25.7209 -11.6815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 25.7209 -10.7200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 24.8883 -10.2393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 24.0556 -9.7586 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 24.8883 -9.2779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 26.5536 -12.1622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 26.5536 -10.2393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 1 2 2 0 0 0 0 2 3 1 0 0 0 0 3 4 2 0 0 0 0 4 5 1 0 0 0 0 5 6 2 0 0 0 0 6 1 1 0 0 0 0 4 7 1 0 0 0 0 7 8 2 0 0 0 0 8 9 1 0 0 0 0 9 10 2 0 0 0 0 10 5 1 0 0 0 0 11 12 1 0 0 0 0 12 13 1 0 0 0 0 13 14 1 0 0 0 0 14 15 1 0 0 0 0 15 16 1 0 0 0 0 16 11 1 0 0 0 0 14 18 1 6 0 0 0 3 19 1 0 0 0 0 1 20 1 0 0 0 0 9 21 1 0 0 0 0 21 22 2 0 0 0 0 22 23 1 0 0 0 0 23 24 2 0 0 0 0 24 25 1 0 0 0 0 25 26 2 0 0 0 0 26 21 1 0 0 0 0 25 27 1 0 0 0 0 24 28 1 0 0 0 0 17 8 1 0 0 0 0 13 29 1 1 0 0 0 11 30 1 1 0 0 0 12 31 1 6 0 0 0 23 32 1 0 0 0 0 18 33 1 0 0 0 0 17 16 1 6 0 0 0 34 35 1 0 0 0 0 35 36 1 0 0 0 0 36 37 1 0 0 0 0 37 38 1 0 0 0 0 38 39 1 0 0 0 0 39 34 1 0 0 0 0 34 40 1 6 0 0 0 39 41 1 6 0 0 0 37 42 1 6 0 0 0 38 43 1 1 0 0 0 34 30 1 0 0 0 0 M CHG 1 10 1 S SKP 7 CAS_RN 178275-92-8 NAME Delphinidin-3-O-(2''-O-beta-xylopyranosyl-beta-glucopyranoside) ID PR100461 FORMULA C26H29O16 EXACTMASS 597.1455598800001 AVERAGEMASS 597.4988599999999 SMILES C(C5O)(O)COC(C5O)(OC(C4O)C(OC(C4O)CO)Oc(c2)c([o+1]c(c3)c(c(O)cc3O)2)c(c1)cc(c(c1O)O)O)[H] M END