Mol:FLIJ1ANR0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 35 38 0 0 0 0 0 0 0 0999 V2000 | + | 35 38 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.8465 -0.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8465 -0.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8465 -0.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8465 -0.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2902 -1.1857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2902 -1.1857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2661 -0.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2661 -0.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2661 -0.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2661 -0.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2902 0.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2902 0.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8224 -1.1857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8224 -1.1857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3787 -0.8645 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.3787 -0.8645 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.9348 -1.1856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9348 -1.1856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9348 -1.8724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9348 -1.8724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5296 -2.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5296 -2.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1244 -1.8724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1244 -1.8724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1244 -1.1856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1244 -1.1856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5296 -0.8422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5296 -0.8422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4026 0.0989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4026 0.0989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3787 -0.4078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3787 -0.4078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8224 -1.7072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8224 -1.7072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0634 0.4584 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.0634 0.4584 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.5601 0.7490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5601 0.7490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5601 1.3302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5601 1.3302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0634 1.6208 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.0634 1.6208 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.5667 1.3302 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.5667 1.3302 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.5667 0.7490 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.5667 0.7490 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.6307 0.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6307 0.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0700 1.6208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0700 1.6208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5733 1.3302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5733 1.3302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5733 0.7490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5733 0.7490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0700 0.4584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0700 0.4584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0758 1.6203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0758 1.6203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0634 2.2010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0634 2.2010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5659 2.4912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5659 2.4912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5609 2.4912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5609 2.4912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2902 -1.8279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2902 -1.8279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9904 -2.3724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9904 -2.3724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8565 -2.8724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8565 -2.8724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 9 2 0 0 0 0 | + | 14 9 2 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 1 15 1 0 0 0 0 | + | 1 15 1 0 0 0 0 |
| − | 8 16 1 0 0 0 0 | + | 8 16 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 18 15 1 6 0 0 0 | + | 18 15 1 6 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 18 1 0 0 0 0 | + | 23 18 1 0 0 0 0 |
| − | 18 24 1 1 0 0 0 | + | 18 24 1 1 0 0 0 |
| − | 22 25 1 0 0 0 0 | + | 22 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 21 30 1 0 0 0 0 | + | 21 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 3 33 1 0 0 0 0 | + | 3 33 1 0 0 0 0 |
| − | 12 34 1 0 0 0 0 | + | 12 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 34 35 | + | M SAL 1 2 34 35 |
| − | M SBL 1 1 37 | + | M SBL 1 1 37 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 37 3.3613 -2.0787 | + | M SVB 1 37 3.3613 -2.0787 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIJ1ANR0002 | + | ID FLIJ1ANR0002 |
| − | KNApSAcK_ID C00009798 | + | KNApSAcK_ID C00009798 |
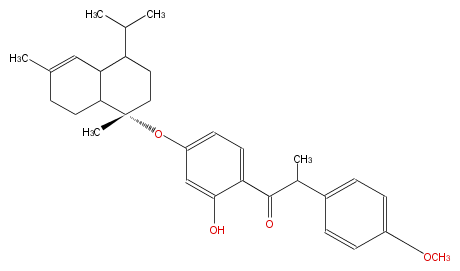
| − | NAME 4-O-T-Cadinylangolensin | + | NAME 4-O-T-Cadinylangolensin |
| − | CAS_RN 75872-84-3 | + | CAS_RN 75872-84-3 |
| − | FORMULA C31H40O4 | + | FORMULA C31H40O4 |
| − | EXACTMASS 476.29265976799996 | + | EXACTMASS 476.29265976799996 |
| − | AVERAGEMASS 476.6469 | + | AVERAGEMASS 476.6469 |
| − | SMILES [C@@](C)(C2)(Oc(c3)ccc(C(C(c(c4)ccc(OC)c4)C)=O)c(O)3)C(C(C(C2)C(C)C)1)CCC(C)=C1 | + | SMILES [C@@](C)(C2)(Oc(c3)ccc(C(C(c(c4)ccc(OC)c4)C)=O)c(O)3)C(C(C(C2)C(C)C)1)CCC(C)=C1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
35 38 0 0 0 0 0 0 0 0999 V2000
-0.8465 -0.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8465 -0.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2902 -1.1857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2661 -0.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2661 -0.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2902 0.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8224 -1.1857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3787 -0.8645 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.9348 -1.1856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9348 -1.8724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5296 -2.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1244 -1.8724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1244 -1.1856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5296 -0.8422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4026 0.0989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3787 -0.4078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8224 -1.7072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0634 0.4584 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.5601 0.7490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5601 1.3302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0634 1.6208 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.5667 1.3302 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.5667 0.7490 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.6307 0.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0700 1.6208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5733 1.3302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5733 0.7490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0700 0.4584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0758 1.6203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0634 2.2010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5659 2.4912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5609 2.4912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2902 -1.8279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9904 -2.3724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8565 -2.8724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 9 2 0 0 0 0
1 2 1 0 0 0 0
1 15 1 0 0 0 0
8 16 1 0 0 0 0
7 17 2 0 0 0 0
18 15 1 6 0 0 0
18 19 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 18 1 0 0 0 0
18 24 1 1 0 0 0
22 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
26 29 1 0 0 0 0
21 30 1 0 0 0 0
30 31 1 0 0 0 0
30 32 1 0 0 0 0
3 33 1 0 0 0 0
12 34 1 0 0 0 0
34 35 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 34 35
M SBL 1 1 37
M SMT 1 OCH3
M SVB 1 37 3.3613 -2.0787
S SKP 8
ID FLIJ1ANR0002
KNApSAcK_ID C00009798
NAME 4-O-T-Cadinylangolensin
CAS_RN 75872-84-3
FORMULA C31H40O4
EXACTMASS 476.29265976799996
AVERAGEMASS 476.6469
SMILES [C@@](C)(C2)(Oc(c3)ccc(C(C(c(c4)ccc(OC)c4)C)=O)c(O)3)C(C(C(C2)C(C)C)1)CCC(C)=C1
M END