Mol:FLIF1LGF0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 46 0 0 0 0 0 0 0 0999 V2000 | + | 41 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4390 2.0083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4390 2.0083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8827 1.6871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8827 1.6871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5841 1.6395 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.5841 1.6395 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.2379 1.1825 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.2379 1.1825 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.7393 1.3764 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.7393 1.3764 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.2196 1.3706 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.2196 1.3706 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.6078 1.7312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6078 1.7312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1171 1.5484 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.1171 1.5484 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.4245 1.4143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4245 1.4143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7777 1.1562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7777 1.1562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4536 0.8968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4536 0.8968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4289 0.0165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4289 0.0165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1274 0.3377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1274 0.3377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6835 0.0166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6835 0.0166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6835 -0.6494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6835 -0.6494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2602 -0.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2602 -0.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8369 -0.6494 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.8369 -0.6494 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.8369 0.0166 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.8369 0.0166 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.2602 0.3496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2602 0.3496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4287 -0.6255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4287 -0.6255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1274 -0.9466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1274 -0.9466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2602 -1.6478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2602 -1.6478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4133 -0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4133 -0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4133 -1.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4133 -1.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9646 -1.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9646 -1.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5160 -1.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5160 -1.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5160 -0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5160 -0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9646 -0.6638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9646 -0.6638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1395 2.1162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1395 2.1162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8827 2.5087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8827 2.5087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0062 0.9660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0062 0.9660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6450 1.0331 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.6450 1.0331 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.9063 0.4463 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9063 0.4463 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9904 0.0166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9904 0.0166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4137 0.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4137 0.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5258 -2.0962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5258 -2.0962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2403 -2.5087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2403 -2.5087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5160 -1.6188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5160 -1.6188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5160 -1.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5160 -1.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6908 2.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6908 2.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1033 3.0066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1033 3.0066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 3 4 1 1 0 0 0 | + | 3 4 1 1 0 0 0 |
| − | 4 5 1 1 0 0 0 | + | 4 5 1 1 0 0 0 |
| − | 6 5 1 1 0 0 0 | + | 6 5 1 1 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 3 1 0 0 0 0 | + | 8 3 1 0 0 0 0 |
| − | 3 9 1 0 0 0 0 | + | 3 9 1 0 0 0 0 |
| − | 4 10 1 0 0 0 0 | + | 4 10 1 0 0 0 0 |
| − | 5 11 1 0 0 0 0 | + | 5 11 1 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 12 20 1 0 0 0 0 | + | 12 20 1 0 0 0 0 |
| − | 20 21 2 0 0 0 0 | + | 20 21 2 0 0 0 0 |
| − | 21 15 1 0 0 0 0 | + | 21 15 1 0 0 0 0 |
| − | 16 22 2 0 0 0 0 | + | 16 22 2 0 0 0 0 |
| − | 17 23 1 0 0 0 0 | + | 17 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 2 29 1 0 0 0 0 | + | 2 29 1 0 0 0 0 |
| − | 2 30 1 0 0 0 0 | + | 2 30 1 0 0 0 0 |
| − | 13 31 1 0 0 0 0 | + | 13 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 12 1 0 0 0 0 | + | 33 12 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 18 1 0 0 0 0 | + | 35 18 1 0 0 0 0 |
| − | 32 2 1 0 0 0 0 | + | 32 2 1 0 0 0 0 |
| − | 25 36 1 0 0 0 0 | + | 25 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 26 38 1 0 0 0 0 | + | 26 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 8 40 1 0 0 0 0 | + | 8 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 40 41 | + | M SAL 3 2 40 41 |
| − | M SBL 3 1 45 | + | M SBL 3 1 45 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 45 -3.3227 2.4649 | + | M SVB 3 45 -3.3227 2.4649 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 38 39 | + | M SAL 2 2 38 39 |
| − | M SBL 2 1 43 | + | M SBL 2 1 43 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 43 3.7101 -1.825 | + | M SVB 2 43 3.7101 -1.825 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 36 37 | + | M SAL 1 2 36 37 |
| − | M SBL 1 1 41 | + | M SBL 1 1 41 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 41 2.5258 -2.0962 | + | M SVB 1 41 2.5258 -2.0962 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIF1LGF0003 | + | ID FLIF1LGF0003 |
| − | KNApSAcK_ID C00010178 | + | KNApSAcK_ID C00010178 |
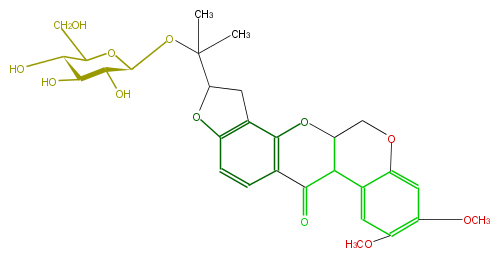
| − | NAME Dalpanol O-glucoside | + | NAME Dalpanol O-glucoside |
| − | CAS_RN 52059-86-6 | + | CAS_RN 52059-86-6 |
| − | FORMULA C29H34O12 | + | FORMULA C29H34O12 |
| − | EXACTMASS 574.2050265519999 | + | EXACTMASS 574.2050265519999 |
| − | AVERAGEMASS 574.5730599999999 | + | AVERAGEMASS 574.5730599999999 |
| − | SMILES O[C@H]([C@@H]1O)C(O[C@@H](OC(C(O6)Cc(c56)c(c4cc5)OC(C2)C(C(=O)4)c(c3)c(cc(OC)c3OC)O2)(C)C)[C@H]1O)CO | + | SMILES O[C@H]([C@@H]1O)C(O[C@@H](OC(C(O6)Cc(c56)c(c4cc5)OC(C2)C(C(=O)4)c(c3)c(cc(OC)c3OC)O2)(C)C)[C@H]1O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 46 0 0 0 0 0 0 0 0999 V2000
-1.4390 2.0083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8827 1.6871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5841 1.6395 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.2379 1.1825 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.7393 1.3764 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.2196 1.3706 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.6078 1.7312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1171 1.5484 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.4245 1.4143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7777 1.1562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4536 0.8968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4289 0.0165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1274 0.3377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6835 0.0166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6835 -0.6494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2602 -0.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8369 -0.6494 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.8369 0.0166 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.2602 0.3496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4287 -0.6255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1274 -0.9466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2602 -1.6478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4133 -0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4133 -1.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9646 -1.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5160 -1.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5160 -0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9646 -0.6638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1395 2.1162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8827 2.5087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0062 0.9660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6450 1.0331 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.9063 0.4463 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9904 0.0166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4137 0.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5258 -2.0962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2403 -2.5087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5160 -1.6188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5160 -1.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6908 2.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1033 3.0066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
3 4 1 1 0 0 0
4 5 1 1 0 0 0
6 5 1 1 0 0 0
6 7 1 0 0 0 0
7 8 1 0 0 0 0
8 3 1 0 0 0 0
3 9 1 0 0 0 0
4 10 1 0 0 0 0
5 11 1 0 0 0 0
6 1 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 19 1 0 0 0 0
19 14 1 0 0 0 0
12 20 1 0 0 0 0
20 21 2 0 0 0 0
21 15 1 0 0 0 0
16 22 2 0 0 0 0
17 23 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
2 29 1 0 0 0 0
2 30 1 0 0 0 0
13 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 12 1 0 0 0 0
28 34 1 0 0 0 0
34 35 1 0 0 0 0
35 18 1 0 0 0 0
32 2 1 0 0 0 0
25 36 1 0 0 0 0
36 37 1 0 0 0 0
26 38 1 0 0 0 0
38 39 1 0 0 0 0
8 40 1 0 0 0 0
40 41 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 40 41
M SBL 3 1 45
M SMT 3 CH2OH
M SVB 3 45 -3.3227 2.4649
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 38 39
M SBL 2 1 43
M SMT 2 OCH3
M SVB 2 43 3.7101 -1.825
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 36 37
M SBL 1 1 41
M SMT 1 OCH3
M SVB 1 41 2.5258 -2.0962
S SKP 8
ID FLIF1LGF0003
KNApSAcK_ID C00010178
NAME Dalpanol O-glucoside
CAS_RN 52059-86-6
FORMULA C29H34O12
EXACTMASS 574.2050265519999
AVERAGEMASS 574.5730599999999
SMILES O[C@H]([C@@H]1O)C(O[C@@H](OC(C(O6)Cc(c56)c(c4cc5)OC(C2)C(C(=O)4)c(c3)c(cc(OC)c3OC)O2)(C)C)[C@H]1O)CO
M END