Mol:FLIAFCGS0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 47 52 0 0 0 0 0 0 0 0999 V2000 | + | 47 52 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.4534 0.8464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4534 0.8464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1029 0.5252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1029 0.5252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6592 0.8464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6592 0.8464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2153 0.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2153 0.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2153 -0.1406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2153 -0.1406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7921 -0.4736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7921 -0.4736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3688 -0.1406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3688 -0.1406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3688 0.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3688 0.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7921 0.8583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7921 0.8583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1031 -0.1168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1031 -0.1168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6592 -0.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6592 -0.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7921 -1.1391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7921 -1.1391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9451 -0.4734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9451 -0.4734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9451 -1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9451 -1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4965 -1.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4965 -1.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0478 -1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0478 -1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0478 -0.4734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0478 -0.4734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4965 -0.1550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4965 -0.1550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5573 0.6426 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.5573 0.6426 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.2110 0.1856 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.2110 0.1856 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.7124 0.3795 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.7124 0.3795 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.1928 0.3738 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.1928 0.3738 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.5809 0.7344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5809 0.7344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0903 0.5515 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.0903 0.5515 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.9443 0.8661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9443 0.8661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5378 -0.2447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5378 -0.2447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4268 -0.1001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4268 -0.1001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6533 -1.3067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6533 -1.3067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0275 -0.7917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0275 -0.7917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6533 -0.2766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6533 -0.2766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4117 -1.9781 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.4117 -1.9781 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.8391 -2.0067 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.8391 -2.0067 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.6671 -1.5001 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.6671 -1.5001 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.3286 -1.1057 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.3286 -1.1057 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.8544 -1.1713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8544 -1.1713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0417 -1.6789 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.0417 -1.6789 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.8316 -2.1310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8316 -2.1310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7195 -2.5334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7195 -2.5334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1161 -1.5895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1161 -1.5895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9373 1.4194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9373 1.4194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3739 2.3190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3739 2.3190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2897 1.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2897 1.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5561 2.1079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5561 2.1079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2195 -0.6025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2195 -0.6025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9340 -1.0150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9340 -1.0150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8416 -1.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8416 -1.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6820 -1.8101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6820 -1.8101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 2 10 1 0 0 0 0 | + | 2 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 5 1 0 0 0 0 | + | 11 5 1 0 0 0 0 |
| − | 6 12 2 0 0 0 0 | + | 6 12 2 0 0 0 0 |
| − | 7 13 1 0 0 0 0 | + | 7 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 1 1 0 0 0 0 | + | 22 1 1 0 0 0 0 |
| − | 16 28 1 0 0 0 0 | + | 16 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 17 1 0 0 0 0 | + | 30 17 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 26 1 0 0 0 0 | + | 34 26 1 0 0 0 0 |
| − | 3 40 1 0 0 0 0 | + | 3 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 24 42 1 0 0 0 0 | + | 24 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 11 44 1 0 0 0 0 | + | 11 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 36 46 1 0 0 0 0 | + | 36 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 46 47 | + | M SAL 4 2 46 47 |
| − | M SBL 4 1 51 | + | M SBL 4 1 51 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 51 -4.3728 0.5002 | + | M SVB 4 51 -4.3728 0.5002 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 42 43 | + | M SAL 3 2 42 43 |
| − | M SBL 3 1 47 | + | M SBL 3 1 47 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 47 -2.2897 1.4283 | + | M SVB 3 47 -2.2897 1.4283 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 44 45 | + | M SAL 2 2 44 45 |
| − | M SBL 2 1 49 | + | M SBL 2 1 49 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 49 0.2195 -0.6025 | + | M SVB 2 49 0.2195 -0.6025 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 45 0.9373 1.4194 | + | M SVB 1 45 0.9373 1.4194 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIAFCGS0003 | + | ID FLIAFCGS0003 |
| − | KNApSAcK_ID C00010136 | + | KNApSAcK_ID C00010136 |
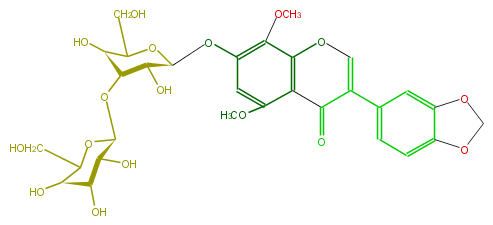
| − | NAME Platycarpanetin 7-O-laminaribioside | + | NAME Platycarpanetin 7-O-laminaribioside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C30H34O17 | + | FORMULA C30H34O17 |
| − | EXACTMASS 666.179599662 | + | EXACTMASS 666.179599662 |
| − | AVERAGEMASS 666.58076 | + | AVERAGEMASS 666.58076 |
| − | SMILES C(O1)Oc(c2)c1cc(C(C6=O)=COc(c65)c(c(cc5OC)O[C@@H]([C@H]3O)OC(CO)[C@@H]([C@@H]3O[C@@H]([C@@H](O)4)OC([C@H](O)[C@@H]4O)CO)O)OC)c2 | + | SMILES C(O1)Oc(c2)c1cc(C(C6=O)=COc(c65)c(c(cc5OC)O[C@@H]([C@H]3O)OC(CO)[C@@H]([C@@H]3O[C@@H]([C@@H](O)4)OC([C@H](O)[C@@H]4O)CO)O)OC)c2 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
47 52 0 0 0 0 0 0 0 0999 V2000
-0.4534 0.8464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1029 0.5252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6592 0.8464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2153 0.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2153 -0.1406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7921 -0.4736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3688 -0.1406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3688 0.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7921 0.8583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1031 -0.1168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6592 -0.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7921 -1.1391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9451 -0.4734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9451 -1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4965 -1.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0478 -1.1100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0478 -0.4734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4965 -0.1550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5573 0.6426 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.2110 0.1856 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.7124 0.3795 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.1928 0.3738 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.5809 0.7344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0903 0.5515 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.9443 0.8661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5378 -0.2447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4268 -0.1001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6533 -1.3067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0275 -0.7917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6533 -0.2766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4117 -1.9781 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.8391 -2.0067 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.6671 -1.5001 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.3286 -1.1057 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.8544 -1.1713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0417 -1.6789 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.8316 -2.1310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7195 -2.5334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1161 -1.5895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9373 1.4194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3739 2.3190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2897 1.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5561 2.1079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2195 -0.6025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9340 -1.0150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8416 -1.2682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6820 -1.8101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
2 10 1 0 0 0 0
10 11 2 0 0 0 0
11 5 1 0 0 0 0
6 12 2 0 0 0 0
7 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 1 1 0 0 0 0
16 28 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
30 17 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 26 1 0 0 0 0
3 40 1 0 0 0 0
40 41 1 0 0 0 0
24 42 1 0 0 0 0
42 43 1 0 0 0 0
11 44 1 0 0 0 0
44 45 1 0 0 0 0
36 46 1 0 0 0 0
46 47 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 46 47
M SBL 4 1 51
M SMT 4 CH2OH
M SVB 4 51 -4.3728 0.5002
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 42 43
M SBL 3 1 47
M SMT 3 CH2OH
M SVB 3 47 -2.2897 1.4283
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 44 45
M SBL 2 1 49
M SMT 2 OCH3
M SVB 2 49 0.2195 -0.6025
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 OCH3
M SVB 1 45 0.9373 1.4194
S SKP 8
ID FLIAFCGS0003
KNApSAcK_ID C00010136
NAME Platycarpanetin 7-O-laminaribioside
CAS_RN -
FORMULA C30H34O17
EXACTMASS 666.179599662
AVERAGEMASS 666.58076
SMILES C(O1)Oc(c2)c1cc(C(C6=O)=COc(c65)c(c(cc5OC)O[C@@H]([C@H]3O)OC(CO)[C@@H]([C@@H]3O[C@@H]([C@@H](O)4)OC([C@H](O)[C@@H]4O)CO)O)OC)c2
M END