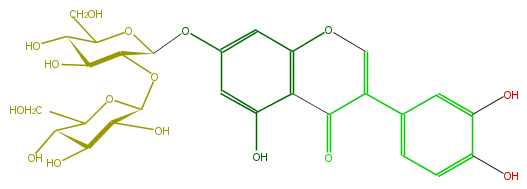
Mol:FLIAACGS0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4657 1.1022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4657 1.1022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7448 0.6859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7448 0.6859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0238 1.1022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0238 1.1022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6969 0.6860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6969 0.6860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6969 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6969 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4444 -0.6085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4444 -0.6085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1917 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1917 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1917 0.6860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1917 0.6860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4444 1.1176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4444 1.1176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7445 -0.1461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7445 -0.1461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0238 -0.5622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0238 -0.5622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4444 -1.4709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4444 -1.4709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9387 -0.6082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9387 -0.6082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9387 -1.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9387 -1.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6532 -1.8459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6532 -1.8459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3678 -1.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3678 -1.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3678 -0.6082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3678 -0.6082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6532 -0.1957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6532 -0.1957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8829 0.9824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8829 0.9824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4341 0.3902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4341 0.3902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7879 0.6415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7879 0.6415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1145 0.6339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1145 0.6339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6175 1.1013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6175 1.1013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2775 0.8644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2775 0.8644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4950 0.8030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4950 0.8030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0990 0.4285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0990 0.4285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0863 0.1629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0863 0.1629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0238 -1.4450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0238 -1.4450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0822 -1.8457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0822 -1.8457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0822 -0.1957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0822 -0.1957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1091 -0.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1091 -0.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4520 -1.2734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4520 -1.2734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9727 -0.7725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9727 -0.7725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3592 -0.4947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3592 -0.4947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0126 -0.2837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0126 -0.2837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5106 -0.7775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5106 -0.7775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6022 -1.4753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6022 -1.4753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0598 -1.5848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0598 -1.5848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0225 -0.9332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0225 -0.9332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8441 1.4630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8441 1.4630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0822 1.8459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0822 1.8459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3332 -0.5071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3332 -0.5071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0829 -0.8481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0829 -0.8481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 2 10 1 0 0 0 0 | + | 2 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 5 1 0 0 0 0 | + | 11 5 1 0 0 0 0 |
| − | 6 12 2 0 0 0 0 | + | 6 12 2 0 0 0 0 |
| − | 7 13 1 0 0 0 0 | + | 7 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 1 1 0 0 0 0 | + | 22 1 1 0 0 0 0 |
| − | 11 28 1 0 0 0 0 | + | 11 28 1 0 0 0 0 |
| − | 16 29 1 0 0 0 0 | + | 16 29 1 0 0 0 0 |
| − | 17 30 1 0 0 0 0 | + | 17 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 27 1 0 0 0 0 | + | 34 27 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 24 40 1 0 0 0 0 | + | 24 40 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 45 0.5665 -0.5987 | + | M SBV 1 45 0.5665 -0.5987 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 47 0.8226 -0.2703 | + | M SBV 2 47 0.8226 -0.2703 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FLIAACGS0004 | + | ID FLIAACGS0004 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES C(=O)(c23)C(=COc(cc(OC(O5)C(C(O)C(C5CO)O)OC(O4)C(C(O)C(C4CO)O)O)cc(O)3)2)c(c1)ccc(c(O)1)O | + | SMILES C(=O)(c23)C(=COc(cc(OC(O5)C(C(O)C(C5CO)O)OC(O4)C(C(O)C(C4CO)O)O)cc(O)3)2)c(c1)ccc(c(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.4657 1.1022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7448 0.6859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0238 1.1022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6969 0.6860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6969 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4444 -0.6085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1917 -0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1917 0.6860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4444 1.1176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7445 -0.1461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0238 -0.5622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4444 -1.4709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9387 -0.6082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9387 -1.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6532 -1.8459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3678 -1.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3678 -0.6082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6532 -0.1957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8829 0.9824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4341 0.3902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7879 0.6415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1145 0.6339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6175 1.1013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2775 0.8644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4950 0.8030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0990 0.4285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0863 0.1629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0238 -1.4450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0822 -1.8457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0822 -0.1957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1091 -0.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4520 -1.2734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9727 -0.7725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3592 -0.4947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0126 -0.2837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5106 -0.7775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6022 -1.4753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0598 -1.5848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0225 -0.9332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8441 1.4630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0822 1.8459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3332 -0.5071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0829 -0.8481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
2 10 1 0 0 0 0
10 11 2 0 0 0 0
11 5 1 0 0 0 0
6 12 2 0 0 0 0
7 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 1 1 0 0 0 0
11 28 1 0 0 0 0
16 29 1 0 0 0 0
17 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 27 1 0 0 0 0
40 41 1 0 0 0 0
24 40 1 0 0 0 0
42 43 1 0 0 0 0
36 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 ^ CH2OH
M SBV 1 45 0.5665 -0.5987
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 47
M SMT 2 ^ CH2OH
M SBV 2 47 0.8226 -0.2703
S SKP 5
ID FLIAACGS0004
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES C(=O)(c23)C(=COc(cc(OC(O5)C(C(O)C(C5CO)O)OC(O4)C(C(O)C(C4CO)O)O)cc(O)3)2)c(c1)ccc(c(O)1)O
M END