Mol:FLIA1AGS0009
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 39 43 0 0 0 0 0 0 0 0999 V2000 | + | 39 43 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1994 1.4211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1994 1.4211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4786 1.8373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4786 1.8373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7579 1.4213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7579 1.4213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7579 0.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7579 0.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0103 0.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0103 0.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7369 0.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7369 0.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7369 1.4213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7369 1.4213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0103 1.8529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0103 1.8529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1994 0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1994 0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4786 0.1731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4786 0.1731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0103 -0.5508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0103 -0.5508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4839 0.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4839 0.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4839 -0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4839 -0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1984 -1.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1984 -1.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9130 -0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9130 -0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9130 0.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9130 0.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1984 0.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1984 0.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6801 -1.1533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6801 -1.1533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8925 1.7795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8925 1.7795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2301 1.7537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2301 1.7537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7658 1.1410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7658 1.1410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0974 1.4010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0974 1.4010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4525 1.4079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4525 1.4079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9212 1.8767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9212 1.8767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6040 1.6315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6040 1.6315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8671 1.6383 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8671 1.6383 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4314 1.2492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4314 1.2492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4635 0.8815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4635 0.8815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0687 -0.6021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0687 -0.6021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6658 -1.0103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6658 -1.0103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3934 -1.0103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3934 -1.0103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0512 -0.5500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0512 -0.5500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0113 -0.1340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0113 -0.1340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6658 -0.5762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6658 -0.5762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3934 -0.5887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3934 -0.5887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0823 2.2032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0823 2.2032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.0512 2.6550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.0512 2.6550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3934 -1.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3934 -1.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1826 -2.6550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1826 -2.6550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 2 0 0 0 0 | + | 6 7 2 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 3 1 0 0 0 0 | + | 8 3 1 0 0 0 0 |
| − | 1 9 1 0 0 0 0 | + | 1 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 5 11 2 0 0 0 0 | + | 5 11 2 0 0 0 0 |
| − | 6 12 1 0 0 0 0 | + | 6 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 15 1 0 0 0 0 | + | 18 15 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 19 23 1 0 0 0 0 | + | 19 23 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 29 1 0 0 0 0 | + | 33 29 1 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 31 35 1 0 0 0 0 | + | 31 35 1 0 0 0 0 |
| − | 18 29 1 0 0 0 0 | + | 18 29 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 25 36 1 0 0 0 0 | + | 25 36 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 36 37 | + | M SAL 1 2 36 37 |
| − | M SBL 1 1 41 | + | M SBL 1 1 41 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 41 0.4783 -0.5717 | + | M SBV 1 41 0.4783 -0.5717 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 38 39 | + | M SAL 2 2 38 39 |
| − | M SBL 2 1 43 | + | M SBL 2 1 43 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 43 0.0000 0.6296 | + | M SBV 2 43 0.0000 0.6296 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FLIA1AGS0009 | + | ID FLIA1AGS0009 |
| − | FORMULA C26H28O13 | + | FORMULA C26H28O13 |
| − | EXACTMASS 548.152990982 | + | EXACTMASS 548.152990982 |
| − | AVERAGEMASS 548.49272 | + | AVERAGEMASS 548.49272 |
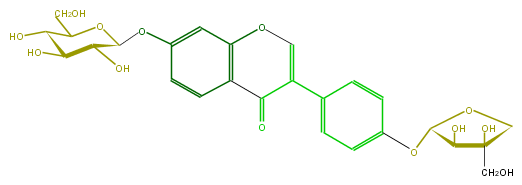
| − | SMILES O(c(c5)cc(c2c5)OC=C(c(c4)ccc(c4)OC(O3)C(C(O)(C3)CO)O)C2=O)C(C(O)1)OC(C(O)C(O)1)CO | + | SMILES O(c(c5)cc(c2c5)OC=C(c(c4)ccc(c4)OC(O3)C(C(O)(C3)CO)O)C2=O)C(C(O)1)OC(C(O)C(O)1)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
39 43 0 0 0 0 0 0 0 0999 V2000
-2.1994 1.4211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4786 1.8373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7579 1.4213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7579 0.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0103 0.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7369 0.5582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7369 1.4213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0103 1.8529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1994 0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4786 0.1731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0103 -0.5508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4839 0.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4839 -0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1984 -1.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9130 -0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9130 0.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1984 0.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6801 -1.1533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8925 1.7795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2301 1.7537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7658 1.1410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0974 1.4010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4525 1.4079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9212 1.8767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6040 1.6315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8671 1.6383 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4314 1.2492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4635 0.8815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0687 -0.6021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6658 -1.0103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3934 -1.0103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0512 -0.5500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0113 -0.1340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6658 -0.5762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3934 -0.5887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0823 2.2032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.0512 2.6550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3934 -1.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1826 -2.6550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 7 2 0 0 0 0
7 8 1 0 0 0 0
8 3 1 0 0 0 0
1 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
5 11 2 0 0 0 0
6 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 15 1 0 0 0 0
1 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
19 23 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 29 1 0 0 0 0
30 34 1 0 0 0 0
31 35 1 0 0 0 0
18 29 1 0 0 0 0
36 37 1 0 0 0 0
25 36 1 0 0 0 0
38 39 1 0 0 0 0
31 38 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 36 37
M SBL 1 1 41
M SMT 1 ^ CH2OH
M SBV 1 41 0.4783 -0.5717
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 38 39
M SBL 2 1 43
M SMT 2 CH2OH
M SBV 2 43 0.0000 0.6296
S SKP 5
ID FLIA1AGS0009
FORMULA C26H28O13
EXACTMASS 548.152990982
AVERAGEMASS 548.49272
SMILES O(c(c5)cc(c2c5)OC=C(c(c4)ccc(c4)OC(O3)C(C(O)(C3)CO)O)C2=O)C(C(O)1)OC(C(O)C(O)1)CO
M END