Mol:FL7DACGO0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.3497 1.1855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3497 1.1855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3497 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3497 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3648 -0.0520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3648 -0.0520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0792 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0792 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0792 1.1855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0792 1.1855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3648 1.5980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3648 1.5980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7937 -0.0520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7937 -0.0520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5082 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5082 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5082 1.1855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5082 1.1855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7937 1.5980 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 1.7937 1.5980 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2062 1.5885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2062 1.5885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9207 1.1760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9207 1.1760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6352 1.5885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6352 1.5885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6352 2.4135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6352 2.4135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9207 2.8260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9207 2.8260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2062 2.4135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2062 2.4135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2887 2.7908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2887 2.7908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3194 1.1934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3194 1.1934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3648 -0.7287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3648 -0.7287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9526 1.5335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9526 1.5335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1797 -0.7177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1797 -0.7177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7671 -1.4324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7671 -1.4324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9688 -1.2232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9688 -1.2232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1753 -1.4501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1753 -1.4501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5878 -0.7353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5878 -0.7353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3862 -0.9444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3862 -0.9444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5432 -0.3586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5432 -0.3586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0231 -0.0816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0231 -0.0816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6629 -1.2009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6629 -1.2009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2158 -1.1734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2158 -1.1734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1402 -1.8632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1402 -1.8632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8362 -1.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8362 -1.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4236 -2.0264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4236 -2.0264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6253 -1.8172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6253 -1.8172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8318 -2.0441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8318 -2.0441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2443 -1.3293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2443 -1.3293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0427 -1.5384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0427 -1.5384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1997 -0.9526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1997 -0.9526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6796 -0.6756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6796 -0.6756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3194 -1.7949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3194 -1.7949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8723 -1.7674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8723 -1.7674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7967 -2.4572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7967 -2.4572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1402 -2.5277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1402 -2.5277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6569 -2.8260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6569 -2.8260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6646 -2.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6646 -2.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 13 18 1 0 0 0 0 | + | 13 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 19 24 1 0 0 0 0 | + | 19 24 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 31 43 1 0 0 0 0 | + | 31 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7DACGS0005 | + | ID FL7DACGS0005 |
| − | KNApSAcK_ID C00014756 | + | KNApSAcK_ID C00014756 |
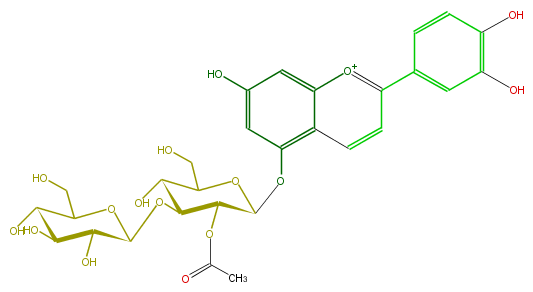
| − | NAME Luteolinidin 5-(3-glucoside-2-acetylglucoside) | + | NAME Luteolinidin 5-(3-glucoside-2-acetylglucoside) |
| − | CAS_RN 347861-61-4 | + | CAS_RN 347861-61-4 |
| − | FORMULA C29H33O16 | + | FORMULA C29H33O16 |
| − | EXACTMASS 637.176860008 | + | EXACTMASS 637.176860008 |
| − | AVERAGEMASS 637.56272 | + | AVERAGEMASS 637.56272 |
| − | SMILES c(c12)cc(c(c5)ccc(O)c5O)[o+1]c1cc(cc2OC(C(OC(C)=O)3)OC(C(C3OC(O4)C(O)C(C(O)C4CO)O)O)CO)O | + | SMILES c(c12)cc(c(c5)ccc(O)c5O)[o+1]c1cc(cc2OC(C(OC(C)=O)3)OC(C(C3OC(O4)C(O)C(C(O)C4CO)O)O)CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
-0.3497 1.1855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3497 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3648 -0.0520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0792 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0792 1.1855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3648 1.5980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7937 -0.0520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5082 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5082 1.1855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7937 1.5980 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
3.2062 1.5885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9207 1.1760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6352 1.5885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6352 2.4135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9207 2.8260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2062 2.4135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2887 2.7908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3194 1.1934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3648 -0.7287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9526 1.5335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1797 -0.7177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7671 -1.4324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9688 -1.2232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1753 -1.4501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5878 -0.7353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3862 -0.9444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5432 -0.3586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0231 -0.0816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6629 -1.2009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2158 -1.1734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1402 -1.8632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8362 -1.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4236 -2.0264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6253 -1.8172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8318 -2.0441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2443 -1.3293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0427 -1.5384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1997 -0.9526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6796 -0.6756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3194 -1.7949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8723 -1.7674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7967 -2.4572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1402 -2.5277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6569 -2.8260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6646 -2.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
13 18 1 0 0 0 0
3 19 1 0 0 0 0
1 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
21 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
19 24 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 30 1 0 0 0 0
31 43 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
S SKP 8
ID FL7DACGS0005
KNApSAcK_ID C00014756
NAME Luteolinidin 5-(3-glucoside-2-acetylglucoside)
CAS_RN 347861-61-4
FORMULA C29H33O16
EXACTMASS 637.176860008
AVERAGEMASS 637.56272
SMILES c(c12)cc(c(c5)ccc(O)c5O)[o+1]c1cc(cc2OC(C(OC(C)=O)3)OC(C(C3OC(O4)C(O)C(C(O)C4CO)O)O)CO)O
M END