Mol:FL7AAIGLS002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 60 0 0 0 0 0 0 0 0999 V2000 | + | 56 60 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.7341 -0.1059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7341 -0.1059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1236 0.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1236 0.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1236 0.9515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1236 0.9515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7341 1.3039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7341 1.3039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3445 0.9515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3445 0.9515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3445 0.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3445 0.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4868 -0.1059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4868 -0.1059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0973 0.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0973 0.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0973 0.9515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0973 0.9515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4868 1.3039 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.4868 1.3039 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6788 1.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6788 1.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2624 0.9503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2624 0.9503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8459 1.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8459 1.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8459 1.9610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8459 1.9610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2624 2.2980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2624 2.2980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6788 1.9610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6788 1.9610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3972 2.2793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3972 2.2793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8439 1.2398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8439 1.2398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7341 -0.6009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7341 -0.6009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6685 -0.0832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6685 -0.0832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2624 2.8817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2624 2.8817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8039 3.1943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8039 3.1943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3678 0.9859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3678 0.9859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9283 0.9859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9283 0.9859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1154 -1.2652 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.1154 -1.2652 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.4749 -1.7852 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.4749 -1.7852 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.8042 -1.3047 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.8042 -1.3047 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.9823 -1.2331 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.9823 -1.2331 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.6228 -0.7131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6228 -0.7131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2936 -1.1935 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.2936 -1.1935 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.8213 -0.7989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8213 -0.7989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8213 -0.2541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8213 -0.2541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5371 -1.6869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5371 -1.6869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7513 -2.2638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7513 -2.2638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4645 -1.8931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4645 -1.8931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4645 -2.5336 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4645 -2.5336 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0645 -2.5336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0645 -2.5336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8246 -2.5336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8246 -2.5336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4645 -3.1943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4645 -3.1943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7590 -0.2032 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.7590 -0.2032 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.1735 -0.7844 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1735 -0.7844 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.9985 -0.7738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9985 -0.7738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7137 -1.1850 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.7137 -1.1850 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.2993 -0.6038 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.2993 -0.6038 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.4743 -0.6144 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.4743 -0.6144 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.2709 -0.3340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2709 -0.3340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9064 -0.1823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9064 -0.1823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4108 -1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4108 -1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1385 -2.8642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1385 -2.8642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2993 -0.6038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2993 -0.6038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2679 0.1925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2679 0.1925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7641 -0.3037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7641 -0.3037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2661 0.1984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2661 0.1984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7519 -0.2873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7519 -0.2873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2661 0.7687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2661 0.7687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2679 0.6696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2679 0.6696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 3 1 0 0 0 0 | + | 10 3 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 5 18 1 0 0 0 0 | + | 5 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 13 23 1 0 0 0 0 | + | 13 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 25 1 0 0 0 0 | + | 30 25 1 0 0 0 0 |
| − | 19 28 1 0 0 0 0 | + | 19 28 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 27 35 1 0 0 0 0 | + | 27 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 36 38 2 0 0 0 0 | + | 36 38 2 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 43 42 1 1 0 0 0 | + | 43 42 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 40 1 0 0 0 0 | + | 45 40 1 0 0 0 0 |
| − | 20 41 1 0 0 0 0 | + | 20 41 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 43 48 1 0 0 0 0 | + | 43 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 32 51 1 0 0 0 0 | + | 32 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 2 0 0 0 0 | + | 53 54 2 0 0 0 0 |
| − | 53 55 1 0 0 0 0 | + | 53 55 1 0 0 0 0 |
| − | 51 56 2 0 0 0 0 | + | 51 56 2 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 48 49 | + | M SAL 4 2 48 49 |
| − | M SBL 4 1 52 | + | M SBL 4 1 52 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 52 4.3768 -1.0074 | + | M SVB 4 52 4.3768 -1.0074 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 23 24 | + | M SAL 3 2 23 24 |
| − | M SBL 3 1 25 | + | M SBL 3 1 25 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 25 3.3678 0.9859 | + | M SVB 3 25 3.3678 0.9859 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 21 22 | + | M SAL 2 2 21 22 |
| − | M SBL 2 1 23 | + | M SBL 2 1 23 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 23 2.2624 2.8817 | + | M SVB 2 23 2.2624 2.8817 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 53 54 55 | + | M SAL 1 3 53 54 55 |
| − | M SBL 1 1 57 | + | M SBL 1 1 57 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SVB 1 57 -4.2661 0.1984 | + | M SVB 1 57 -4.2661 0.1984 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAIGLS002 | + | ID FL7AAIGLS002 |
| − | KNApSAcK_ID C00011344 | + | KNApSAcK_ID C00011344 |
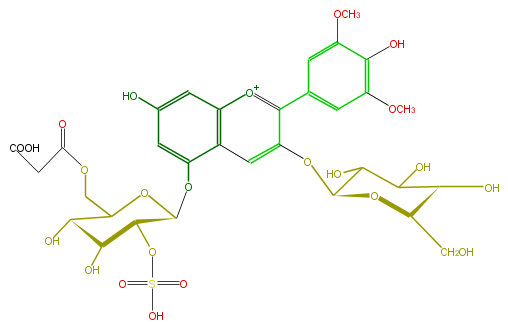
| − | NAME Malvidin 3-glucoside-5-(6'''-malonyl-2'''-sulfatoglucoside) | + | NAME Malvidin 3-glucoside-5-(6'''-malonyl-2'''-sulfatoglucoside) |
| − | CAS_RN 160206-03-1 | + | CAS_RN 160206-03-1 |
| − | FORMULA C32H37O23S | + | FORMULA C32H37O23S |
| − | EXACTMASS 821.14463318 | + | EXACTMASS 821.14463318 |
| − | AVERAGEMASS 821.68838 | + | AVERAGEMASS 821.68838 |
| − | SMILES C(O)([C@@H]1Oc(c3c(c5)cc(OC)c(O)c(OC)5)cc(c2O[C@@H]([C@@H](OS(O)(=O)=O)4)OC(COC(CC(O)=O)=O)[C@H](O)[C@H](O)4)c([o+1]3)cc(c2)O)C([C@H]([C@H](O1)CO)O)O | + | SMILES C(O)([C@@H]1Oc(c3c(c5)cc(OC)c(O)c(OC)5)cc(c2O[C@@H]([C@@H](OS(O)(=O)=O)4)OC(COC(CC(O)=O)=O)[C@H](O)[C@H](O)4)c([o+1]3)cc(c2)O)C([C@H]([C@H](O1)CO)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 60 0 0 0 0 0 0 0 0999 V2000
-0.7341 -0.1059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1236 0.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1236 0.9515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7341 1.3039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3445 0.9515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3445 0.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4868 -0.1059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0973 0.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0973 0.9515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4868 1.3039 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.6788 1.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2624 0.9503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8459 1.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8459 1.9610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2624 2.2980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6788 1.9610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3972 2.2793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8439 1.2398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7341 -0.6009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6685 -0.0832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2624 2.8817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8039 3.1943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3678 0.9859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9283 0.9859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1154 -1.2652 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.4749 -1.7852 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.8042 -1.3047 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.9823 -1.2331 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.6228 -0.7131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2936 -1.1935 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.8213 -0.7989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8213 -0.2541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5371 -1.6869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7513 -2.2638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4645 -1.8931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4645 -2.5336 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
-2.0645 -2.5336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8246 -2.5336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4645 -3.1943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7590 -0.2032 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.1735 -0.7844 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.9985 -0.7738 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7137 -1.1850 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.2993 -0.6038 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.4743 -0.6144 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.2709 -0.3340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9064 -0.1823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4108 -1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1385 -2.8642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2993 -0.6038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2679 0.1925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7641 -0.3037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2661 0.1984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7519 -0.2873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2661 0.7687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2679 0.6696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
2 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 3 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
5 18 1 0 0 0 0
1 19 1 0 0 0 0
8 20 1 0 0 0 0
15 21 1 0 0 0 0
21 22 1 0 0 0 0
13 23 1 0 0 0 0
23 24 1 0 0 0 0
25 26 1 1 0 0 0
26 27 1 1 0 0 0
28 27 1 1 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
30 25 1 0 0 0 0
19 28 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
27 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
36 38 2 0 0 0 0
36 39 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 1 0 0 0
43 42 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 40 1 0 0 0 0
20 41 1 0 0 0 0
40 46 1 0 0 0 0
45 47 1 0 0 0 0
43 48 1 0 0 0 0
48 49 1 0 0 0 0
44 50 1 0 0 0 0
32 51 1 0 0 0 0
51 52 1 0 0 0 0
52 53 1 0 0 0 0
53 54 2 0 0 0 0
53 55 1 0 0 0 0
51 56 2 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 48 49
M SBL 4 1 52
M SMT 4 CH2OH
M SVB 4 52 4.3768 -1.0074
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 23 24
M SBL 3 1 25
M SMT 3 OCH3
M SVB 3 25 3.3678 0.9859
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 21 22
M SBL 2 1 23
M SMT 2 OCH3
M SVB 2 23 2.2624 2.8817
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 53 54 55
M SBL 1 1 57
M SMT 1 COOH
M SVB 1 57 -4.2661 0.1984
S SKP 8
ID FL7AAIGLS002
KNApSAcK_ID C00011344
NAME Malvidin 3-glucoside-5-(6'''-malonyl-2'''-sulfatoglucoside)
CAS_RN 160206-03-1
FORMULA C32H37O23S
EXACTMASS 821.14463318
AVERAGEMASS 821.68838
SMILES C(O)([C@@H]1Oc(c3c(c5)cc(OC)c(O)c(OC)5)cc(c2O[C@@H]([C@@H](OS(O)(=O)=O)4)OC(COC(CC(O)=O)=O)[C@H](O)[C@H](O)4)c([o+1]3)cc(c2)O)C([C@H]([C@H](O1)CO)O)O
M END