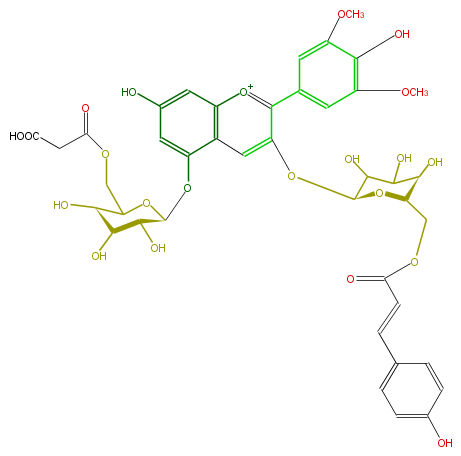
Mol:FL7AAIGL0020
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 63 68 0 0 0 0 0 0 0 0999 V2000 | + | 63 68 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.2971 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2971 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2971 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2971 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7408 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7408 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1845 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1845 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1845 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1845 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7408 1.4708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7408 1.4708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6282 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6282 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0719 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0719 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0719 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0719 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6282 1.4708 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.6282 1.4708 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5158 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5158 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0512 1.1434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0512 1.1434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6181 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6181 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6181 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6181 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0512 2.4528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0512 2.4528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5158 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5158 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8532 1.4707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8532 1.4707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4841 2.6254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4841 2.6254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7408 -0.4560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7408 -0.4560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6558 -0.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6558 -0.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8470 -0.2372 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.8470 -0.2372 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.5788 -0.7017 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.5788 -0.7017 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.0946 -0.5543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0946 -0.5543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6136 -0.7017 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.6136 -0.7017 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.8818 -0.2372 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.8818 -0.2372 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.3660 -0.3845 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.3660 -0.3845 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.4826 0.1274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4826 0.1274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5077 0.1442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5077 0.1442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1435 0.0747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1435 0.0747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0159 -1.1041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0159 -1.1041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7947 -1.9294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7947 -1.9294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1851 -2.2814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1851 -2.2814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5193 -2.2814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5193 -2.2814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4727 -2.7774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4727 -2.7774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0737 -3.3651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0737 -3.3651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4267 -3.9766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4267 -3.9766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1219 -4.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1219 -4.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4267 -5.0324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4267 -5.0324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0363 -5.0325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0363 -5.0325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3412 -4.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3412 -4.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0364 -3.9766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0364 -3.9766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3409 -5.5600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3409 -5.5600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6108 -0.8539 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.6108 -0.8539 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -4.2397 -1.3439 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -4.2397 -1.3439 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.7052 -1.1360 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.7052 -1.1360 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.1894 -1.1305 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.1894 -1.1305 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.5642 -0.7556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5642 -0.7556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0318 -1.0024 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -4.0318 -1.0024 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -5.2018 -0.8022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2018 -0.8022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5900 -1.8051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5900 -1.8051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3989 -1.6502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3989 -1.6502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3811 -0.3975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3811 -0.3975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3811 0.2255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3811 0.2255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7803 0.6247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7803 0.6247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3079 0.3201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3079 0.3201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7809 0.5932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7809 0.5932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1846 0.3601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1846 0.3601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7809 1.1390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7809 1.1390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7803 1.1503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7803 1.1503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3346 3.0289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3346 3.0289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7760 3.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7760 3.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6181 1.4707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6181 1.4707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6181 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6181 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 46 19 1 0 0 0 0 | + | 46 19 1 0 0 0 0 |
| − | 48 52 1 0 0 0 0 | + | 48 52 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 2 0 0 0 0 | + | 56 57 2 0 0 0 0 |
| − | 56 58 1 0 0 0 0 | + | 56 58 1 0 0 0 0 |
| − | 54 59 2 0 0 0 0 | + | 54 59 2 0 0 0 0 |
| − | 15 60 1 0 0 0 0 | + | 15 60 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 13 62 1 0 0 0 0 | + | 13 62 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 62 63 | + | M SAL 3 2 62 63 |
| − | M SBL 3 1 67 | + | M SBL 3 1 67 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 67 0.8277 1.2645 | + | M SVB 3 67 0.8277 1.2645 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 60 61 | + | M SAL 2 2 60 61 |
| − | M SBL 2 1 65 | + | M SBL 2 1 65 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 65 0.3346 3.0289 | + | M SVB 2 65 0.3346 3.0289 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 56 57 58 | + | M SAL 1 3 56 57 58 |
| − | M SBL 1 1 61 | + | M SBL 1 1 61 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SVB 1 61 -5.7809 0.5932 | + | M SVB 1 61 -5.7809 0.5932 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAIGL0020 | + | ID FL7AAIGL0020 |
| − | KNApSAcK_ID C00006911 | + | KNApSAcK_ID C00006911 |
| − | NAME Salviamalvin | + | NAME Salviamalvin |
| − | CAS_RN 128508-48-5 | + | CAS_RN 128508-48-5 |
| − | FORMULA C41H43O22 | + | FORMULA C41H43O22 |
| − | EXACTMASS 887.22459806 | + | EXACTMASS 887.22459806 |
| − | AVERAGEMASS 887.76692 | + | AVERAGEMASS 887.76692 |
| − | SMILES [C@@H]([C@@H](O)6)([C@H](O)[C@@H](OC6COC(=O)CC(O)=O)Oc(c21)cc(cc1[o+1]c(c(c5)cc(c(c5OC)O)OC)c(O[C@H](O3)C(C([C@@H](O)[C@@H](COC(C=Cc(c4)ccc(O)c4)=O)3)O)O)c2)O)O | + | SMILES [C@@H]([C@@H](O)6)([C@H](O)[C@@H](OC6COC(=O)CC(O)=O)Oc(c21)cc(cc1[o+1]c(c(c5)cc(c(c5OC)O)OC)c(O[C@H](O3)C(C([C@@H](O)[C@@H](COC(C=Cc(c4)ccc(O)c4)=O)3)O)O)c2)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
63 68 0 0 0 0 0 0 0 0999 V2000
-3.2971 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2971 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7408 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1845 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1845 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7408 1.4708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6282 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0719 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0719 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6282 1.4708 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-0.5158 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0512 1.1434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6181 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6181 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0512 2.4528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5158 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8532 1.4707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4841 2.6254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7408 -0.4560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6558 -0.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8470 -0.2372 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.5788 -0.7017 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.0946 -0.5543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6136 -0.7017 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.8818 -0.2372 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.3660 -0.3845 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.4826 0.1274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5077 0.1442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1435 0.0747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0159 -1.1041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7947 -1.9294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1851 -2.2814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5193 -2.2814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4727 -2.7774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0737 -3.3651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4267 -3.9766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1219 -4.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4267 -5.0324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0363 -5.0325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3412 -4.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0364 -3.9766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3409 -5.5600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6108 -0.8539 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-4.2397 -1.3439 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.7052 -1.1360 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.1894 -1.1305 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.5642 -0.7556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0318 -1.0024 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-5.2018 -0.8022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5900 -1.8051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3989 -1.6502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3811 -0.3975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3811 0.2255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7803 0.6247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3079 0.3201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7809 0.5932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.1846 0.3601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7809 1.1390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7803 1.1503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3346 3.0289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7760 3.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6181 1.4707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6181 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
24 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 51 1 0 0 0 0
46 19 1 0 0 0 0
48 52 1 0 0 0 0
52 53 1 0 0 0 0
53 54 1 0 0 0 0
54 55 1 0 0 0 0
55 56 1 0 0 0 0
56 57 2 0 0 0 0
56 58 1 0 0 0 0
54 59 2 0 0 0 0
15 60 1 0 0 0 0
60 61 1 0 0 0 0
13 62 1 0 0 0 0
62 63 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 62 63
M SBL 3 1 67
M SMT 3 OCH3
M SVB 3 67 0.8277 1.2645
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 60 61
M SBL 2 1 65
M SMT 2 OCH3
M SVB 2 65 0.3346 3.0289
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 56 57 58
M SBL 1 1 61
M SMT 1 COOH
M SVB 1 61 -5.7809 0.5932
S SKP 8
ID FL7AAIGL0020
KNApSAcK_ID C00006911
NAME Salviamalvin
CAS_RN 128508-48-5
FORMULA C41H43O22
EXACTMASS 887.22459806
AVERAGEMASS 887.76692
SMILES [C@@H]([C@@H](O)6)([C@H](O)[C@@H](OC6COC(=O)CC(O)=O)Oc(c21)cc(cc1[o+1]c(c(c5)cc(c(c5OC)O)OC)c(O[C@H](O3)C(C([C@@H](O)[C@@H](COC(C=Cc(c4)ccc(O)c4)=O)3)O)O)c2)O)O
M END