Mol:FL7AAGGL0061
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 91 98 0 0 0 0 0 0 0 0999 V2000 | + | 91 98 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.6321 0.8666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6321 0.8666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6321 0.0416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6321 0.0416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0824 -0.3709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0824 -0.3709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7968 0.0416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7968 0.0416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7968 0.8666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7968 0.8666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0824 1.2791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0824 1.2791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5113 -0.3709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5113 -0.3709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2258 0.0416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2258 0.0416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2258 0.8666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2258 0.8666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5113 1.2791 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 1.5113 1.2791 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0023 1.3149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0023 1.3149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7168 0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7168 0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4313 1.3149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4313 1.3149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4313 2.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4313 2.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7168 2.5524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7168 2.5524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0023 2.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0023 2.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0715 2.5095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0715 2.5095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0843 -0.4541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0843 -0.4541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2121 1.2014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2121 1.2014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0824 -1.1336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0824 -1.1336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7168 3.2836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7168 3.2836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0963 0.9310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0963 0.9310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3354 -2.1048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3354 -2.1048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0952 -2.4268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0952 -2.4268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6065 -1.7791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6065 -1.7791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3766 -1.4823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3766 -1.4823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6168 -1.1602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6168 -1.1602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1053 -1.8079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1053 -1.8079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6272 -1.4348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6272 -1.4348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0175 -2.5877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0175 -2.5877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5810 -2.4902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5810 -2.4902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8551 -2.3932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8551 -2.3932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9476 1.3241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9476 1.3241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5350 0.6094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5350 0.6094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7367 0.8185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7367 0.8185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9432 0.5917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9432 0.5917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3558 1.3064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3558 1.3064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1542 1.0973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1542 1.0973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3111 1.6832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3111 1.6832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4309 0.8409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4309 0.8409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9837 0.8684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9837 0.8684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9082 0.1786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9082 0.1786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7782 -4.6396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7782 -4.6396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3727 -3.9225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3727 -3.9225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4512 -3.9151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4512 -3.9151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7910 -3.2127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7910 -3.2127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3855 -2.4955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3855 -2.4955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4384 -2.4881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4384 -2.4881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8038 -1.7857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8038 -1.7857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8374 1.9328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8374 1.9328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2584 2.6409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2584 2.6409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8557 3.3597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8557 3.3597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0822 2.6304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0822 2.6304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5033 3.3385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5033 3.3385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.3271 3.3280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.3271 3.3280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.7487 4.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.7487 4.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.5737 4.0265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.5737 4.0265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.9770 3.3068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.9770 3.3068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.5553 2.5977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.5553 2.5977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.7304 2.6083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.7304 2.6083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -8.8008 3.2962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -8.8008 3.2962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.9581 1.8789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.9581 1.8789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -8.7819 1.8684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -8.7819 1.8684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.9947 4.7347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.9947 4.7347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -8.8185 4.7241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -8.8185 4.7241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5018 -1.4443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5018 -1.4443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3256 -1.3957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3256 -1.3957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4964 -0.5882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4964 -0.5882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0548 0.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0548 0.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2309 -0.0291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2309 -0.0291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0600 -0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0600 -0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1019 -0.7884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1019 -0.7884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7172 -2.0928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7172 -2.0928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8930 -1.6807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8930 -1.6807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9916 -1.0283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9916 -1.0283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8109 -1.7723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8109 -1.7723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2731 -2.5099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2731 -2.5099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8481 -3.2157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8481 -3.2157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0968 -2.5251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0968 -2.5251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4956 -3.2461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4956 -3.2461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3193 -3.2613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3193 -3.2613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7186 -3.9833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7186 -3.9833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5434 -3.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5434 -3.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9690 -3.2917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9690 -3.2917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5698 -2.5698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5698 -2.5698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7449 -2.5546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7449 -2.5546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7928 -3.3069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7928 -3.3069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9948 -1.8640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9948 -1.8640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8185 -1.8792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8185 -1.8792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9421 -4.7195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9421 -4.7195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.7659 -4.7347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.7659 -4.7347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 13 22 1 0 0 0 0 | + | 13 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 26 18 1 0 0 0 0 | + | 26 18 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 47 49 1 0 0 0 0 | + | 47 49 1 0 0 0 0 |
| − | 49 29 1 0 0 0 0 | + | 49 29 1 0 0 0 0 |
| − | 36 19 1 0 0 0 0 | + | 36 19 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 51 53 1 0 0 0 0 | + | 51 53 1 0 0 0 0 |
| − | 53 54 2 0 0 0 0 | + | 53 54 2 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 2 0 0 0 0 | + | 55 56 2 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 2 0 0 0 0 | + | 57 58 2 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 2 0 0 0 0 | + | 59 60 2 0 0 0 0 |
| − | 60 55 1 0 0 0 0 | + | 60 55 1 0 0 0 0 |
| − | 58 61 1 0 0 0 0 | + | 58 61 1 0 0 0 0 |
| − | 59 62 1 0 0 0 0 | + | 59 62 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 57 64 1 0 0 0 0 | + | 57 64 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 50 39 1 0 0 0 0 | + | 50 39 1 0 0 0 0 |
| − | 66 67 1 1 0 0 0 | + | 66 67 1 1 0 0 0 |
| − | 67 68 1 1 0 0 0 | + | 67 68 1 1 0 0 0 |
| − | 69 68 1 1 0 0 0 | + | 69 68 1 1 0 0 0 |
| − | 69 70 1 0 0 0 0 | + | 69 70 1 0 0 0 0 |
| − | 70 71 1 0 0 0 0 | + | 70 71 1 0 0 0 0 |
| − | 71 66 1 0 0 0 0 | + | 71 66 1 0 0 0 0 |
| − | 71 72 1 0 0 0 0 | + | 71 72 1 0 0 0 0 |
| − | 66 73 1 0 0 0 0 | + | 66 73 1 0 0 0 0 |
| − | 67 74 1 0 0 0 0 | + | 67 74 1 0 0 0 0 |
| − | 68 75 1 0 0 0 0 | + | 68 75 1 0 0 0 0 |
| − | 76 77 1 0 0 0 0 | + | 76 77 1 0 0 0 0 |
| − | 77 78 2 0 0 0 0 | + | 77 78 2 0 0 0 0 |
| − | 77 79 1 0 0 0 0 | + | 77 79 1 0 0 0 0 |
| − | 79 80 2 0 0 0 0 | + | 79 80 2 0 0 0 0 |
| − | 80 81 1 0 0 0 0 | + | 80 81 1 0 0 0 0 |
| − | 81 82 2 0 0 0 0 | + | 81 82 2 0 0 0 0 |
| − | 82 83 1 0 0 0 0 | + | 82 83 1 0 0 0 0 |
| − | 83 84 2 0 0 0 0 | + | 83 84 2 0 0 0 0 |
| − | 84 85 1 0 0 0 0 | + | 84 85 1 0 0 0 0 |
| − | 85 86 2 0 0 0 0 | + | 85 86 2 0 0 0 0 |
| − | 86 81 1 0 0 0 0 | + | 86 81 1 0 0 0 0 |
| − | 84 87 1 0 0 0 0 | + | 84 87 1 0 0 0 0 |
| − | 85 88 1 0 0 0 0 | + | 85 88 1 0 0 0 0 |
| − | 88 89 1 0 0 0 0 | + | 88 89 1 0 0 0 0 |
| − | 83 90 1 0 0 0 0 | + | 83 90 1 0 0 0 0 |
| − | 90 91 1 0 0 0 0 | + | 90 91 1 0 0 0 0 |
| − | 76 72 1 0 0 0 0 | + | 76 72 1 0 0 0 0 |
| − | 69 22 1 0 0 0 0 | + | 69 22 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAGGL0061 | + | ID FL7AAGGL0061 |
| − | KNApSAcK_ID C00014812 | + | KNApSAcK_ID C00014812 |
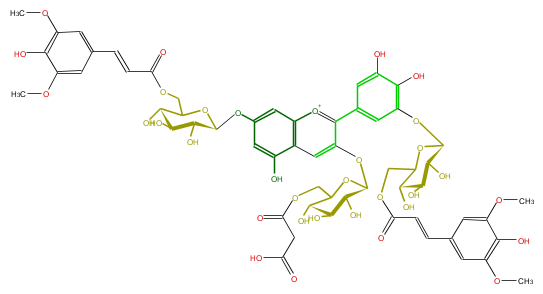
| − | NAME Delphinidin 3-(6-malonylglucoside)-7,3'-di-(6-sinapoylglucoside) | + | NAME Delphinidin 3-(6-malonylglucoside)-7,3'-di-(6-sinapoylglucoside) |
| − | CAS_RN 799854-16-3 | + | CAS_RN 799854-16-3 |
| − | FORMULA C58H63O33 | + | FORMULA C58H63O33 |
| − | EXACTMASS 1287.325159542 | + | EXACTMASS 1287.325159542 |
| − | AVERAGEMASS 1288.10102 | + | AVERAGEMASS 1288.10102 |
| − | SMILES OC(C1Oc(c(c(c8)cc(c(c(O)8)O)OC(O6)C(O)C(O)C(O)C6COC(C=Cc(c7)cc(c(O)c7OC)OC)=O)5)cc(c([o+1]5)2)c(O)cc(OC(O3)C(O)C(O)C(C3COC(=O)C=Cc(c4)cc(OC)c(O)c4OC)O)c2)C(O)C(O)C(O1)COC(=O)CC(O)=O | + | SMILES OC(C1Oc(c(c(c8)cc(c(c(O)8)O)OC(O6)C(O)C(O)C(O)C6COC(C=Cc(c7)cc(c(O)c7OC)OC)=O)5)cc(c([o+1]5)2)c(O)cc(OC(O3)C(O)C(O)C(C3COC(=O)C=Cc(c4)cc(OC)c(O)c4OC)O)c2)C(O)C(O)C(O1)COC(=O)CC(O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
91 98 0 0 0 0 0 0 0 0999 V2000
-0.6321 0.8666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6321 0.0416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0824 -0.3709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7968 0.0416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7968 0.8666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0824 1.2791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5113 -0.3709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2258 0.0416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2258 0.8666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5113 1.2791 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
3.0023 1.3149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7168 0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4313 1.3149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4313 2.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7168 2.5524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0023 2.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0715 2.5095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0843 -0.4541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2121 1.2014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0824 -1.1336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7168 3.2836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0963 0.9310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3354 -2.1048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0952 -2.4268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6065 -1.7791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3766 -1.4823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6168 -1.1602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1053 -1.8079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6272 -1.4348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0175 -2.5877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5810 -2.4902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8551 -2.3932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9476 1.3241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5350 0.6094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7367 0.8185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9432 0.5917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3558 1.3064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1542 1.0973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3111 1.6832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4309 0.8409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9837 0.8684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9082 0.1786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7782 -4.6396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3727 -3.9225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4512 -3.9151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7910 -3.2127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3855 -2.4955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4384 -2.4881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8038 -1.7857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8374 1.9328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2584 2.6409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8557 3.3597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0822 2.6304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5033 3.3385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.3271 3.3280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.7487 4.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.5737 4.0265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.9770 3.3068 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.5553 2.5977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.7304 2.6083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-8.8008 3.2962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-7.9581 1.8789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-8.7819 1.8684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.9947 4.7347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-8.8185 4.7241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5018 -1.4443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3256 -1.3957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4964 -0.5882 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0548 0.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2309 -0.0291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0600 -0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1019 -0.7884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7172 -2.0928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8930 -1.6807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9916 -1.0283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8109 -1.7723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2731 -2.5099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8481 -3.2157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0968 -2.5251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4956 -3.2461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3193 -3.2613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.7186 -3.9833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5434 -3.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9690 -3.2917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5698 -2.5698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.7449 -2.5546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.7928 -3.3069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.9948 -1.8640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.8185 -1.8792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9421 -4.7195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.7659 -4.7347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
8 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
15 21 1 0 0 0 0
13 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
25 32 1 0 0 0 0
26 18 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
33 40 1 0 0 0 0
34 41 1 0 0 0 0
35 42 1 0 0 0 0
43 44 2 0 0 0 0
44 45 1 0 0 0 0
44 46 1 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
47 49 1 0 0 0 0
49 29 1 0 0 0 0
36 19 1 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
51 53 1 0 0 0 0
53 54 2 0 0 0 0
54 55 1 0 0 0 0
55 56 2 0 0 0 0
56 57 1 0 0 0 0
57 58 2 0 0 0 0
58 59 1 0 0 0 0
59 60 2 0 0 0 0
60 55 1 0 0 0 0
58 61 1 0 0 0 0
59 62 1 0 0 0 0
62 63 1 0 0 0 0
57 64 1 0 0 0 0
64 65 1 0 0 0 0
50 39 1 0 0 0 0
66 67 1 1 0 0 0
67 68 1 1 0 0 0
69 68 1 1 0 0 0
69 70 1 0 0 0 0
70 71 1 0 0 0 0
71 66 1 0 0 0 0
71 72 1 0 0 0 0
66 73 1 0 0 0 0
67 74 1 0 0 0 0
68 75 1 0 0 0 0
76 77 1 0 0 0 0
77 78 2 0 0 0 0
77 79 1 0 0 0 0
79 80 2 0 0 0 0
80 81 1 0 0 0 0
81 82 2 0 0 0 0
82 83 1 0 0 0 0
83 84 2 0 0 0 0
84 85 1 0 0 0 0
85 86 2 0 0 0 0
86 81 1 0 0 0 0
84 87 1 0 0 0 0
85 88 1 0 0 0 0
88 89 1 0 0 0 0
83 90 1 0 0 0 0
90 91 1 0 0 0 0
76 72 1 0 0 0 0
69 22 1 0 0 0 0
S SKP 8
ID FL7AAGGL0061
KNApSAcK_ID C00014812
NAME Delphinidin 3-(6-malonylglucoside)-7,3'-di-(6-sinapoylglucoside)
CAS_RN 799854-16-3
FORMULA C58H63O33
EXACTMASS 1287.325159542
AVERAGEMASS 1288.10102
SMILES OC(C1Oc(c(c(c8)cc(c(c(O)8)O)OC(O6)C(O)C(O)C(O)C6COC(C=Cc(c7)cc(c(O)c7OC)OC)=O)5)cc(c([o+1]5)2)c(O)cc(OC(O3)C(O)C(O)C(C3COC(=O)C=Cc(c4)cc(OC)c(O)c4OC)O)c2)C(O)C(O)C(O1)COC(=O)CC(O)=O
M END