Mol:FL7AADGL0015
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.9203 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9203 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9203 1.1987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9203 1.1987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3640 0.8775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3640 0.8775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8077 1.1987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8077 1.1987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8077 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8077 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3640 2.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3640 2.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2514 0.8775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2514 0.8775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6951 1.1987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6951 1.1987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6951 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6951 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2514 2.1622 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.2514 2.1622 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1390 2.1621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1390 2.1621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4279 1.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4279 1.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9949 2.1621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9949 2.1621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9949 2.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9949 2.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4279 3.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4279 3.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1390 2.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1390 2.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4764 2.1621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4764 2.1621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8609 3.3168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8609 3.3168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3640 0.2354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3640 0.2354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2790 0.4780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2790 0.4780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2238 0.4542 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.2238 0.4542 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.9556 -0.0104 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.9556 -0.0104 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.4714 0.1371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4714 0.1371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9903 -0.0104 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.9903 -0.0104 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.2586 0.4542 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.2586 0.4542 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.7428 0.3068 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.7428 0.3068 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.8593 0.8188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8593 0.8188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8845 0.8356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8845 0.8356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5203 0.7661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5203 0.7661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5400 0.1369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5400 0.1369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1442 -0.4673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1442 -0.4673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1439 -1.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1439 -1.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4764 -1.7471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4764 -1.7471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5707 -1.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5707 -1.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2612 -1.8094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2612 -1.8094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5554 -1.8097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5554 -1.8097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2505 -2.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2505 -2.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6412 -2.3378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6412 -2.3378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3367 -1.8102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3367 -1.8102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6416 -1.2823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6416 -1.2823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2510 -1.2821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2510 -1.2821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2721 -1.8104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2721 -1.8104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3365 -2.8652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3365 -2.8652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7706 -2.9519 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -1.7706 -2.9519 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.4124 -3.4247 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.4124 -3.4247 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.8966 -3.2241 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.8966 -3.2241 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.3989 -3.2188 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.3989 -3.2188 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.7606 -2.8570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7606 -2.8570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2874 -3.0462 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.2874 -3.0462 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.2311 -2.9115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2311 -2.9115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7505 -3.8698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7505 -3.8698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6010 -3.7203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6010 -3.7203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7113 3.7203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7113 3.7203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1526 4.6176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1526 4.6176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5597 -2.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5597 -2.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5457 -2.1475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5457 -2.1475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 38 43 1 0 0 0 0 | + | 38 43 1 0 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 47 46 1 1 0 0 0 | + | 47 46 1 1 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | 47 43 1 0 0 0 0 | + | 47 43 1 0 0 0 0 |
| − | 15 53 1 0 0 0 0 | + | 15 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 49 55 1 0 0 0 0 | + | 49 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 55 56 | + | M SAL 2 2 55 56 |
| − | M SBL 2 1 60 | + | M SBL 2 1 60 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 60 -1.5597 -2.3142 | + | M SVB 2 60 -1.5597 -2.3142 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 58 0.7113 3.7203 | + | M SVB 1 58 0.7113 3.7203 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AADGL0015 | + | ID FL7AADGL0015 |
| − | KNApSAcK_ID C00006863 | + | KNApSAcK_ID C00006863 |
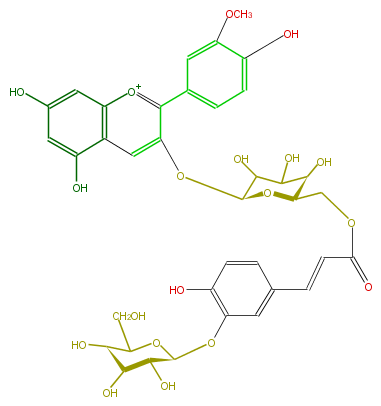
| − | NAME Penidin 3-[6-(3-glucosylcaffeyl)glucoside] | + | NAME Penidin 3-[6-(3-glucosylcaffeyl)glucoside] |
| − | CAS_RN 175737-95-8 | + | CAS_RN 175737-95-8 |
| − | FORMULA C37H39O19 | + | FORMULA C37H39O19 |
| − | EXACTMASS 787.208554066 | + | EXACTMASS 787.208554066 |
| − | AVERAGEMASS 787.69416 | + | AVERAGEMASS 787.69416 |
| − | SMILES c(c(O)6)c(c(c(c6)O)4)[o+1]c(c(c5)cc(c(c5)O)OC)c(c4)O[C@H](O1)C(O)C([C@H]([C@H]1COC(C=Cc(c2)ccc(O)c2O[C@H](O3)[C@H]([C@@H](O)[C@@H](O)C3CO)O)=O)O)O | + | SMILES c(c(O)6)c(c(c(c6)O)4)[o+1]c(c(c5)cc(c(c5)O)OC)c(c4)O[C@H](O1)C(O)C([C@H]([C@H]1COC(C=Cc(c2)ccc(O)c2O[C@H](O3)[C@H]([C@@H](O)[C@@H](O)C3CO)O)=O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
-2.9203 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9203 1.1987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3640 0.8775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8077 1.1987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8077 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3640 2.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2514 0.8775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6951 1.1987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6951 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2514 2.1622 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-0.1390 2.1621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4279 1.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9949 2.1621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9949 2.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4279 3.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1390 2.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4764 2.1621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8609 3.3168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3640 0.2354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2790 0.4780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2238 0.4542 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.9556 -0.0104 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.4714 0.1371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9903 -0.0104 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.2586 0.4542 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.7428 0.3068 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.8593 0.8188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8845 0.8356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5203 0.7661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5400 0.1369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1442 -0.4673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1439 -1.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4764 -1.7471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5707 -1.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2612 -1.8094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5554 -1.8097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2505 -2.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6412 -2.3378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3367 -1.8102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6416 -1.2823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2510 -1.2821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2721 -1.8104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3365 -2.8652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7706 -2.9519 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.4124 -3.4247 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.8966 -3.2241 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.3989 -3.2188 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.7606 -2.8570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2874 -3.0462 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.2311 -2.9115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7505 -3.8698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6010 -3.7203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7113 3.7203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1526 4.6176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5597 -2.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5457 -2.1475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
24 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
38 43 1 0 0 0 0
44 45 1 1 0 0 0
45 46 1 1 0 0 0
47 46 1 1 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
49 44 1 0 0 0 0
44 50 1 0 0 0 0
45 51 1 0 0 0 0
46 52 1 0 0 0 0
47 43 1 0 0 0 0
15 53 1 0 0 0 0
53 54 1 0 0 0 0
49 55 1 0 0 0 0
55 56 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 55 56
M SBL 2 1 60
M SMT 2 CH2OH
M SVB 2 60 -1.5597 -2.3142
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 58
M SMT 1 OCH3
M SVB 1 58 0.7113 3.7203
S SKP 8
ID FL7AADGL0015
KNApSAcK_ID C00006863
NAME Penidin 3-[6-(3-glucosylcaffeyl)glucoside]
CAS_RN 175737-95-8
FORMULA C37H39O19
EXACTMASS 787.208554066
AVERAGEMASS 787.69416
SMILES c(c(O)6)c(c(c(c6)O)4)[o+1]c(c(c5)cc(c(c5)O)OC)c(c4)O[C@H](O1)C(O)C([C@H]([C@H]1COC(C=Cc(c2)ccc(O)c2O[C@H](O3)[C@H]([C@@H](O)[C@@H](O)C3CO)O)=O)O)O
M END