Mol:FL7AACGL0105
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.4273 2.9201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4273 2.9201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4273 2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4273 2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7129 1.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7129 1.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9984 2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9984 2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9984 2.9201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9984 2.9201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7129 3.3326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7129 3.3326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2839 1.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2839 1.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5695 2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5695 2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5695 2.9201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5695 2.9201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2839 3.3326 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.2839 3.3326 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2071 3.3684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2071 3.3684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9215 2.9559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9215 2.9559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6360 3.3684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6360 3.3684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6360 4.1934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6360 4.1934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9215 4.6059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9215 4.6059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2071 4.1934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2071 4.1934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2762 4.5630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2762 4.5630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0747 1.7232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0747 1.7232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0073 3.2549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0073 3.2549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7129 1.1202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7129 1.1202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2961 2.9874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2961 2.9874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4933 -0.4400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4933 -0.4400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6930 -0.6413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6930 -0.6413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2873 0.0774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2873 0.0774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4279 0.4891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4279 0.4891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3724 0.6905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3724 0.6905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7781 -0.0282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7781 -0.0282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3080 0.2670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3080 0.2670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8409 0.1153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8409 0.1153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4933 -1.2103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4933 -1.2103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1459 -0.8740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1459 -0.8740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9408 -0.2718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9408 -0.2718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5335 -0.9893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5335 -0.9893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9512 -1.7007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9512 -1.7007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7762 -1.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7762 -1.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1835 -0.9772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1835 -0.9772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7657 -0.2658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7657 -0.2658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1725 0.4507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1725 0.4507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0073 -0.9712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0073 -0.9712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1933 -2.4052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1933 -2.4052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7328 -1.1834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7328 -1.1834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5006 -1.9739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5006 -1.9739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1643 -0.5870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1643 -0.5870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0427 -2.4417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0427 -2.4417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7821 -2.4565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7821 -2.4565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1818 -3.1782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1818 -3.1782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7566 -3.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7566 -3.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0683 -3.8704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0683 -3.8704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4680 -3.1487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4680 -3.1487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2917 -3.1339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2917 -3.1339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4930 -4.5764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4930 -4.5764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1557 -4.6059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1557 -4.6059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2068 -1.7504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2068 -1.7504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0305 -1.7652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0305 -1.7652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 47 52 1 0 0 0 0 | + | 47 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 53 54 2 0 0 0 0 | + | 53 54 2 0 0 0 0 |
| − | 43 24 1 0 0 0 0 | + | 43 24 1 0 0 0 0 |
| − | 31 53 1 0 0 0 0 | + | 31 53 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0105 | + | ID FL7AACGL0105 |
| − | KNApSAcK_ID C00014761 | + | KNApSAcK_ID C00014761 |
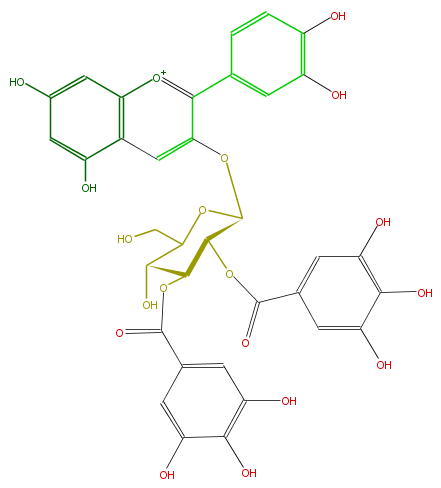
| − | NAME Cyanidin 3-(2,3-di-galloylglucoside) | + | NAME Cyanidin 3-(2,3-di-galloylglucoside) |
| − | CAS_RN 259254-15-4 | + | CAS_RN 259254-15-4 |
| − | FORMULA C35H29O19 | + | FORMULA C35H29O19 |
| − | EXACTMASS 753.130303746 | + | EXACTMASS 753.130303746 |
| − | AVERAGEMASS 753.5933600000001 | + | AVERAGEMASS 753.5933600000001 |
| − | SMILES c(c(O)6)c(c(c(O)c6)5)[o+1]c(c(c5)OC(C2OC(=O)c(c4)cc(c(O)c(O)4)O)OC(CO)C(C2OC(c(c3)cc(c(O)c3O)O)=O)O)c(c1)ccc(c(O)1)O | + | SMILES c(c(O)6)c(c(c(O)c6)5)[o+1]c(c(c5)OC(C2OC(=O)c(c4)cc(c(O)c(O)4)O)OC(CO)C(C2OC(c(c3)cc(c(O)c3O)O)=O)O)c(c1)ccc(c(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-3.4273 2.9201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4273 2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7129 1.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9984 2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9984 2.9201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7129 3.3326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2839 1.6826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5695 2.0951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5695 2.9201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2839 3.3326 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.2071 3.3684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9215 2.9559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6360 3.3684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6360 4.1934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9215 4.6059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2071 4.1934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2762 4.5630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0747 1.7232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0073 3.2549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7129 1.1202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2961 2.9874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4933 -0.4400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6930 -0.6413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2873 0.0774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4279 0.4891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3724 0.6905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7781 -0.0282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3080 0.2670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8409 0.1153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4933 -1.2103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1459 -0.8740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9408 -0.2718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5335 -0.9893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9512 -1.7007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7762 -1.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1835 -0.9772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7657 -0.2658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1725 0.4507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0073 -0.9712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1933 -2.4052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7328 -1.1834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5006 -1.9739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1643 -0.5870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0427 -2.4417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7821 -2.4565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1818 -3.1782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7566 -3.8852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0683 -3.8704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4680 -3.1487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2917 -3.1339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4930 -4.5764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1557 -4.6059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2068 -1.7504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0305 -1.7652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
8 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
13 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
25 18 1 0 0 0 0
32 33 2 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
36 39 1 0 0 0 0
35 40 1 0 0 0 0
33 41 1 0 0 0 0
41 42 2 0 0 0 0
41 43 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 44 1 0 0 0 0
49 50 1 0 0 0 0
48 51 1 0 0 0 0
47 52 1 0 0 0 0
45 53 1 0 0 0 0
53 54 2 0 0 0 0
43 24 1 0 0 0 0
31 53 1 0 0 0 0
S SKP 8
ID FL7AACGL0105
KNApSAcK_ID C00014761
NAME Cyanidin 3-(2,3-di-galloylglucoside)
CAS_RN 259254-15-4
FORMULA C35H29O19
EXACTMASS 753.130303746
AVERAGEMASS 753.5933600000001
SMILES c(c(O)6)c(c(c(O)c6)5)[o+1]c(c(c5)OC(C2OC(=O)c(c4)cc(c(O)c(O)4)O)OC(CO)C(C2OC(c(c3)cc(c(O)c3O)O)=O)O)c(c1)ccc(c(O)1)O
M END