Mol:FL7AACGL0067
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 66 72 0 0 0 0 0 0 0 0999 V2000 | + | 66 72 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8315 0.6153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8315 0.6153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8315 -0.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8315 -0.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2752 -0.3482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2752 -0.3482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7189 -0.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7189 -0.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7189 0.6153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7189 0.6153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2752 0.9365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2752 0.9365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1626 -0.3482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1626 -0.3482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6063 -0.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6063 -0.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6063 0.6153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6063 0.6153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1626 0.9365 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.1626 0.9365 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0502 0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0502 0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5168 0.6091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5168 0.6091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0838 0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0838 0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0838 1.5911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0838 1.5911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5168 1.9184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5168 1.9184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0502 1.5911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0502 1.5911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3876 0.9364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3876 0.9364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6506 1.9183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6506 1.9183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2752 -0.9903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2752 -0.9903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1902 -0.7477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1902 -0.7477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9090 -0.4511 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.9090 -0.4511 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.3107 -0.9814 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.3107 -0.9814 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.8891 -0.7564 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.8891 -0.7564 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.4473 -0.7504 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.4473 -0.7504 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.0417 -0.3447 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.0417 -0.3447 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.5356 -0.6118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5356 -0.6118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6844 -1.0118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6844 -1.0118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2206 -1.3128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2206 -1.3128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4385 -0.8825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4385 -0.8825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5168 2.5729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5168 2.5729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4339 -1.2645 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.4339 -1.2645 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -4.0627 -1.7545 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -4.0627 -1.7545 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.5282 -1.5466 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.5282 -1.5466 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.0125 -1.5410 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.0125 -1.5410 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.3873 -1.1662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3873 -1.1662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8549 -1.4130 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.8549 -1.4130 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.9477 -0.9678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9477 -0.9678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4132 -2.2157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4132 -2.2157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2220 -2.0607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2220 -2.0607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2255 -0.8378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2255 -0.8378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8867 -0.4341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8867 -0.4341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0417 -0.3447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0417 -0.3447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8583 0.2973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8583 0.2973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3940 0.5741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3940 0.5741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9944 0.2274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9944 0.2274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3940 1.2138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3940 1.2138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8259 1.4631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8259 1.4631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8259 2.1429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8259 2.1429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3869 2.4668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3869 2.4668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3869 3.1147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3869 3.1147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8259 3.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8259 3.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2648 3.1147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2648 3.1147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2648 2.4668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2648 2.4668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8259 4.0862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8259 4.0862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0154 -3.1973 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.0154 -3.1973 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 0.5975 -3.0471 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.5975 -3.0471 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.5698 -2.4524 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.5698 -2.4524 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.8604 -1.9392 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.8604 -1.9392 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.2220 -2.1645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2220 -2.1645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2783 -2.7842 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.2783 -2.7842 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.0154 -3.6701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0154 -3.6701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1031 -2.4451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1031 -2.4451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9478 3.4385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9478 3.4385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3191 -4.0862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3191 -4.0862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3293 -2.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3293 -2.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2296 -2.8066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2296 -2.8066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 22 27 1 0 0 0 0 | + | 22 27 1 0 0 0 0 |
| − | 23 28 1 0 0 0 0 | + | 23 28 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 15 30 1 0 0 0 0 | + | 15 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 19 1 0 0 0 0 | + | 34 19 1 0 0 0 0 |
| − | 25 42 1 0 0 0 0 | + | 25 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 44 43 1 0 0 0 0 | + | 44 43 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 53 48 1 0 0 0 0 | + | 53 48 1 0 0 0 0 |
| − | 51 54 1 0 0 0 0 | + | 51 54 1 0 0 0 0 |
| − | 55 56 1 1 0 0 0 | + | 55 56 1 1 0 0 0 |
| − | 56 57 1 1 0 0 0 | + | 56 57 1 1 0 0 0 |
| − | 58 57 1 1 0 0 0 | + | 58 57 1 1 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 55 1 0 0 0 0 | + | 60 55 1 0 0 0 0 |
| − | 55 61 1 0 0 0 0 | + | 55 61 1 0 0 0 0 |
| − | 57 62 1 0 0 0 0 | + | 57 62 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 27 58 1 0 0 0 0 | + | 27 58 1 0 0 0 0 |
| − | 50 63 1 0 0 0 0 | + | 50 63 1 0 0 0 0 |
| − | 56 64 1 0 0 0 0 | + | 56 64 1 0 0 0 0 |
| − | 60 65 1 0 0 0 0 | + | 60 65 1 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 65 66 | + | M SAL 2 2 65 66 |
| − | M SBL 2 1 71 | + | M SBL 2 1 71 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 71 -0.3293 -2.3713 | + | M SVB 2 71 -0.3293 -2.3713 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 42 | + | M SBL 1 1 42 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 42 -4.3198 -0.9109 | + | M SVB 1 42 -4.3198 -0.9109 |
| − | S SKP 8 | + | S SKP 8 |
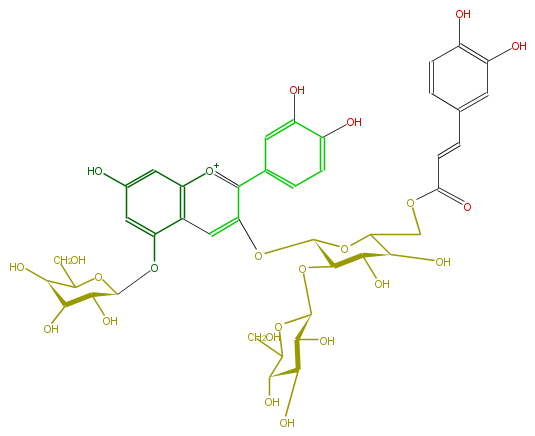
| − | ID FL7AACGL0067 | + | ID FL7AACGL0067 |
| − | KNApSAcK_ID C00006843 | + | KNApSAcK_ID C00006843 |
| − | NAME Cyanidin 3-(6''-caffeylsophoroside)-5-glucoside | + | NAME Cyanidin 3-(6''-caffeylsophoroside)-5-glucoside |
| − | CAS_RN 36618-57-2,164864-59-9,81360-21-6 | + | CAS_RN 36618-57-2,164864-59-9,81360-21-6 |
| − | FORMULA C42H47O24 | + | FORMULA C42H47O24 |
| − | EXACTMASS 935.245727432 | + | EXACTMASS 935.245727432 |
| − | AVERAGEMASS 935.80818 | + | AVERAGEMASS 935.80818 |
| − | SMILES [C@@H]([C@@H]7O)(O)C(O[C@H]([C@@H](O)7)Oc(c6)c(c5cc6O)cc(c([o+1]5)c(c4)ccc(c4O)O)O[C@H]([C@@H]2O[C@H](O3)[C@H]([C@H]([C@H](C3CO)O)O)O)OC([C@@H](O)[C@H]2O)COC(C=Cc(c1)cc(c(c1)O)O)=O)CO | + | SMILES [C@@H]([C@@H]7O)(O)C(O[C@H]([C@@H](O)7)Oc(c6)c(c5cc6O)cc(c([o+1]5)c(c4)ccc(c4O)O)O[C@H]([C@@H]2O[C@H](O3)[C@H]([C@H]([C@H](C3CO)O)O)O)OC([C@@H](O)[C@H]2O)COC(C=Cc(c1)cc(c(c1)O)O)=O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
66 72 0 0 0 0 0 0 0 0999 V2000
-2.8315 0.6153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8315 -0.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2752 -0.3482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7189 -0.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7189 0.6153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2752 0.9365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1626 -0.3482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6063 -0.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6063 0.6153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1626 0.9365 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-0.0502 0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5168 0.6091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0838 0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0838 1.5911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5168 1.9184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0502 1.5911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3876 0.9364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6506 1.9183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2752 -0.9903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1902 -0.7477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9090 -0.4511 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.3107 -0.9814 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.8891 -0.7564 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.4473 -0.7504 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.0417 -0.3447 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.5356 -0.6118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6844 -1.0118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2206 -1.3128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4385 -0.8825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5168 2.5729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4339 -1.2645 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-4.0627 -1.7545 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.5282 -1.5466 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.0125 -1.5410 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.3873 -1.1662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8549 -1.4130 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.9477 -0.9678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4132 -2.2157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2220 -2.0607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2255 -0.8378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8867 -0.4341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0417 -0.3447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8583 0.2973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3940 0.5741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9944 0.2274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3940 1.2138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8259 1.4631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8259 2.1429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3869 2.4668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3869 3.1147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8259 3.4386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2648 3.1147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2648 2.4668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8259 4.0862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0154 -3.1973 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
0.5975 -3.0471 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.5698 -2.4524 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.8604 -1.9392 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.2220 -2.1645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2783 -2.7842 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.0154 -3.6701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1031 -2.4451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9478 3.4385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3191 -4.0862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3293 -2.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2296 -2.8066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
22 27 1 0 0 0 0
23 28 1 0 0 0 0
24 29 1 0 0 0 0
15 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
36 40 1 0 0 0 0
40 41 1 0 0 0 0
34 19 1 0 0 0 0
25 42 1 0 0 0 0
42 43 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
44 43 1 0 0 0 0
48 49 2 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 52 1 0 0 0 0
52 53 2 0 0 0 0
53 48 1 0 0 0 0
51 54 1 0 0 0 0
55 56 1 1 0 0 0
56 57 1 1 0 0 0
58 57 1 1 0 0 0
58 59 1 0 0 0 0
59 60 1 0 0 0 0
60 55 1 0 0 0 0
55 61 1 0 0 0 0
57 62 1 0 0 0 0
20 21 1 0 0 0 0
27 58 1 0 0 0 0
50 63 1 0 0 0 0
56 64 1 0 0 0 0
60 65 1 0 0 0 0
65 66 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 65 66
M SBL 2 1 71
M SMT 2 CH2OH
M SVB 2 71 -0.3293 -2.3713
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 42
M SMT 1 CH2OH
M SVB 1 42 -4.3198 -0.9109
S SKP 8
ID FL7AACGL0067
KNApSAcK_ID C00006843
NAME Cyanidin 3-(6''-caffeylsophoroside)-5-glucoside
CAS_RN 36618-57-2,164864-59-9,81360-21-6
FORMULA C42H47O24
EXACTMASS 935.245727432
AVERAGEMASS 935.80818
SMILES [C@@H]([C@@H]7O)(O)C(O[C@H]([C@@H](O)7)Oc(c6)c(c5cc6O)cc(c([o+1]5)c(c4)ccc(c4O)O)O[C@H]([C@@H]2O[C@H](O3)[C@H]([C@H]([C@H](C3CO)O)O)O)OC([C@@H](O)[C@H]2O)COC(C=Cc(c1)cc(c(c1)O)O)=O)CO
M END