Mol:FL7AACGL0054
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 61 66 0 0 0 0 0 0 0 0999 V2000 | + | 61 66 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3429 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3429 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3429 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3429 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7866 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7866 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2303 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2303 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2303 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2303 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7866 1.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7866 1.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3260 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3260 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8823 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8823 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8823 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8823 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3260 1.1416 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.3260 1.1416 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4384 1.1415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4384 1.1415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0053 0.8141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0053 0.8141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5723 1.1415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5723 1.1415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5723 1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5723 1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0053 2.1235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0053 2.1235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4384 1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4384 1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8990 1.1415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8990 1.1415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1391 2.1234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1391 2.1234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7866 -0.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7866 -0.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2984 -0.5427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2984 -0.5427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0053 2.7780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0053 2.7780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9454 -1.0595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9454 -1.0595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5742 -1.5494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5742 -1.5494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0397 -1.3416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0397 -1.3416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5240 -1.3360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5240 -1.3360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8988 -0.9611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8988 -0.9611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3664 -1.2079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3664 -1.2079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4592 -1.3561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4592 -1.3561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1529 -1.5776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1529 -1.5776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7335 -1.8557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7335 -1.8557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8312 -0.7059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8312 -0.7059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5589 -0.2013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5589 -0.2013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2581 -0.7224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2581 -0.7224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8366 -0.5570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8366 -0.5570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4185 -0.7224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4185 -0.7224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7194 -0.2013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7194 -0.2013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1409 -0.3666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1409 -0.3666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0002 -0.3510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0002 -0.3510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3791 0.0459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3791 0.0459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4228 -0.3898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4228 -0.3898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9853 -0.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9853 -0.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8312 -0.0045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8312 -0.0045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3772 -1.7371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3772 -1.7371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1985 -2.5112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1985 -2.5112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4675 -2.6225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4675 -2.6225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0984 -2.0052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0984 -2.0052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4158 -2.0224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4158 -2.0224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0850 -1.4494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0850 -1.4494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4233 -1.4494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4233 -1.4494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0924 -2.0224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0924 -2.0224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4233 -2.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4233 -2.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0850 -2.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0850 -2.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4311 -2.0224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4311 -2.0224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7736 -2.7780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7736 -2.7780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9140 -0.7983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9140 -0.7983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3751 0.3095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3751 0.3095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8154 0.0553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8154 0.0553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2681 0.3166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2681 0.3166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7736 0.0247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7736 0.0247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3751 0.9365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3751 0.9365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2681 0.8996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2681 0.8996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 33 20 1 0 0 0 0 | + | 33 20 1 0 0 0 0 |
| − | 31 42 1 0 0 0 0 | + | 31 42 1 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 44 54 2 0 0 0 0 | + | 44 54 2 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 49 55 1 0 0 0 0 | + | 49 55 1 0 0 0 0 |
| − | 42 56 1 0 0 0 0 | + | 42 56 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 2 0 0 0 0 | + | 58 59 2 0 0 0 0 |
| − | 56 60 2 0 0 0 0 | + | 56 60 2 0 0 0 0 |
| − | 58 61 1 0 0 0 0 | + | 58 61 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0054 | + | ID FL7AACGL0054 |
| − | KNApSAcK_ID C00006825 | + | KNApSAcK_ID C00006825 |
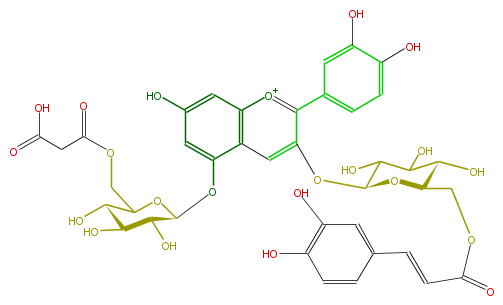
| − | NAME Cyanidin 3-(6''-caffeylglucoside)-5-(6'''-malonylglucoside) | + | NAME Cyanidin 3-(6''-caffeylglucoside)-5-(6'''-malonylglucoside) |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C39H39O22 | + | FORMULA C39H39O22 |
| − | EXACTMASS 859.193297932 | + | EXACTMASS 859.193297932 |
| − | AVERAGEMASS 859.7137600000001 | + | AVERAGEMASS 859.7137600000001 |
| − | SMILES O(C(O6)C(O)C(O)C(O)C6COC(CC(O)=O)=O)c(c1)c(c2)c([o+1]c(c(c5)cc(c(c5)O)O)c2OC(O3)C(C(C(C(COC(C=Cc(c4)ccc(O)c4O)=O)3)O)O)O)cc1O | + | SMILES O(C(O6)C(O)C(O)C(O)C6COC(CC(O)=O)=O)c(c1)c(c2)c([o+1]c(c(c5)cc(c(c5)O)O)c2OC(O3)C(C(C(C(COC(C=Cc(c4)ccc(O)c4O)=O)3)O)O)O)cc1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
61 66 0 0 0 0 0 0 0 0999 V2000
-1.3429 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3429 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7866 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2303 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2303 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7866 1.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3260 -0.1431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8823 0.1780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8823 0.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3260 1.1416 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.4384 1.1415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0053 0.8141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5723 1.1415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5723 1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0053 2.1235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4384 1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8990 1.1415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1391 2.1234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7866 -0.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2984 -0.5427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0053 2.7780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9454 -1.0595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5742 -1.5494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0397 -1.3416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5240 -1.3360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8988 -0.9611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3664 -1.2079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4592 -1.3561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1529 -1.5776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7335 -1.8557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8312 -0.7059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5589 -0.2013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2581 -0.7224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8366 -0.5570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4185 -0.7224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7194 -0.2013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1409 -0.3666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0002 -0.3510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3791 0.0459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4228 -0.3898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9853 -0.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8312 -0.0045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3772 -1.7371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1985 -2.5112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4675 -2.6225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0984 -2.0052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4158 -2.0224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0850 -1.4494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4233 -1.4494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0924 -2.0224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4233 -2.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0850 -2.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4311 -2.0224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7736 -2.7780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9140 -0.7983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3751 0.3095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8154 0.0553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2681 0.3166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7736 0.0247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3751 0.9365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2681 0.8996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
15 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
27 31 1 0 0 0 0
25 19 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
35 41 1 0 0 0 0
33 20 1 0 0 0 0
31 42 1 0 0 0 0
41 43 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
44 45 1 0 0 0 0
44 54 2 0 0 0 0
43 44 1 0 0 0 0
49 55 1 0 0 0 0
42 56 1 0 0 0 0
56 57 1 0 0 0 0
57 58 1 0 0 0 0
58 59 2 0 0 0 0
56 60 2 0 0 0 0
58 61 1 0 0 0 0
S SKP 8
ID FL7AACGL0054
KNApSAcK_ID C00006825
NAME Cyanidin 3-(6''-caffeylglucoside)-5-(6'''-malonylglucoside)
CAS_RN -
FORMULA C39H39O22
EXACTMASS 859.193297932
AVERAGEMASS 859.7137600000001
SMILES O(C(O6)C(O)C(O)C(O)C6COC(CC(O)=O)=O)c(c1)c(c2)c([o+1]c(c(c5)cc(c(c5)O)O)c2OC(O3)C(C(C(C(COC(C=Cc(c4)ccc(O)c4O)=O)3)O)O)O)cc1O
M END