Mol:FL7AACGL0034
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 49 53 0 0 0 0 0 0 0 0999 V2000 | + | 49 53 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.2641 1.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2641 1.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2641 0.8333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2641 0.8333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7078 0.5121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7078 0.5121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1515 0.8333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1515 0.8333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1515 1.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1515 1.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7078 1.7968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7078 1.7968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5952 0.5121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5952 0.5121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0389 0.8333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0389 0.8333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0389 1.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0389 1.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5952 1.7968 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.5952 1.7968 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4828 1.7967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4828 1.7967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0842 1.4694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0842 1.4694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6512 1.7967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6512 1.7967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6512 2.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6512 2.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0842 2.7787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0842 2.7787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4828 2.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4828 2.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8202 1.7967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8202 1.7967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2180 2.7786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2180 2.7786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7078 -0.1300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7078 -0.1300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6217 -1.4404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6217 -1.4404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9728 -1.9039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9728 -1.9039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4785 -1.7073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4785 -1.7073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9664 -1.6538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9664 -1.6538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6118 -1.3474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6118 -1.3474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1695 -1.5808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1695 -1.5808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4254 -1.9305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4254 -1.9305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7682 -2.1936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7682 -2.1936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4055 -1.4003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4055 -1.4003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0842 3.4332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0842 3.4332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0568 -0.8067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0568 -0.8067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6100 -0.4873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6100 -0.4873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4345 -1.1015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4345 -1.1015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6100 -1.7195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6100 -1.7195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0568 -2.0390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0568 -2.0390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2323 -1.4247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2323 -1.4247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2157 -0.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2157 -0.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6368 -1.9265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6368 -1.9265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1611 -2.4283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1611 -2.4283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6100 -2.8353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6100 -2.8353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6426 0.4370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6426 0.4370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3254 -3.2484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3254 -3.2484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8189 -2.9634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8189 -2.9634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8189 -2.3182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8189 -2.3182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4244 -3.3130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4244 -3.3130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8887 -3.0206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8887 -3.0206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9087 -2.3154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9087 -2.3154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3417 -3.4332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3417 -3.4332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1057 -1.0622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1057 -1.0622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8202 -1.4747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8202 -1.4747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 21 26 1 0 0 0 0 | + | 21 26 1 0 0 0 0 |
| − | 22 27 1 0 0 0 0 | + | 22 27 1 0 0 0 0 |
| − | 23 28 1 0 0 0 0 | + | 23 28 1 0 0 0 0 |
| − | 15 29 1 0 0 0 0 | + | 15 29 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 8 40 1 0 0 0 0 | + | 8 40 1 0 0 0 0 |
| − | 40 31 1 0 0 0 0 | + | 40 31 1 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 45 47 2 0 0 0 0 | + | 45 47 2 0 0 0 0 |
| − | 37 20 1 0 0 0 0 | + | 37 20 1 0 0 0 0 |
| − | 24 48 1 0 0 0 0 | + | 24 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 48 49 | + | M SAL 1 2 48 49 |
| − | M SBL 1 1 52 | + | M SBL 1 1 52 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 52 -6.8911 6.8552 | + | M SBV 1 52 -6.8911 6.8552 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0034 | + | ID FL7AACGL0034 |
| − | KNApSAcK_ID C00006804 | + | KNApSAcK_ID C00006804 |
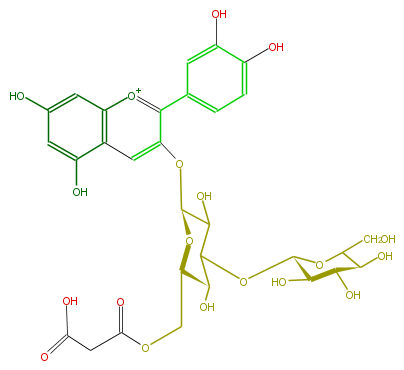
| − | NAME Cyanidin 3-(6''-malonyllaminaribioside) | + | NAME Cyanidin 3-(6''-malonyllaminaribioside) |
| − | CAS_RN 157201-93-9 | + | CAS_RN 157201-93-9 |
| − | FORMULA C30H33O19 | + | FORMULA C30H33O19 |
| − | EXACTMASS 697.161603874 | + | EXACTMASS 697.161603874 |
| − | AVERAGEMASS 697.57162 | + | AVERAGEMASS 697.57162 |
| − | SMILES OC(C5O)C(O)C(OC(CO)5)OC(C4O)C(O)C(OC4COC(=O)CC(O)=O)Oc(c(c(c3)cc(O)c(O)c3)1)cc(c2O)c(cc(c2)O)[o+1]1 | + | SMILES OC(C5O)C(O)C(OC(CO)5)OC(C4O)C(O)C(OC4COC(=O)CC(O)=O)Oc(c(c(c3)cc(O)c(O)c3)1)cc(c2O)c(cc(c2)O)[o+1]1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
49 53 0 0 0 0 0 0 0 0999 V2000
-3.2641 1.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2641 0.8333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7078 0.5121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1515 0.8333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1515 1.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7078 1.7968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5952 0.5121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0389 0.8333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0389 1.4756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5952 1.7968 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-0.4828 1.7967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0842 1.4694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6512 1.7967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6512 2.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0842 2.7787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4828 2.4514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8202 1.7967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2180 2.7786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7078 -0.1300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6217 -1.4404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9728 -1.9039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4785 -1.7073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9664 -1.6538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6118 -1.3474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1695 -1.5808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4254 -1.9305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7682 -2.1936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4055 -1.4003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0842 3.4332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0568 -0.8067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6100 -0.4873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4345 -1.1015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6100 -1.7195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0568 -2.0390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2323 -1.4247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2157 -0.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6368 -1.9265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1611 -2.4283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6100 -2.8353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6426 0.4370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3254 -3.2484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8189 -2.9634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8189 -2.3182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4244 -3.3130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8887 -3.0206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9087 -2.3154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3417 -3.4332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1057 -1.0622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8202 -1.4747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
21 26 1 0 0 0 0
22 27 1 0 0 0 0
23 28 1 0 0 0 0
15 29 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
35 37 1 0 0 0 0
34 38 1 0 0 0 0
33 39 1 0 0 0 0
33 32 1 1 0 0 0
8 40 1 0 0 0 0
40 31 1 0 0 0 0
39 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 2 0 0 0 0
42 44 1 0 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
45 47 2 0 0 0 0
37 20 1 0 0 0 0
24 48 1 0 0 0 0
48 49 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 48 49
M SBL 1 1 52
M SMT 1 CH2OH
M SBV 1 52 -6.8911 6.8552
S SKP 8
ID FL7AACGL0034
KNApSAcK_ID C00006804
NAME Cyanidin 3-(6''-malonyllaminaribioside)
CAS_RN 157201-93-9
FORMULA C30H33O19
EXACTMASS 697.161603874
AVERAGEMASS 697.57162
SMILES OC(C5O)C(O)C(OC(CO)5)OC(C4O)C(O)C(OC4COC(=O)CC(O)=O)Oc(c(c(c3)cc(O)c(O)c3)1)cc(c2O)c(cc(c2)O)[o+1]1
M END