Mol:FL7AACGA0007
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3116 -0.1182 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3116 -0.1182 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3116 -0.7606 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3116 -0.7606 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7553 -1.0818 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7553 -1.0818 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1990 -0.7606 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1990 -0.7606 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1990 -0.1182 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1990 -0.1182 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7553 0.2030 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7553 0.2030 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3573 -1.0818 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3573 -1.0818 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9136 -0.7606 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9136 -0.7606 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9136 -0.1182 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9136 -0.1182 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3573 0.2030 0.1672 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.3573 0.2030 0.1672 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4697 0.2028 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4697 0.2028 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0367 -0.1245 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0367 -0.1245 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6037 0.2028 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6037 0.2028 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6037 0.8575 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6037 0.8575 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0367 1.1849 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0367 1.1849 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4697 0.8575 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4697 0.8575 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8677 0.2028 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8677 0.2028 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1705 1.1848 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1705 1.1848 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7553 -1.7239 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7553 -1.7239 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3297 -1.4813 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3297 -1.4813 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0367 1.8394 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0367 1.8394 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9140 -1.9981 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9140 -1.9981 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5429 -2.4880 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5429 -2.4880 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0084 -2.2802 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0084 -2.2802 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4926 -2.2746 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4926 -2.2746 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8674 -1.8997 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8674 -1.8997 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3350 -2.1465 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3350 -2.1465 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4279 -2.2947 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4279 -2.2947 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1215 -2.5162 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1215 -2.5162 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7021 -2.7943 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7021 -2.7943 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7999 -1.6445 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7999 -1.6445 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7999 -1.1082 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7999 -1.1082 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5649 -0.7154 0.1543 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5649 -0.7154 0.1543 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1402 -1.0648 -0.0120 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1402 -1.0648 -0.0120 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5651 -0.0758 0.1447 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5651 -0.0758 0.1447 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9791 0.1715 0.0177 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9791 0.1715 0.0177 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9793 0.8512 0.0075 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9793 0.8512 0.0075 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5171 1.1725 -0.1575 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5171 1.1725 -0.1575 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5173 1.8203 -0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5173 1.8203 -0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9798 2.1467 -0.0118 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9798 2.1467 -0.0118 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4420 1.8254 0.1532 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4420 1.8254 0.1532 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4418 1.1777 0.1629 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4418 1.1777 0.1629 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9800 2.7943 -0.0215 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9800 2.7943 -0.0215 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5903 -1.1400 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5903 -1.1400 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2895 -1.6610 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2895 -1.6610 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8679 -1.4957 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8679 -1.4957 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4499 -1.6610 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4499 -1.6610 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7508 -1.1400 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7508 -1.1400 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1723 -1.3052 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1723 -1.3052 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0316 -1.2897 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0316 -1.2897 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4105 -0.8927 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4105 -0.8927 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7508 -0.3804 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7508 -0.3804 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8029 -1.6343 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8029 -1.6343 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5173 -2.0468 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5173 -2.0468 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 49 51 1 0 0 0 0 | + | 49 51 1 0 0 0 0 |
| − | 48 52 1 0 0 0 0 | + | 48 52 1 0 0 0 0 |
| − | 45 20 1 0 0 0 0 | + | 45 20 1 0 0 0 0 |
| − | 47 53 1 0 0 0 0 | + | 47 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 58 -7.6576 5.8485 | + | M SBV 1 58 -7.6576 5.8485 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGA0007 | + | ID FL7AACGA0007 |
| − | KNApSAcK_ID C00006813 | + | KNApSAcK_ID C00006813 |
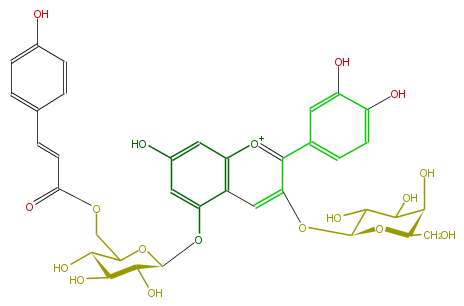
| − | NAME Cyanidin 3-galactoside-5-(6-p-coumarylglucoside) | + | NAME Cyanidin 3-galactoside-5-(6-p-coumarylglucoside) |
| − | CAS_RN 151676-99-2 | + | CAS_RN 151676-99-2 |
| − | FORMULA C36H37O18 | + | FORMULA C36H37O18 |
| − | EXACTMASS 757.1979893800001 | + | EXACTMASS 757.1979893800001 |
| − | AVERAGEMASS 757.66818 | + | AVERAGEMASS 757.66818 |
| − | SMILES C(C(C6O)OC(C(C(O)6)O)Oc(c2)c(c4)c([o+1]c(c(OC(O5)C(C(O)C(C(CO)5)O)O)4)c(c3)ccc(O)c(O)3)cc2O)OC(C=Cc(c1)ccc(c1)O)=O | + | SMILES C(C(C6O)OC(C(C(O)6)O)Oc(c2)c(c4)c([o+1]c(c(OC(O5)C(C(O)C(C(CO)5)O)O)4)c(c3)ccc(O)c(O)3)cc2O)OC(C=Cc(c1)ccc(c1)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-1.3116 -0.1182 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3116 -0.7606 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7553 -1.0818 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1990 -0.7606 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1990 -0.1182 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7553 0.2030 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3573 -1.0818 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9136 -0.7606 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9136 -0.1182 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3573 0.2030 0.1672 O 0 3 0 0 0 0 0 0 0 0 0 0
1.4697 0.2028 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0367 -0.1245 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6037 0.2028 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6037 0.8575 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0367 1.1849 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4697 0.8575 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8677 0.2028 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1705 1.1848 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7553 -1.7239 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3297 -1.4813 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0367 1.8394 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9140 -1.9981 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5429 -2.4880 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0084 -2.2802 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4926 -2.2746 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8674 -1.8997 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3350 -2.1465 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4279 -2.2947 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1215 -2.5162 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7021 -2.7943 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7999 -1.6445 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7999 -1.1082 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5649 -0.7154 0.1543 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1402 -1.0648 -0.0120 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5651 -0.0758 0.1447 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9791 0.1715 0.0177 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9793 0.8512 0.0075 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5171 1.1725 -0.1575 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5173 1.8203 -0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9798 2.1467 -0.0118 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4420 1.8254 0.1532 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4418 1.1777 0.1629 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9800 2.7943 -0.0215 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5903 -1.1400 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2895 -1.6610 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8679 -1.4957 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4499 -1.6610 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7508 -1.1400 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1723 -1.3052 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0316 -1.2897 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4105 -0.8927 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7508 -0.3804 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8029 -1.6343 0.1672 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5173 -2.0468 0.1672 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
15 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
27 31 1 0 0 0 0
31 32 1 0 0 0 0
25 19 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
32 33 1 0 0 0 0
44 45 1 0 0 0 0
45 46 1 1 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
48 49 1 0 0 0 0
49 44 1 0 0 0 0
44 50 1 0 0 0 0
49 51 1 0 0 0 0
48 52 1 0 0 0 0
45 20 1 0 0 0 0
47 53 1 0 0 0 0
53 54 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 58
M SMT 1 CH2OH
M SBV 1 58 -7.6576 5.8485
S SKP 8
ID FL7AACGA0007
KNApSAcK_ID C00006813
NAME Cyanidin 3-galactoside-5-(6-p-coumarylglucoside)
CAS_RN 151676-99-2
FORMULA C36H37O18
EXACTMASS 757.1979893800001
AVERAGEMASS 757.66818
SMILES C(C(C6O)OC(C(C(O)6)O)Oc(c2)c(c4)c([o+1]c(c(OC(O5)C(C(O)C(C(CO)5)O)O)4)c(c3)ccc(O)c(O)3)cc2O)OC(C=Cc(c1)ccc(c1)O)=O
M END