Mol:FL7AAAGL0051
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 88 96 0 0 0 0 0 0 0 0999 V2000 | + | 88 96 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.0666 2.5038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0666 2.5038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7259 2.8844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7259 2.8844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7259 3.6458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7259 3.6458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0666 4.0264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0666 4.0264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5928 3.6458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5928 3.6458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5928 2.8844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5928 2.8844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2877 3.9701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2877 3.9701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2850 2.4848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2850 2.4848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9327 2.8587 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.9327 2.8587 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5803 2.4848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5803 2.4848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5803 1.7369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5803 1.7369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9327 1.3630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9327 1.3630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2850 1.7369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2850 1.7369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2280 2.8587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2280 2.8587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8757 2.4848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8757 2.4848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8757 1.7369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8757 1.7369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2280 1.3630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2280 1.3630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3581 2.7633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3581 2.7633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2280 0.7371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2280 0.7371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8874 0.0528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8874 0.0528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2527 -0.4743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2527 -0.4743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5766 -0.0014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5766 -0.0014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7540 0.0610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7540 0.0610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3886 0.5882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3886 0.5882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0648 0.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0648 0.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2218 0.7014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2218 0.7014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7223 0.9904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7223 0.9904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2889 -0.3487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2889 -0.3487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7617 -0.3379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7617 -0.3379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5766 -0.5987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5766 -0.5987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6834 1.3896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6834 1.3896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0054 -1.1765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0054 -1.1765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2132 -0.9461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2132 -0.9461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2262 -0.1212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2262 -0.1212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8169 0.5952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8169 0.5952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6092 0.3648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6092 0.3648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5963 -0.4601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5963 -0.4601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6073 0.2361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6073 0.2361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1774 0.5374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1774 0.5374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5899 -0.1770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5899 -0.1770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3880 0.0320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3880 0.0320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1812 -0.1948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1812 -0.1948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7688 0.5198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7688 0.5198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9707 0.3108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9707 0.3108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1027 -0.0465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1027 -0.0465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5227 -0.4705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5227 -0.4705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6759 -0.0622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6759 -0.0622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3980 0.1565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3980 0.1565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9845 0.4952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9845 0.4952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7468 -1.6244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7468 -1.6244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4027 -1.2743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4027 -1.2743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3429 -0.4601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3429 -0.4601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6909 -1.0628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6909 -1.0628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3835 -1.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3835 -1.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3835 -0.4652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3835 -0.4652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8886 -1.3544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8886 -1.3544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8886 -2.0673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8886 -2.0673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5124 -2.4274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5124 -2.4274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2352 -2.2338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2352 -2.2338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7642 -2.7629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7642 -2.7629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5706 -3.4856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5706 -3.4856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8479 -3.6792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8479 -3.6792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3188 -3.1502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3188 -3.1502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8479 -4.2777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8479 -4.2777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9203 -3.8353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9203 -3.8353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5830 0.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5830 0.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2370 -0.4496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2370 -0.4496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3067 0.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3067 0.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5042 1.0808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5042 1.0808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0050 1.3699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0050 1.3699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6548 1.1958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6548 1.1958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1305 1.6715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1305 1.6715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9564 2.3213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9564 2.3213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3066 2.4954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3066 2.4954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8309 2.0197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8309 2.0197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2889 2.6538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2889 2.6538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1618 3.0355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1618 3.0355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2234 3.7054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2234 3.7054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6359 2.9910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6359 2.9910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4340 3.2000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4340 3.2000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2272 2.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2272 2.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8148 3.6878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8148 3.6878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0167 3.4788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0167 3.4788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5711 2.6883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5711 2.6883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1187 3.1296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1187 3.1296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7282 3.4195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7282 3.4195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8782 3.9955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8782 3.9955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1604 4.2777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1604 4.2777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 6 8 1 0 0 0 0 | + | 6 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 8 1 0 0 0 0 | + | 13 8 1 0 0 0 0 |
| − | 10 14 1 0 0 0 0 | + | 10 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 11 1 0 0 0 0 | + | 17 11 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 17 19 1 0 0 0 0 | + | 17 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 19 23 1 0 0 0 0 | + | 19 23 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 20 28 1 0 0 0 0 | + | 20 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 13 31 1 0 0 0 0 | + | 13 31 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 31 35 1 0 0 0 0 | + | 31 35 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 39 40 1 1 0 0 0 | + | 39 40 1 1 0 0 0 |
| − | 40 41 1 1 0 0 0 | + | 40 41 1 1 0 0 0 |
| − | 42 41 1 1 0 0 0 | + | 42 41 1 1 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 39 1 0 0 0 0 | + | 44 39 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 40 45 1 0 0 0 0 | + | 40 45 1 0 0 0 0 |
| − | 41 46 1 0 0 0 0 | + | 41 46 1 0 0 0 0 |
| − | 42 47 1 0 0 0 0 | + | 42 47 1 0 0 0 0 |
| − | 43 48 1 0 0 0 0 | + | 43 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 32 50 1 0 0 0 0 | + | 32 50 1 0 0 0 0 |
| − | 33 51 1 0 0 0 0 | + | 33 51 1 0 0 0 0 |
| − | 37 52 1 0 0 0 0 | + | 37 52 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 54 56 1 0 0 0 0 | + | 54 56 1 0 0 0 0 |
| − | 56 57 2 0 0 0 0 | + | 56 57 2 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 2 0 0 0 0 | + | 58 59 2 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 61 2 0 0 0 0 | + | 60 61 2 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 62 63 2 0 0 0 0 | + | 62 63 2 0 0 0 0 |
| − | 63 58 1 0 0 0 0 | + | 63 58 1 0 0 0 0 |
| − | 62 64 1 0 0 0 0 | + | 62 64 1 0 0 0 0 |
| − | 61 65 1 0 0 0 0 | + | 61 65 1 0 0 0 0 |
| − | 49 66 1 0 0 0 0 | + | 49 66 1 0 0 0 0 |
| − | 66 67 2 0 0 0 0 | + | 66 67 2 0 0 0 0 |
| − | 66 68 1 0 0 0 0 | + | 66 68 1 0 0 0 0 |
| − | 68 69 2 0 0 0 0 | + | 68 69 2 0 0 0 0 |
| − | 69 70 1 0 0 0 0 | + | 69 70 1 0 0 0 0 |
| − | 70 71 2 0 0 0 0 | + | 70 71 2 0 0 0 0 |
| − | 71 72 1 0 0 0 0 | + | 71 72 1 0 0 0 0 |
| − | 72 73 2 0 0 0 0 | + | 72 73 2 0 0 0 0 |
| − | 73 74 1 0 0 0 0 | + | 73 74 1 0 0 0 0 |
| − | 74 75 2 0 0 0 0 | + | 74 75 2 0 0 0 0 |
| − | 75 70 1 0 0 0 0 | + | 75 70 1 0 0 0 0 |
| − | 73 76 1 0 0 0 0 | + | 73 76 1 0 0 0 0 |
| − | 74 77 1 0 0 0 0 | + | 74 77 1 0 0 0 0 |
| − | 78 79 1 1 0 0 0 | + | 78 79 1 1 0 0 0 |
| − | 79 80 1 1 0 0 0 | + | 79 80 1 1 0 0 0 |
| − | 81 80 1 1 0 0 0 | + | 81 80 1 1 0 0 0 |
| − | 81 82 1 0 0 0 0 | + | 81 82 1 0 0 0 0 |
| − | 82 83 1 0 0 0 0 | + | 82 83 1 0 0 0 0 |
| − | 83 78 1 0 0 0 0 | + | 83 78 1 0 0 0 0 |
| − | 77 81 1 0 0 0 0 | + | 77 81 1 0 0 0 0 |
| − | 80 84 1 0 0 0 0 | + | 80 84 1 0 0 0 0 |
| − | 79 85 1 0 0 0 0 | + | 79 85 1 0 0 0 0 |
| − | 78 86 1 0 0 0 0 | + | 78 86 1 0 0 0 0 |
| − | 83 87 1 0 0 0 0 | + | 83 87 1 0 0 0 0 |
| − | 87 88 1 0 0 0 0 | + | 87 88 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGL0051 | + | ID FL7AAAGL0051 |
| − | KNApSAcK_ID C00011088 | + | KNApSAcK_ID C00011088 |
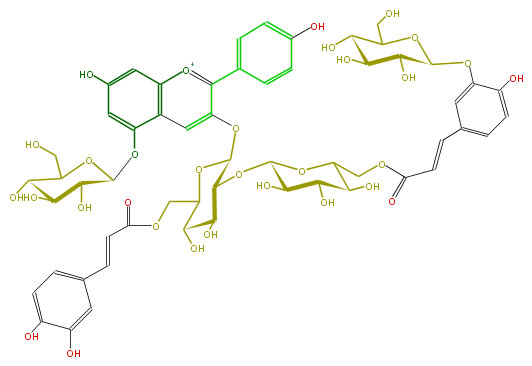
| − | NAME Pelargonidin 3-O-[2-O-(6-O-(trans-3-O-(beta-D-glucopyranosyl)caffeyl)-beta-D-glucopyranosyl)-6-O-(trans-caffeyl)-beta-D-glucopyranoside]-5-O-[beta-D-glucopyranoside] | + | NAME Pelargonidin 3-O-[2-O-(6-O-(trans-3-O-(beta-D-glucopyranosyl)caffeyl)-beta-D-glucopyranosyl)-6-O-(trans-caffeyl)-beta-D-glucopyranoside]-5-O-[beta-D-glucopyranoside] |
| − | CAS_RN 140635-53-6 | + | CAS_RN 140635-53-6 |
| − | FORMULA C57H63O31 | + | FORMULA C57H63O31 |
| − | EXACTMASS 1243.335330298 | + | EXACTMASS 1243.335330298 |
| − | AVERAGEMASS 1244.09152 | + | AVERAGEMASS 1244.09152 |
| − | SMILES C(C(Oc(c9)c([o+1]c(c97)cc(cc7OC(C8O)OC(CO)C(O)C8O)O)c(c6)ccc(O)c6)1)(OC(O3)C(C(O)C(C3COC(=O)C=Cc(c4)cc(OC(C5O)OC(C(C5O)O)CO)c(O)c4)O)O)C(C(O)C(COC(=O)C=Cc(c2)cc(O)c(O)c2)O1)O | + | SMILES C(C(Oc(c9)c([o+1]c(c97)cc(cc7OC(C8O)OC(CO)C(O)C8O)O)c(c6)ccc(O)c6)1)(OC(O3)C(C(O)C(C3COC(=O)C=Cc(c4)cc(OC(C5O)OC(C(C5O)O)CO)c(O)c4)O)O)C(C(O)C(COC(=O)C=Cc(c2)cc(O)c(O)c2)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
88 96 0 0 0 0 0 0 0 0999 V2000
0.0666 2.5038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7259 2.8844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7259 3.6458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0666 4.0264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5928 3.6458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5928 2.8844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2877 3.9701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2850 2.4848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9327 2.8587 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-2.5803 2.4848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5803 1.7369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9327 1.3630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2850 1.7369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2280 2.8587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8757 2.4848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8757 1.7369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2280 1.3630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3581 2.7633 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2280 0.7371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8874 0.0528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2527 -0.4743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5766 -0.0014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7540 0.0610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3886 0.5882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0648 0.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2218 0.7014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7223 0.9904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.2889 -0.3487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7617 -0.3379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5766 -0.5987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6834 1.3896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0054 -1.1765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2132 -0.9461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2262 -0.1212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8169 0.5952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6092 0.3648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5963 -0.4601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6073 0.2361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1774 0.5374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5899 -0.1770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3880 0.0320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1812 -0.1948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7688 0.5198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9707 0.3108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1027 -0.0465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5227 -0.4705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6759 -0.0622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3980 0.1565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9845 0.4952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7468 -1.6244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4027 -1.2743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3429 -0.4601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6909 -1.0628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3835 -1.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3835 -0.4652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8886 -1.3544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8886 -2.0673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5124 -2.4274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2352 -2.2338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7642 -2.7629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5706 -3.4856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8479 -3.6792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3188 -3.1502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8479 -4.2777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9203 -3.8353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5830 0.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2370 -0.4496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3067 0.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5042 1.0808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0050 1.3699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6548 1.1958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1305 1.6715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9564 2.3213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3066 2.4954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8309 2.0197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2889 2.6538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1618 3.0355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2234 3.7054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6359 2.9910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4340 3.2000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2272 2.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8148 3.6878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0167 3.4788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5711 2.6883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1187 3.1296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7282 3.4195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8782 3.9955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1604 4.2777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
6 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 8 1 0 0 0 0
10 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 11 1 0 0 0 0
15 18 1 0 0 0 0
17 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
19 23 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
20 28 1 0 0 0 0
21 29 1 0 0 0 0
22 30 1 0 0 0 0
13 31 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
31 35 1 0 0 0 0
34 38 1 0 0 0 0
39 40 1 1 0 0 0
40 41 1 1 0 0 0
42 41 1 1 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
44 39 1 0 0 0 0
38 39 1 0 0 0 0
40 45 1 0 0 0 0
41 46 1 0 0 0 0
42 47 1 0 0 0 0
43 48 1 0 0 0 0
48 49 1 0 0 0 0
32 50 1 0 0 0 0
33 51 1 0 0 0 0
37 52 1 0 0 0 0
52 53 1 0 0 0 0
53 54 1 0 0 0 0
54 55 2 0 0 0 0
54 56 1 0 0 0 0
56 57 2 0 0 0 0
57 58 1 0 0 0 0
58 59 2 0 0 0 0
59 60 1 0 0 0 0
60 61 2 0 0 0 0
61 62 1 0 0 0 0
62 63 2 0 0 0 0
63 58 1 0 0 0 0
62 64 1 0 0 0 0
61 65 1 0 0 0 0
49 66 1 0 0 0 0
66 67 2 0 0 0 0
66 68 1 0 0 0 0
68 69 2 0 0 0 0
69 70 1 0 0 0 0
70 71 2 0 0 0 0
71 72 1 0 0 0 0
72 73 2 0 0 0 0
73 74 1 0 0 0 0
74 75 2 0 0 0 0
75 70 1 0 0 0 0
73 76 1 0 0 0 0
74 77 1 0 0 0 0
78 79 1 1 0 0 0
79 80 1 1 0 0 0
81 80 1 1 0 0 0
81 82 1 0 0 0 0
82 83 1 0 0 0 0
83 78 1 0 0 0 0
77 81 1 0 0 0 0
80 84 1 0 0 0 0
79 85 1 0 0 0 0
78 86 1 0 0 0 0
83 87 1 0 0 0 0
87 88 1 0 0 0 0
S SKP 8
ID FL7AAAGL0051
KNApSAcK_ID C00011088
NAME Pelargonidin 3-O-[2-O-(6-O-(trans-3-O-(beta-D-glucopyranosyl)caffeyl)-beta-D-glucopyranosyl)-6-O-(trans-caffeyl)-beta-D-glucopyranoside]-5-O-[beta-D-glucopyranoside]
CAS_RN 140635-53-6
FORMULA C57H63O31
EXACTMASS 1243.335330298
AVERAGEMASS 1244.09152
SMILES C(C(Oc(c9)c([o+1]c(c97)cc(cc7OC(C8O)OC(CO)C(O)C8O)O)c(c6)ccc(O)c6)1)(OC(O3)C(C(O)C(C3COC(=O)C=Cc(c4)cc(OC(C5O)OC(C(C5O)O)CO)c(O)c4)O)O)C(C(O)C(COC(=O)C=Cc(c2)cc(O)c(O)c2)O1)O
M END