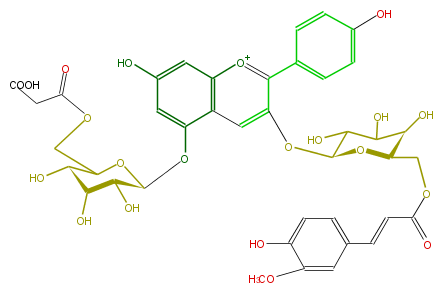
Mol:FL7AAAGL0037
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 61 66 0 0 0 0 0 0 0 0999 V2000 | + | 61 66 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.8224 1.5524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8224 1.5524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8224 0.9100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8224 0.9100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2661 0.5889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2661 0.5889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2902 0.9100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2902 0.9100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2902 1.5524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2902 1.5524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2661 1.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2661 1.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8465 0.5889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8465 0.5889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4028 0.9100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4028 0.9100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4028 1.5524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4028 1.5524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8465 1.8736 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.8465 1.8736 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9589 1.8735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9589 1.8735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5259 1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5259 1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0929 1.8735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0929 1.8735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0929 2.5281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0929 2.5281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5259 2.8555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5259 2.8555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9589 2.5281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9589 2.5281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3785 1.8735 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3785 1.8735 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6597 2.8554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6597 2.8554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2661 -0.0533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2661 -0.0533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8189 0.1893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8189 0.1893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9764 0.4894 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.9764 0.4894 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.6755 -0.0316 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.6755 -0.0316 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.2540 0.1337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2540 0.1337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8360 -0.0316 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.8360 -0.0316 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.1369 0.4894 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.1369 0.4894 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.5584 0.3241 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.5584 0.3241 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.4177 0.3397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4177 0.3397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5999 0.7987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5999 0.7987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4913 0.8438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4913 0.8438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3876 -0.0316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3876 -0.0316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5898 -0.2243 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.5898 -0.2243 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.2187 -0.7143 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.2187 -0.7143 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.6842 -0.5064 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.6842 -0.5064 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.0732 -0.6545 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.0732 -0.6545 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.5432 -0.1260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5432 -0.1260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0108 -0.3728 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.0108 -0.3728 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.1473 -0.4272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1473 -0.4272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3413 -1.2804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3413 -1.2804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3779 -1.0205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3779 -1.0205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8685 0.1413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8685 0.1413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5781 -0.7428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5781 -0.7428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2887 -1.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2887 -1.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7736 -1.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7736 -1.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4792 -1.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4792 -1.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9899 -1.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9899 -1.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7007 -1.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7007 -1.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1223 -1.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1223 -1.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8331 -1.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8331 -1.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1223 -2.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1223 -2.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7007 -2.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7007 -2.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2556 -1.7539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2556 -1.7539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5844 -1.7562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5844 -1.7562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1996 0.7777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1996 0.7777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7453 1.2355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7453 1.2355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2420 1.0547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2420 1.0547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6954 1.4351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6954 1.4351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3472 1.1979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3472 1.1979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6528 1.7599 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6528 1.7599 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5750 2.1176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5750 2.1176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4583 -2.4430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4583 -2.4430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1727 -2.8555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1727 -2.8555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 34 19 1 0 0 0 0 | + | 34 19 1 0 0 0 0 |
| − | 30 41 1 0 0 0 0 | + | 30 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 42 52 2 0 0 0 0 | + | 42 52 2 0 0 0 0 |
| − | 40 53 1 0 0 0 0 | + | 40 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 2 0 0 0 0 | + | 56 57 2 0 0 0 0 |
| − | 54 58 2 0 0 0 0 | + | 54 58 2 0 0 0 0 |
| − | 56 59 1 0 0 0 0 | + | 56 59 1 0 0 0 0 |
| − | 49 60 1 0 0 0 0 | + | 49 60 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 60 61 | + | M SAL 2 2 60 61 |
| − | M SBL 2 1 65 | + | M SBL 2 1 65 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 65 1.4583 -2.443 | + | M SVB 2 65 1.4583 -2.443 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 56 57 59 | + | M SAL 1 3 56 57 59 |
| − | M SBL 1 1 61 | + | M SBL 1 1 61 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SVB 1 61 -3.9837 1.1151 | + | M SVB 1 61 -3.9837 1.1151 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGL0037 | + | ID FL7AAAGL0037 |
| − | KNApSAcK_ID C00006776 | + | KNApSAcK_ID C00006776 |
| − | NAME Palargonidin 3-(6''-ferulylglucoside)-5-(6'''-malonylglucoside) | + | NAME Palargonidin 3-(6''-ferulylglucoside)-5-(6'''-malonylglucoside) |
| − | CAS_RN 168647-45-8 | + | CAS_RN 168647-45-8 |
| − | FORMULA C40H41O21 | + | FORMULA C40H41O21 |
| − | EXACTMASS 857.214033374 | + | EXACTMASS 857.214033374 |
| − | AVERAGEMASS 857.74094 | + | AVERAGEMASS 857.74094 |
| − | SMILES O[C@@H]([C@H]6O)[C@@H](OC([C@@H]6O)COC(=O)CC(O)=O)Oc(c1)c(c2)c([o+1]c(c(c5)ccc(c5)O)c2O[C@@H](C4O)O[C@@H]([C@H](O)C(O)4)COC(C=Cc(c3)cc(OC)c(c3)O)=O)cc1O | + | SMILES O[C@@H]([C@H]6O)[C@@H](OC([C@@H]6O)COC(=O)CC(O)=O)Oc(c1)c(c2)c([o+1]c(c(c5)ccc(c5)O)c2O[C@@H](C4O)O[C@@H]([C@H](O)C(O)4)COC(C=Cc(c3)cc(OC)c(c3)O)=O)cc1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
61 66 0 0 0 0 0 0 0 0999 V2000
-0.8224 1.5524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8224 0.9100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2661 0.5889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2902 0.9100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2902 1.5524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2661 1.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8465 0.5889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4028 0.9100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4028 1.5524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8465 1.8736 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.9589 1.8735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5259 1.5461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0929 1.8735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0929 2.5281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5259 2.8555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9589 2.5281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3785 1.8735 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6597 2.8554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2661 -0.0533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8189 0.1893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9764 0.4894 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.6755 -0.0316 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.2540 0.1337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8360 -0.0316 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.1369 0.4894 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.5584 0.3241 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.4177 0.3397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5999 0.7987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4913 0.8438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3876 -0.0316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5898 -0.2243 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.2187 -0.7143 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.6842 -0.5064 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.0732 -0.6545 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.5432 -0.1260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0108 -0.3728 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.1473 -0.4272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3413 -1.2804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3779 -1.0205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8685 0.1413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5781 -0.7428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2887 -1.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7736 -1.2441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4792 -1.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9899 -1.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7007 -1.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1223 -1.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8331 -1.7539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1223 -2.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7007 -2.2548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2556 -1.7539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5844 -1.7562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1996 0.7777 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7453 1.2355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2420 1.0547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6954 1.4351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3472 1.1979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6528 1.7599 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5750 2.1176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4583 -2.4430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1727 -2.8555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
22 20 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
36 40 1 0 0 0 0
34 19 1 0 0 0 0
30 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 2 0 0 0 0
44 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 45 1 0 0 0 0
48 51 1 0 0 0 0
42 52 2 0 0 0 0
40 53 1 0 0 0 0
53 54 1 0 0 0 0
54 55 1 0 0 0 0
55 56 1 0 0 0 0
56 57 2 0 0 0 0
54 58 2 0 0 0 0
56 59 1 0 0 0 0
49 60 1 0 0 0 0
60 61 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 60 61
M SBL 2 1 65
M SMT 2 OCH3
M SVB 2 65 1.4583 -2.443
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 56 57 59
M SBL 1 1 61
M SMT 1 COOH
M SVB 1 61 -3.9837 1.1151
S SKP 8
ID FL7AAAGL0037
KNApSAcK_ID C00006776
NAME Palargonidin 3-(6''-ferulylglucoside)-5-(6'''-malonylglucoside)
CAS_RN 168647-45-8
FORMULA C40H41O21
EXACTMASS 857.214033374
AVERAGEMASS 857.74094
SMILES O[C@@H]([C@H]6O)[C@@H](OC([C@@H]6O)COC(=O)CC(O)=O)Oc(c1)c(c2)c([o+1]c(c(c5)ccc(c5)O)c2O[C@@H](C4O)O[C@@H]([C@H](O)C(O)4)COC(C=Cc(c3)cc(OC)c(c3)O)=O)cc1O
M END