Mol:FL7AAAGL0017
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.4579 1.2141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4579 1.2141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4579 0.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4579 0.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9016 0.2506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9016 0.2506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3453 0.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3453 0.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3453 1.2141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3453 1.2141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9016 1.5353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9016 1.5353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7890 0.2506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7890 0.2506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2327 0.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2327 0.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2327 1.2141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2327 1.2141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7890 1.5353 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -2.7890 1.5353 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6766 1.5352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6766 1.5352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1097 1.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1097 1.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5427 1.5352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5427 1.5352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5427 2.1899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5427 2.1899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1097 2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1097 2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6766 2.1899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6766 2.1899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0140 1.5352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0140 1.5352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0241 2.5171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0241 2.5171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9016 -0.3916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9016 -0.3916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8166 -0.1489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8166 -0.1489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3138 -0.1727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3138 -0.1727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5820 -0.6373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5820 -0.6373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0662 -0.4899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0662 -0.4899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4527 -0.6373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4527 -0.6373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7210 -0.1727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7210 -0.1727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2052 -0.3201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2052 -0.3201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8119 -0.3062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8119 -0.3062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3469 0.2087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3469 0.2087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1282 -0.1727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1282 -0.1727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0024 -0.4900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0024 -0.4900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2005 -1.2009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2005 -1.2009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7755 -1.5328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7755 -1.5328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4797 -2.0452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4797 -2.0452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2625 -1.5290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2625 -1.5290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5719 -2.0034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5719 -2.0034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2348 -2.0034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2348 -2.0034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5314 -2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5314 -2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1248 -2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1248 -2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4214 -2.0034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4214 -2.0034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1248 -1.4896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1248 -1.4896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5314 -1.4896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5314 -1.4896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0140 -2.0034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0140 -2.0034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGL0017 | + | ID FL7AAAGL0017 |
| − | KNApSAcK_ID C00006756 | + | KNApSAcK_ID C00006756 |
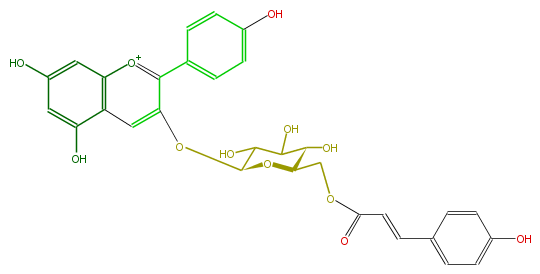
| − | NAME Pelargonidin 3-p-coumarylglucoside | + | NAME Pelargonidin 3-p-coumarylglucoside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C30H27O12 | + | FORMULA C30H27O12 |
| − | EXACTMASS 579.150251328 | + | EXACTMASS 579.150251328 |
| − | AVERAGEMASS 579.52818 | + | AVERAGEMASS 579.52818 |
| − | SMILES OC(C(Oc(c(c(c5)ccc(O)c5)3)cc(c4O)c(cc(O)c4)[o+1]3)1)C(O)C(O)C(COC(C=Cc(c2)ccc(O)c2)=O)O1 | + | SMILES OC(C(Oc(c(c(c5)ccc(O)c5)3)cc(c4O)c(cc(O)c4)[o+1]3)1)C(O)C(O)C(COC(C=Cc(c2)ccc(O)c2)=O)O1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-4.4579 1.2141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4579 0.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9016 0.2506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3453 0.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3453 1.2141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9016 1.5353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7890 0.2506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2327 0.5718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2327 1.2141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7890 1.5353 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-1.6766 1.5352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1097 1.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5427 1.5352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5427 2.1899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1097 2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6766 2.1899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0140 1.5352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0241 2.5171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9016 -0.3916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8166 -0.1489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3138 -0.1727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5820 -0.6373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0662 -0.4899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4527 -0.6373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7210 -0.1727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2052 -0.3201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8119 -0.3062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3469 0.2087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1282 -0.1727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0024 -0.4900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2005 -1.2009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7755 -1.5328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4797 -2.0452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2625 -1.5290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5719 -2.0034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2348 -2.0034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5314 -2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1248 -2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4214 -2.0034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1248 -1.4896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5314 -1.4896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0140 -2.0034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
24 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
S SKP 8
ID FL7AAAGL0017
KNApSAcK_ID C00006756
NAME Pelargonidin 3-p-coumarylglucoside
CAS_RN -
FORMULA C30H27O12
EXACTMASS 579.150251328
AVERAGEMASS 579.52818
SMILES OC(C(Oc(c(c(c5)ccc(O)c5)3)cc(c4O)c(cc(O)c4)[o+1]3)1)C(O)C(O)C(COC(C=Cc(c2)ccc(O)c2)=O)O1
M END