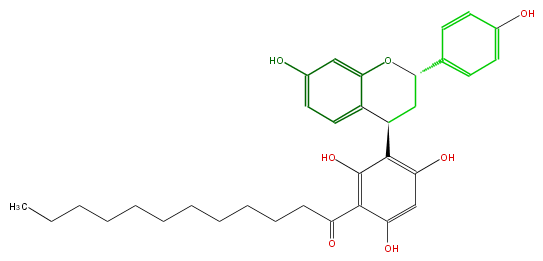
Mol:FL6F1ANC0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 40 43 0 0 0 0 0 0 0 0999 V2000 | + | 40 43 0 0 0 0 0 0 0 0999 V2000 |
| − | 3.7319 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7319 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0174 1.8503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0174 1.8503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3030 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3030 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3030 0.6128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3030 0.6128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0174 0.2003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0174 0.2003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7319 0.6128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7319 0.6128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5885 1.8503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5885 1.8503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8740 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8740 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8740 0.6128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8740 0.6128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5885 0.2003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5885 0.2003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1595 1.8503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1595 1.8503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4464 1.8503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4464 1.8503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1608 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1608 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8753 1.8503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8753 1.8503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8753 2.6753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8753 2.6753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1608 3.0878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1608 3.0878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4464 2.6753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4464 2.6753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5898 3.0878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5898 3.0878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0000 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0000 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7376 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7376 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7376 -1.1328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7376 -1.1328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0000 -0.7069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0000 -0.7069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2623 -1.1328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2623 -1.1328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2623 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2623 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0000 -3.0878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0000 -3.0878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4753 -0.7069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4753 -0.7069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5246 -0.7069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5246 -0.7069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7869 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7869 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5246 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5246 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6884 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6884 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0493 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0493 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1638 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1638 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4261 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4261 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6391 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6391 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9014 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9014 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1144 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1144 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3768 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3768 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.5898 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.5898 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8521 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8521 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5246 -2.9715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5246 -2.9715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 2 0 0 0 0 | + | 3 7 2 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 2 0 0 0 0 | + | 10 4 2 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 1 12 1 6 0 0 0 | + | 1 12 1 6 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 19 20 2 0 0 0 0 | + | 19 20 2 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 2 0 0 0 0 | + | 21 22 2 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 5 22 1 1 0 0 0 | + | 5 22 1 1 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 28 1 0 0 0 0 | + | 31 28 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 30 1 0 0 0 0 | + | 33 30 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 32 1 0 0 0 0 | + | 35 32 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 34 1 0 0 0 0 | + | 37 34 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 36 1 0 0 0 0 | + | 39 36 1 0 0 0 0 |
| − | 29 40 2 0 0 0 0 | + | 29 40 2 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL6F1ANC0002 | + | ID FL6F1ANC0002 |
| − | KNApSAcK_ID C00013273 | + | KNApSAcK_ID C00013273 |
| − | NAME (+)-Myristinin A;Myristinin A;trans-(+)-1-[3-[3,4-Dihydro-7-hydroxy-2-(4-hydroxyphenyl)-2H-1-benzopyran-4-yl]-2,4,6-trihydroxyphenyl]-1-dodecanone | + | NAME (+)-Myristinin A;Myristinin A;trans-(+)-1-[3-[3,4-Dihydro-7-hydroxy-2-(4-hydroxyphenyl)-2H-1-benzopyran-4-yl]-2,4,6-trihydroxyphenyl]-1-dodecanone |
| − | CAS_RN 145904-69-4 | + | CAS_RN 145904-69-4 |
| − | FORMULA C33H40O7 | + | FORMULA C33H40O7 |
| − | EXACTMASS 548.2774036339999 | + | EXACTMASS 548.2774036339999 |
| − | AVERAGEMASS 548.6665 | + | AVERAGEMASS 548.6665 |
| − | SMILES c(c4)(O)cc(O2)c(c4)C(CC(c(c3)ccc(O)c3)2)c(c1O)c(O)cc(c1C(CCCCCCCCCCC)=O)O | + | SMILES c(c4)(O)cc(O2)c(c4)C(CC(c(c3)ccc(O)c3)2)c(c1O)c(O)cc(c1C(CCCCCCCCCCC)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
40 43 0 0 0 0 0 0 0 0999 V2000
3.7319 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0174 1.8503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3030 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3030 0.6128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0174 0.2003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7319 0.6128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5885 1.8503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8740 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8740 0.6128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5885 0.2003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1595 1.8503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4464 1.8503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1608 1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8753 1.8503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8753 2.6753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1608 3.0878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4464 2.6753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5898 3.0878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0000 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7376 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7376 -1.1328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0000 -0.7069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2623 -1.1328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2623 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0000 -3.0878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4753 -0.7069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5246 -0.7069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7869 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5246 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6884 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0493 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1638 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4261 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6391 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9014 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1144 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3768 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.5898 -1.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8521 -2.4105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5246 -2.9715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 1 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 1 0 0 0 0
3 7 2 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 4 2 0 0 0 0
8 11 1 0 0 0 0
1 12 1 6 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
19 20 2 0 0 0 0
20 21 1 0 0 0 0
21 22 2 0 0 0 0
22 23 1 0 0 0 0
23 24 2 0 0 0 0
24 19 1 0 0 0 0
5 22 1 1 0 0 0
19 25 1 0 0 0 0
26 21 1 0 0 0 0
23 27 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
30 31 1 0 0 0 0
31 28 1 0 0 0 0
32 33 1 0 0 0 0
33 30 1 0 0 0 0
34 35 1 0 0 0 0
35 32 1 0 0 0 0
36 37 1 0 0 0 0
37 34 1 0 0 0 0
38 39 1 0 0 0 0
39 36 1 0 0 0 0
29 40 2 0 0 0 0
S SKP 8
ID FL6F1ANC0002
KNApSAcK_ID C00013273
NAME (+)-Myristinin A;Myristinin A;trans-(+)-1-[3-[3,4-Dihydro-7-hydroxy-2-(4-hydroxyphenyl)-2H-1-benzopyran-4-yl]-2,4,6-trihydroxyphenyl]-1-dodecanone
CAS_RN 145904-69-4
FORMULA C33H40O7
EXACTMASS 548.2774036339999
AVERAGEMASS 548.6665
SMILES c(c4)(O)cc(O2)c(c4)C(CC(c(c3)ccc(O)c3)2)c(c1O)c(O)cc(c1C(CCCCCCCCCCC)=O)O
M END