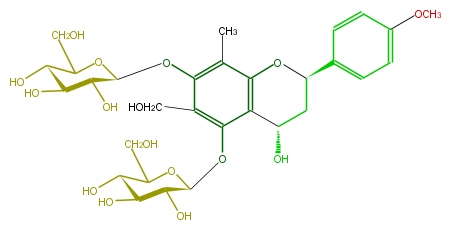
Mol:FL64ABGM0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.8595 0.7174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8595 0.7174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8595 0.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8595 0.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3032 -0.2461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3032 -0.2461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2531 0.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2531 0.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2531 0.7174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2531 0.7174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3032 1.0386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3032 1.0386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8094 -0.2461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8094 -0.2461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3657 0.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3657 0.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3657 0.7174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3657 0.7174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8094 1.0386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8094 1.0386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9218 1.0385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9218 1.0385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4888 0.7111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4888 0.7111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0558 1.0385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0558 1.0385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0558 1.6932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0558 1.6932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4888 2.0205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4888 2.0205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9218 1.6932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9218 1.6932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3032 -0.8882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3032 -0.8882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4156 1.0385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4156 1.0385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8094 -0.8882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8094 -0.8882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3032 1.6807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3032 1.6807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8232 0.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8232 0.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4520 0.4720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4520 0.4720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9175 0.6798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9175 0.6798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4018 0.6854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4018 0.6854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7766 1.0603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7766 1.0603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2442 0.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2442 0.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3370 0.6653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3370 0.6653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0307 0.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0307 0.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6113 0.1657 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6113 0.1657 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3588 -1.2243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3588 -1.2243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9877 -1.7143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9877 -1.7143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4532 -1.5064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4532 -1.5064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9374 -1.5008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9374 -1.5008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3122 -1.1260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3122 -1.1260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7798 -1.3728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7798 -1.3728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8727 -1.5210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8727 -1.5210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5663 -1.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5663 -1.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1469 -2.0205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1469 -2.0205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5740 0.1665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5740 0.1665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8595 -0.2460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8595 -0.2460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6426 1.5977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6426 1.5977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8457 1.3841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8457 1.3841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6226 2.0204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6226 2.0204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3370 1.6079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3370 1.6079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1782 -0.5886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1782 -0.5886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3813 -0.8021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3813 -0.8021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 1 0 0 0 | + | 9 11 1 1 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 3 17 1 0 0 0 0 | + | 3 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 7 19 1 6 0 0 0 | + | 7 19 1 6 0 0 0 |
| − | 6 20 1 0 0 0 0 | + | 6 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 17 1 0 0 0 0 | + | 33 17 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 2 39 1 0 0 0 0 | + | 2 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 26 41 1 0 0 0 0 | + | 26 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 14 43 1 0 0 0 0 | + | 14 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 35 45 1 0 0 0 0 | + | 35 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 43 -8.3348 5.0370 | + | M SBV 1 43 -8.3348 5.0370 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 45 -8.0188 5.7297 | + | M SBV 2 45 -8.0188 5.7297 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 43 44 | + | M SAL 3 2 43 44 |
| − | M SBL 3 1 47 | + | M SBL 3 1 47 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 47 -7.0536 5.2728 | + | M SBV 3 47 -7.0536 5.2728 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 45 46 | + | M SAL 4 2 45 46 |
| − | M SBL 4 1 49 | + | M SBL 4 1 49 |
| − | M SMT 4 ^CH2OH | + | M SMT 4 ^CH2OH |
| − | M SBV 4 49 -8.0188 5.7297 | + | M SBV 4 49 -8.0188 5.7297 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL64ABGM0003 | + | ID FL64ABGM0003 |
| − | KNApSAcK_ID C00008982 | + | KNApSAcK_ID C00008982 |
| − | NAME Triphyllin A | + | NAME Triphyllin A |
| − | CAS_RN 101395-02-2 | + | CAS_RN 101395-02-2 |
| − | FORMULA C30H40O16 | + | FORMULA C30H40O16 |
| − | EXACTMASS 656.2316352319999 | + | EXACTMASS 656.2316352319999 |
| − | AVERAGEMASS 656.629 | + | AVERAGEMASS 656.629 |
| − | SMILES C(O)(C(O)5)C(OC(C(O)5)Oc(c(CO)3)c(c(c(C)c(OC(O4)C(C(O)C(C4CO)O)O)3)1)C(O)CC(c(c2)ccc(c2)OC)O1)CO | + | SMILES C(O)(C(O)5)C(OC(C(O)5)Oc(c(CO)3)c(c(c(C)c(OC(O4)C(C(O)C(C4CO)O)O)3)1)C(O)CC(c(c2)ccc(c2)OC)O1)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
-0.8595 0.7174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8595 0.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3032 -0.2461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2531 0.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2531 0.7174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3032 1.0386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8094 -0.2461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3657 0.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3657 0.7174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8094 1.0386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9218 1.0385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4888 0.7111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0558 1.0385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0558 1.6932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4888 2.0205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9218 1.6932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3032 -0.8882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4156 1.0385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8094 -0.8882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3032 1.6807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8232 0.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4520 0.4720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9175 0.6798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4018 0.6854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7766 1.0603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2442 0.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3370 0.6653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0307 0.4438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6113 0.1657 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3588 -1.2243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9877 -1.7143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4532 -1.5064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9374 -1.5008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3122 -1.1260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7798 -1.3728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8727 -1.5210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5663 -1.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1469 -2.0205 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5740 0.1665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8595 -0.2460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6426 1.5977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8457 1.3841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6226 2.0204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3370 1.6079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1782 -0.5886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3813 -0.8021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 1 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
3 17 1 0 0 0 0
1 18 1 0 0 0 0
7 19 1 6 0 0 0
6 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 17 1 0 0 0 0
24 18 1 0 0 0 0
2 39 1 0 0 0 0
39 40 1 0 0 0 0
26 41 1 0 0 0 0
41 42 1 0 0 0 0
14 43 1 0 0 0 0
43 44 1 0 0 0 0
35 45 1 0 0 0 0
45 46 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 43
M SMT 1 ^CH2OH
M SBV 1 43 -8.3348 5.0370
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 45
M SMT 2 ^CH2OH
M SBV 2 45 -8.0188 5.7297
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 43 44
M SBL 3 1 47
M SMT 3 OCH3
M SBV 3 47 -7.0536 5.2728
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 45 46
M SBL 4 1 49
M SMT 4 ^CH2OH
M SBV 4 49 -8.0188 5.7297
S SKP 8
ID FL64ABGM0003
KNApSAcK_ID C00008982
NAME Triphyllin A
CAS_RN 101395-02-2
FORMULA C30H40O16
EXACTMASS 656.2316352319999
AVERAGEMASS 656.629
SMILES C(O)(C(O)5)C(OC(C(O)5)Oc(c(CO)3)c(c(c(C)c(OC(O4)C(C(O)C(C4CO)O)O)3)1)C(O)CC(c(c2)ccc(c2)OC)O1)CO
M END