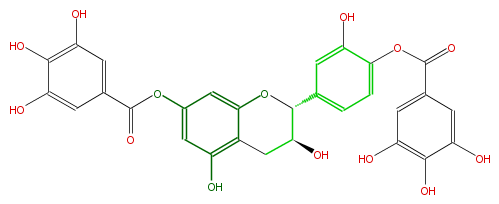
Mol:FL63ACNS0021
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3204 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3204 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3204 -0.7592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3204 -0.7592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7819 -1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7819 -1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2433 -0.7592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2433 -0.7592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2433 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2433 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7819 0.1736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7819 0.1736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2952 -1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2952 -1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8337 -0.7592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8337 -0.7592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8337 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8337 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2952 0.1736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2952 0.1736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8586 0.1734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8586 0.1734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3447 -1.0542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3447 -1.0542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3133 0.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3133 0.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8580 -0.1750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8580 -0.1750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4027 0.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4027 0.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4027 0.7685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4027 0.7685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8580 1.0830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8580 1.0830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3133 0.7685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3133 0.7685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7819 -1.6916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7819 -1.6916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3969 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3969 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9351 0.1734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9351 0.1734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3969 -0.7563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3969 -0.7563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9351 0.8116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9351 0.8116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4878 1.1307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4878 1.1307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0404 0.8116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0404 0.8116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0404 0.1734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0404 0.1734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4878 -0.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4878 -0.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4878 1.7687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4878 1.7687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5930 1.1306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5930 1.1306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5930 -0.1456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5930 -0.1456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9469 1.0827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9469 1.0827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4899 0.7692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4899 0.7692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0329 1.0827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0329 1.0827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4899 0.1422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4899 0.1422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9381 -0.1764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9381 -0.1764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9381 -0.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9381 -0.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4899 -1.1320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4899 -1.1320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0416 -0.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0416 -0.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0416 -0.1764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0416 -0.1764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3868 -1.1318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3868 -1.1318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4899 -1.7687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4899 -1.7687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5930 -1.1318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5930 -1.1318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8580 1.7114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8580 1.7114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 1 11 1 0 0 0 0 | + | 1 11 1 0 0 0 0 |
| − | 8 12 1 1 0 0 0 | + | 8 12 1 1 0 0 0 |
| − | 9 13 1 6 0 0 0 | + | 9 13 1 6 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 11 20 1 0 0 0 0 | + | 11 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 20 22 2 0 0 0 0 | + | 20 22 2 0 0 0 0 |
| − | 21 23 2 0 0 0 0 | + | 21 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 21 1 0 0 0 0 | + | 27 21 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 16 31 1 0 0 0 0 | + | 16 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | 17 43 1 0 0 0 0 | + | 17 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL63ACNS0021 | + | ID FL63ACNS0021 |
| − | KNApSAcK_ID C00008881 | + | KNApSAcK_ID C00008881 |
| − | NAME Catechin 7,4'-di-O-gallate | + | NAME Catechin 7,4'-di-O-gallate |
| − | CAS_RN 110784-25-3 | + | CAS_RN 110784-25-3 |
| − | FORMULA C29H22O14 | + | FORMULA C29H22O14 |
| − | EXACTMASS 594.100955412 | + | EXACTMASS 594.100955412 |
| − | AVERAGEMASS 594.47658 | + | AVERAGEMASS 594.47658 |
| − | SMILES Oc(c4OC(=O)c(c5)cc(O)c(O)c(O)5)cc(cc4)C(O1)C(O)Cc(c(O)2)c1cc(OC(c(c3)cc(c(O)c3O)O)=O)c2 | + | SMILES Oc(c4OC(=O)c(c5)cc(O)c(O)c(O)5)cc(cc4)C(O1)C(O)Cc(c(O)2)c1cc(OC(c(c3)cc(c(O)c3O)O)=O)c2 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.3204 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3204 -0.7592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7819 -1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2433 -0.7592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2433 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7819 0.1736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2952 -1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8337 -0.7592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8337 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2952 0.1736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8586 0.1734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3447 -1.0542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3133 0.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8580 -0.1750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4027 0.1395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4027 0.7685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8580 1.0830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3133 0.7685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7819 -1.6916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3969 -0.1373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9351 0.1734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3969 -0.7563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9351 0.8116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4878 1.1307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0404 0.8116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0404 0.1734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4878 -0.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4878 1.7687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5930 1.1306 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5930 -0.1456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9469 1.0827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4899 0.7692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0329 1.0827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4899 0.1422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9381 -0.1764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9381 -0.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4899 -1.1320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0416 -0.8135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0416 -0.1764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3868 -1.1318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4899 -1.7687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5930 -1.1318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8580 1.7114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
1 11 1 0 0 0 0
8 12 1 1 0 0 0
9 13 1 6 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
3 19 1 0 0 0 0
11 20 1 0 0 0 0
20 21 1 0 0 0 0
20 22 2 0 0 0 0
21 23 2 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 21 1 0 0 0 0
24 28 1 0 0 0 0
25 29 1 0 0 0 0
26 30 1 0 0 0 0
16 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 34 1 0 0 0 0
36 40 1 0 0 0 0
37 41 1 0 0 0 0
38 42 1 0 0 0 0
17 43 1 0 0 0 0
S SKP 8
ID FL63ACNS0021
KNApSAcK_ID C00008881
NAME Catechin 7,4'-di-O-gallate
CAS_RN 110784-25-3
FORMULA C29H22O14
EXACTMASS 594.100955412
AVERAGEMASS 594.47658
SMILES Oc(c4OC(=O)c(c5)cc(O)c(O)c(O)5)cc(cc4)C(O1)C(O)Cc(c(O)2)c1cc(OC(c(c3)cc(c(O)c3O)O)=O)c2
M END