Mol:FL63ACNS0019
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1391 0.2599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1391 0.2599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1391 -0.3619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1391 -0.3619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6006 -0.6728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6006 -0.6728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0620 -0.3619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0620 -0.3619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0620 0.2599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0620 0.2599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6006 0.5708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6006 0.5708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5235 -0.6728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5235 -0.6728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0150 -0.3619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0150 -0.3619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0150 0.2599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0150 0.2599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5235 0.5708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5235 0.5708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6773 0.5707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6773 0.5707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5260 -0.6569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5260 -0.6569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4945 0.5368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4945 0.5368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0393 0.2223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0393 0.2223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5840 0.5368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5840 0.5368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5840 1.1658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5840 1.1658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0393 1.4802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0393 1.4802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4945 1.1658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4945 1.1658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6006 -1.2943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6006 -1.2943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1185 -1.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1185 -1.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6534 -2.3296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6534 -2.3296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7785 -1.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7785 -1.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1274 -1.2601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1274 -1.2601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8253 -1.2601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8253 -1.2601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1743 -1.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1743 -1.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8253 -2.4689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8253 -2.4689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1274 -2.4689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1274 -2.4689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1739 -0.6563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1739 -0.6563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8714 -1.8645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8714 -1.8645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1739 -3.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1739 -3.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1282 1.4799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1282 1.4799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0393 2.1086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0393 2.1086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6724 1.1658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6724 1.1658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2154 1.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2154 1.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6724 0.5388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6724 0.5388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7677 1.1603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7677 1.1603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3201 1.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3201 1.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3201 2.1171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3201 2.1171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7677 2.4360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7677 2.4360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2154 2.1171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2154 2.1171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7677 3.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7677 3.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8714 2.4354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8714 2.4354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8714 1.1609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8714 1.1609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 1 11 1 0 0 0 0 | + | 1 11 1 0 0 0 0 |
| − | 8 12 1 1 0 0 0 | + | 8 12 1 1 0 0 0 |
| − | 9 13 1 6 0 0 0 | + | 9 13 1 6 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 2 0 0 0 0 | + | 20 21 2 0 0 0 0 |
| − | 20 22 1 0 0 0 0 | + | 20 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 16 31 1 0 0 0 0 | + | 16 31 1 0 0 0 0 |
| − | 17 32 1 0 0 0 0 | + | 17 32 1 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 33 35 2 0 0 0 0 | + | 33 35 2 0 0 0 0 |
| − | 34 36 2 0 0 0 0 | + | 34 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 34 1 0 0 0 0 | + | 40 34 1 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL63ACNS0019 | + | ID FL63ACNS0019 |
| − | KNApSAcK_ID C00008879 | + | KNApSAcK_ID C00008879 |
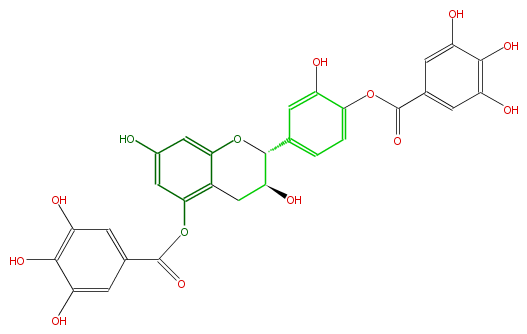
| − | NAME Catechin 5,4'-di-O-gallate | + | NAME Catechin 5,4'-di-O-gallate |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C29H22O14 | + | FORMULA C29H22O14 |
| − | EXACTMASS 594.100955412 | + | EXACTMASS 594.100955412 |
| − | AVERAGEMASS 594.47658 | + | AVERAGEMASS 594.47658 |
| − | SMILES O=C(Oc(c25)cc(O)cc(OC(C(C5)O)c(c4)cc(O)c(c4)OC(=O)c(c3)cc(O)c(O)c(O)3)2)c(c1)cc(c(c1O)O)O | + | SMILES O=C(Oc(c25)cc(O)cc(OC(C(C5)O)c(c4)cc(O)c(c4)OC(=O)c(c3)cc(O)c(O)c(O)3)2)c(c1)cc(c(c1O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.1391 0.2599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1391 -0.3619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6006 -0.6728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0620 -0.3619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0620 0.2599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6006 0.5708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5235 -0.6728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0150 -0.3619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0150 0.2599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5235 0.5708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6773 0.5707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5260 -0.6569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4945 0.5368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0393 0.2223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5840 0.5368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5840 1.1658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0393 1.4802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4945 1.1658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6006 -1.2943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1185 -1.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6534 -2.3296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7785 -1.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1274 -1.2601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8253 -1.2601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1743 -1.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8253 -2.4689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1274 -2.4689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1739 -0.6563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8714 -1.8645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1739 -3.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1282 1.4799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0393 2.1086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6724 1.1658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2154 1.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6724 0.5388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7677 1.1603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3201 1.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3201 2.1171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7677 2.4360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2154 2.1171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7677 3.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8714 2.4354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8714 1.1609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
1 11 1 0 0 0 0
8 12 1 1 0 0 0
9 13 1 6 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
3 19 1 0 0 0 0
19 20 1 0 0 0 0
20 21 2 0 0 0 0
20 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 22 1 0 0 0 0
24 28 1 0 0 0 0
25 29 1 0 0 0 0
26 30 1 0 0 0 0
16 31 1 0 0 0 0
17 32 1 0 0 0 0
31 33 1 0 0 0 0
33 34 1 0 0 0 0
33 35 2 0 0 0 0
34 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 34 1 0 0 0 0
39 41 1 0 0 0 0
38 42 1 0 0 0 0
37 43 1 0 0 0 0
S SKP 8
ID FL63ACNS0019
KNApSAcK_ID C00008879
NAME Catechin 5,4'-di-O-gallate
CAS_RN -
FORMULA C29H22O14
EXACTMASS 594.100955412
AVERAGEMASS 594.47658
SMILES O=C(Oc(c25)cc(O)cc(OC(C(C5)O)c(c4)cc(O)c(c4)OC(=O)c(c3)cc(O)c(O)c(O)3)2)c(c1)cc(c(c1O)O)O
M END