Mol:FL63ACNS0018
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8310 1.0561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8310 1.0561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8310 0.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8310 0.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2925 0.1233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2925 0.1233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7540 0.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7540 0.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7540 1.0561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7540 1.0561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2925 1.3670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2925 1.3670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2155 0.1233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2155 0.1233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3231 0.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3231 0.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3231 1.0561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3231 1.0561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2155 1.3670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2155 1.3670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3693 1.3668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3693 1.3668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8341 0.1392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8341 0.1392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8026 1.3329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8026 1.3329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3473 1.0185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3473 1.0185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8920 1.3329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8920 1.3329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8920 1.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8920 1.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3473 2.2764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3473 2.2764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8026 1.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8026 1.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2925 -0.4982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2925 -0.4982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8104 -1.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8104 -1.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3453 -1.5334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3453 -1.5334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4704 -1.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4704 -1.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8194 -0.4639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8194 -0.4639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5173 -0.4639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5173 -0.4639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8662 -1.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8662 -1.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5173 -1.6727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5173 -1.6727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8194 -1.6727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8194 -1.6727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8658 0.1398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8658 0.1398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5633 -1.0683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5633 -1.0683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8658 -2.2764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8658 -2.2764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4362 1.0188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4362 1.0188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4362 0.3904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4362 0.3904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9792 0.0769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9792 0.0769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8932 0.0769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8932 0.0769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9792 -0.5331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9792 -0.5331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5075 -0.8382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5075 -0.8382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0358 -0.5331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0358 -0.5331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0358 0.0769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0358 0.0769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5075 0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5075 0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5633 0.3814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5633 0.3814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5633 -0.8377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5633 -0.8377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5075 -1.4473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5075 -1.4473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4362 2.2761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4362 2.2761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 1 11 1 0 0 0 0 | + | 1 11 1 0 0 0 0 |
| − | 8 12 1 1 0 0 0 | + | 8 12 1 1 0 0 0 |
| − | 9 13 1 6 0 0 0 | + | 9 13 1 6 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 2 0 0 0 0 | + | 20 21 2 0 0 0 0 |
| − | 20 22 1 0 0 0 0 | + | 20 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 15 31 1 0 0 0 0 | + | 15 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 32 34 2 0 0 0 0 | + | 32 34 2 0 0 0 0 |
| − | 33 35 2 0 0 0 0 | + | 33 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 33 1 0 0 0 0 | + | 39 33 1 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 16 43 1 0 0 0 0 | + | 16 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL63ACNS0018 | + | ID FL63ACNS0018 |
| − | KNApSAcK_ID C00008878 | + | KNApSAcK_ID C00008878 |
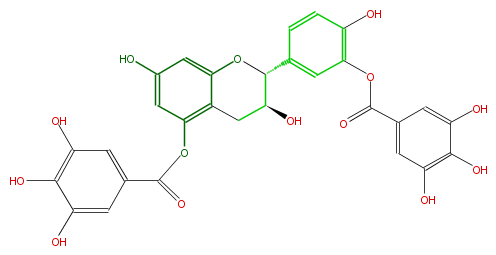
| − | NAME Catechin 5,3'-di-O-gallate | + | NAME Catechin 5,3'-di-O-gallate |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C29H22O14 | + | FORMULA C29H22O14 |
| − | EXACTMASS 594.100955412 | + | EXACTMASS 594.100955412 |
| − | AVERAGEMASS 594.47658 | + | AVERAGEMASS 594.47658 |
| − | SMILES O=C(Oc(c25)cc(O)cc2OC(C(O)C5)c(c3)ccc(c3OC(c(c4)cc(O)c(c(O)4)O)=O)O)c(c1)cc(c(c1O)O)O | + | SMILES O=C(Oc(c25)cc(O)cc2OC(C(O)C5)c(c3)ccc(c3OC(c(c4)cc(O)c(c(O)4)O)=O)O)c(c1)cc(c(c1O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.8310 1.0561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8310 0.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2925 0.1233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7540 0.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7540 1.0561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2925 1.3670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2155 0.1233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3231 0.4342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3231 1.0561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2155 1.3670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3693 1.3668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8341 0.1392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8026 1.3329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3473 1.0185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8920 1.3329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8920 1.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3473 2.2764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8026 1.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2925 -0.4982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8104 -1.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3453 -1.5334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4704 -1.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8194 -0.4639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5173 -0.4639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8662 -1.0683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5173 -1.6727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8194 -1.6727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8658 0.1398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5633 -1.0683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8658 -2.2764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4362 1.0188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4362 0.3904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9792 0.0769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8932 0.0769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9792 -0.5331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5075 -0.8382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0358 -0.5331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0358 0.0769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5075 0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5633 0.3814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5633 -0.8377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5075 -1.4473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4362 2.2761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
1 11 1 0 0 0 0
8 12 1 1 0 0 0
9 13 1 6 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
3 19 1 0 0 0 0
19 20 1 0 0 0 0
20 21 2 0 0 0 0
20 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 22 1 0 0 0 0
24 28 1 0 0 0 0
25 29 1 0 0 0 0
26 30 1 0 0 0 0
15 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
32 34 2 0 0 0 0
33 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 33 1 0 0 0 0
38 40 1 0 0 0 0
37 41 1 0 0 0 0
36 42 1 0 0 0 0
16 43 1 0 0 0 0
S SKP 8
ID FL63ACNS0018
KNApSAcK_ID C00008878
NAME Catechin 5,3'-di-O-gallate
CAS_RN -
FORMULA C29H22O14
EXACTMASS 594.100955412
AVERAGEMASS 594.47658
SMILES O=C(Oc(c25)cc(O)cc2OC(C(O)C5)c(c3)ccc(c3OC(c(c4)cc(O)c(c(O)4)O)=O)O)c(c1)cc(c(c1O)O)O
M END