Mol:FL5FFCGS0021
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 48 52 0 0 0 0 0 0 0 0999 V2000 | + | 48 52 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9860 -0.6657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9860 -0.6657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9860 -1.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9860 -1.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2712 -1.9038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2712 -1.9038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4437 -1.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4437 -1.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4437 -0.6657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4437 -0.6657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2712 -0.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2712 -0.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1585 -1.9038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1585 -1.9038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8733 -1.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8733 -1.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8733 -0.6657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8733 -0.6657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1585 -0.2530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1585 -0.2530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1585 -2.6695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1585 -2.6695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7735 -0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7735 -0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5020 -0.5147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5020 -0.5147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2306 -0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2306 -0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2306 0.7471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2306 0.7471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5020 1.1679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5020 1.1679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7735 0.7471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7735 0.7471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2712 -2.7290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2712 -2.7290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7906 1.0704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7906 1.0704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6219 -0.2530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6219 -0.2530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5234 -1.8664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5234 -1.8664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2712 0.5721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2712 0.5721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0884 -0.2184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0884 -0.2184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6434 -0.8058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6434 -0.8058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0027 -0.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0027 -0.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3350 -0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3350 -0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8337 -0.1006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8337 -0.1006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4882 -0.3356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4882 -0.3356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7906 -0.4070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7906 -0.4070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4008 -0.7387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4008 -0.7387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2851 -1.0078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2851 -1.0078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0231 0.1188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0231 0.1188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3782 -1.7800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3782 -1.7800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7127 -2.3593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7127 -2.3593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0694 -2.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0694 -2.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4223 -2.3593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4223 -2.3593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0879 -1.7800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0879 -1.7800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7311 -1.9638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7311 -1.9638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0097 -1.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0097 -1.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1778 -2.2160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1778 -2.2160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5134 -2.7478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5134 -2.7478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4023 -3.0834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4023 -3.0834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7620 0.6800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7620 0.6800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0441 1.0800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0441 1.0800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4177 0.6104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4177 0.6104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0441 1.7442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0441 1.7442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5020 1.9304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5020 1.9304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0692 3.0834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0692 3.0834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 6 22 1 0 0 0 0 | + | 6 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 37 31 1 0 0 0 0 | + | 37 31 1 0 0 0 0 |
| − | 26 20 1 0 0 0 0 | + | 26 20 1 0 0 0 0 |
| − | 32 43 1 0 0 0 0 | + | 32 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 16 47 1 0 0 0 0 | + | 16 47 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 47 48 | + | M SAL 1 2 47 48 |
| − | M SBL 1 1 52 | + | M SBL 1 1 52 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 52 0.0000 -0.7625 | + | M SBV 1 52 0.0000 -0.7625 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FFCGS0021 | + | ID FL5FFCGS0021 |
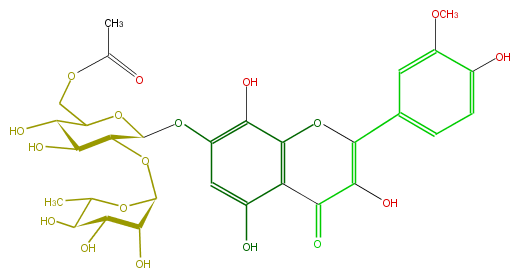
| − | FORMULA C30H34O18 | + | FORMULA C30H34O18 |
| − | EXACTMASS 682.174514284 | + | EXACTMASS 682.174514284 |
| − | AVERAGEMASS 682.58016 | + | AVERAGEMASS 682.58016 |
| − | SMILES c(c31)(O)c(OC(O5)C(C(O)C(C(COC(C)=O)5)O)OC(O4)C(C(O)C(O)C4C)O)cc(c1C(=O)C(=C(O3)c(c2)cc(OC)c(c2)O)O)O | + | SMILES c(c31)(O)c(OC(O5)C(C(O)C(C(COC(C)=O)5)O)OC(O4)C(C(O)C(O)C4C)O)cc(c1C(=O)C(=C(O3)c(c2)cc(OC)c(c2)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
48 52 0 0 0 0 0 0 0 0999 V2000
-0.9860 -0.6657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9860 -1.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2712 -1.9038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4437 -1.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4437 -0.6657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2712 -0.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1585 -1.9038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8733 -1.4910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8733 -0.6657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1585 -0.2530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1585 -2.6695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7735 -0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5020 -0.5147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2306 -0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2306 0.7471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5020 1.1679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7735 0.7471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2712 -2.7290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7906 1.0704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6219 -0.2530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5234 -1.8664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2712 0.5721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0884 -0.2184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6434 -0.8058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0027 -0.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3350 -0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8337 -0.1006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4882 -0.3356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7906 -0.4070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4008 -0.7387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2851 -1.0078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0231 0.1188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3782 -1.7800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7127 -2.3593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0694 -2.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4223 -2.3593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0879 -1.7800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7311 -1.9638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0097 -1.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1778 -2.2160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5134 -2.7478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4023 -3.0834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7620 0.6800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0441 1.0800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4177 0.6104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0441 1.7442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5020 1.9304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0692 3.0834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
4 3 1 0 0 0 0
6 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
28 32 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
35 41 1 0 0 0 0
36 42 1 0 0 0 0
37 31 1 0 0 0 0
26 20 1 0 0 0 0
32 43 1 0 0 0 0
43 44 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
47 48 1 0 0 0 0
16 47 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 47 48
M SBL 1 1 52
M SMT 1 OCH3
M SBV 1 52 0.0000 -0.7625
S SKP 5
ID FL5FFCGS0021
FORMULA C30H34O18
EXACTMASS 682.174514284
AVERAGEMASS 682.58016
SMILES c(c31)(O)c(OC(O5)C(C(O)C(C(COC(C)=O)5)O)OC(O4)C(C(O)C(O)C4C)O)cc(c1C(=O)C(=C(O3)c(c2)cc(OC)c(c2)O)O)O
M END