Mol:FL5FELGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 39 42 0 0 0 0 0 0 0 0999 V2000 | + | 39 42 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1867 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1867 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1867 -0.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1867 -0.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6304 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6304 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0741 -0.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0741 -0.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0741 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0741 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6304 0.4462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6304 0.4462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5178 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5178 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9615 -0.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9615 -0.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9615 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9615 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5178 0.4462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5178 0.4462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5178 -1.3394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5178 -1.3394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4054 0.4461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4054 0.4461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1616 0.1187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1616 0.1187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7286 0.4461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7286 0.4461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7286 1.1008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7286 1.1008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1616 1.4281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1616 1.4281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4054 1.1008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4054 1.1008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6304 -1.4806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6304 -1.4806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5095 0.3614 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.5095 0.3614 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.2087 -0.1596 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.2087 -0.1596 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.7872 0.0057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7872 0.0057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3692 -0.1596 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.3692 -0.1596 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.6701 0.3614 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.6701 0.3614 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.0915 0.1961 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.0915 0.1961 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.9509 0.2117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9509 0.2117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1330 0.6707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1330 0.6707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1041 0.1108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1041 0.1108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2954 0.1188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2954 0.1188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9722 1.4280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9722 1.4280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5397 -0.4810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5397 -0.4810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5397 -1.3061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5397 -1.3061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5042 1.4806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5042 1.4806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2187 1.0681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2187 1.0681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0955 -1.0173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0955 -1.0173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7706 -1.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7706 -1.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9898 0.0732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9898 0.0732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5731 0.6566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5731 0.6566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6627 -0.6929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6627 -0.6929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4473 -0.4380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4473 -0.4380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 14 28 1 0 0 0 0 | + | 14 28 1 0 0 0 0 |
| − | 20 28 1 0 0 0 0 | + | 20 28 1 0 0 0 0 |
| − | 17 29 1 0 0 0 0 | + | 17 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 15 32 1 0 0 0 0 | + | 15 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 8 34 1 0 0 0 0 | + | 8 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 1 36 1 0 0 0 0 | + | 1 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 2 38 1 0 0 0 0 | + | 2 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 30 31 | + | M SAL 5 2 30 31 |
| − | M SBL 5 1 33 | + | M SBL 5 1 33 |
| − | M SMT 5 CH2OH | + | M SMT 5 CH2OH |
| − | M SVB 5 33 3.7328 -0.1727 | + | M SVB 5 33 3.7328 -0.1727 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 38 39 | + | M SAL 4 2 38 39 |
| − | M SBL 4 1 41 | + | M SBL 4 1 41 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 41 -3.6627 -0.6929 | + | M SVB 4 41 -3.6627 -0.6929 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 36 37 | + | M SAL 3 2 36 37 |
| − | M SBL 3 1 39 | + | M SBL 3 1 39 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 39 -3.7426 0.7069 | + | M SVB 3 39 -3.7426 0.7069 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 34 35 | + | M SAL 2 2 34 35 |
| − | M SBL 2 1 37 | + | M SBL 2 1 37 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 37 -0.7626 -0.7236 | + | M SVB 2 37 -0.7626 -0.7236 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 35 1.5042 1.4806 | + | M SVB 1 35 1.5042 1.4806 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FELGS0001 | + | ID FL5FELGS0001 |
| − | KNApSAcK_ID C00005788 | + | KNApSAcK_ID C00005788 |
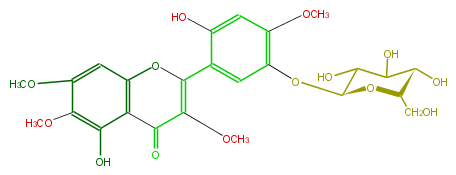
| − | NAME 5,2',5'-Trihydroxy-3,6,7,4'-tetramethoxyflavone 5'-glucoside | + | NAME 5,2',5'-Trihydroxy-3,6,7,4'-tetramethoxyflavone 5'-glucoside |
| − | CAS_RN 71827-15-1 | + | CAS_RN 71827-15-1 |
| − | FORMULA C25H28O14 | + | FORMULA C25H28O14 |
| − | EXACTMASS 552.147905604 | + | EXACTMASS 552.147905604 |
| − | AVERAGEMASS 552.4814200000001 | + | AVERAGEMASS 552.4814200000001 |
| − | SMILES c(c12)(O)c(OC)c(cc1OC(c(c(O)3)cc(O[C@@H](C4O)O[C@@H]([C@H](O)C4O)CO)c(c3)OC)=C(C2=O)OC)OC | + | SMILES c(c12)(O)c(OC)c(cc1OC(c(c(O)3)cc(O[C@@H](C4O)O[C@@H]([C@H](O)C4O)CO)c(c3)OC)=C(C2=O)OC)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
39 42 0 0 0 0 0 0 0 0999 V2000
-3.1867 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1867 -0.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6304 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0741 -0.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0741 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6304 0.4462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5178 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9615 -0.5173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9615 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5178 0.4462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5178 -1.3394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4054 0.4461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1616 0.1187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7286 0.4461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7286 1.1008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1616 1.4281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4054 1.1008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6304 -1.4806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5095 0.3614 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.2087 -0.1596 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.7872 0.0057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3692 -0.1596 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.6701 0.3614 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.0915 0.1961 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.9509 0.2117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1330 0.6707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1041 0.1108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2954 0.1188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9722 1.4280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5397 -0.4810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5397 -1.3061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5042 1.4806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2187 1.0681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0955 -1.0173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7706 -1.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9898 0.0732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5731 0.6566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6627 -0.6929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4473 -0.4380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
24 26 1 0 0 0 0
23 27 1 0 0 0 0
14 28 1 0 0 0 0
20 28 1 0 0 0 0
17 29 1 0 0 0 0
22 30 1 0 0 0 0
30 31 1 0 0 0 0
15 32 1 0 0 0 0
32 33 1 0 0 0 0
8 34 1 0 0 0 0
34 35 1 0 0 0 0
1 36 1 0 0 0 0
36 37 1 0 0 0 0
2 38 1 0 0 0 0
38 39 1 0 0 0 0
M STY 1 5 SUP
M SLB 1 5 5
M SAL 5 2 30 31
M SBL 5 1 33
M SMT 5 CH2OH
M SVB 5 33 3.7328 -0.1727
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 38 39
M SBL 4 1 41
M SMT 4 OCH3
M SVB 4 41 -3.6627 -0.6929
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 36 37
M SBL 3 1 39
M SMT 3 OCH3
M SVB 3 39 -3.7426 0.7069
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 34 35
M SBL 2 1 37
M SMT 2 OCH3
M SVB 2 37 -0.7626 -0.7236
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 35
M SMT 1 OCH3
M SVB 1 35 1.5042 1.4806
S SKP 8
ID FL5FELGS0001
KNApSAcK_ID C00005788
NAME 5,2',5'-Trihydroxy-3,6,7,4'-tetramethoxyflavone 5'-glucoside
CAS_RN 71827-15-1
FORMULA C25H28O14
EXACTMASS 552.147905604
AVERAGEMASS 552.4814200000001
SMILES c(c12)(O)c(OC)c(cc1OC(c(c(O)3)cc(O[C@@H](C4O)O[C@@H]([C@H](O)C4O)CO)c(c3)OC)=C(C2=O)OC)OC
M END