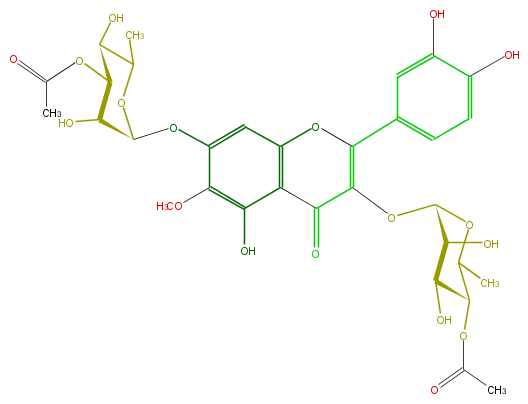
Mol:FL5FECGS0047
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 50 54 0 0 0 0 0 0 0 0999 V2000 | + | 50 54 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.0444 1.0877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0444 1.0877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0444 0.2626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0444 0.2626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3299 -0.1498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3299 -0.1498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3846 0.2626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3846 0.2626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3846 1.0877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3846 1.0877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3299 1.5003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3299 1.5003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0991 -0.1498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0991 -0.1498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8136 0.2626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8136 0.2626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8136 1.0877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8136 1.0877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0991 1.5003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0991 1.5003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0991 -1.0365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0991 -1.0365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7132 1.6590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7132 1.6590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4415 1.2386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4415 1.2386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1698 1.6590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1698 1.6590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1698 2.4999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1698 2.4999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4415 2.9203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4415 2.9203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7132 2.4999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7132 2.4999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3299 -0.9747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3299 -0.9747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9491 2.9499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9491 2.9499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7539 1.4830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7539 1.4830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6186 -0.3113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6186 -0.3113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4794 -1.5987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4794 -1.5987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1671 -1.9957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1671 -1.9957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9489 -1.2322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9489 -1.2322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1671 -0.4641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1671 -0.4641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4794 -0.0669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4794 -0.0669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6975 -0.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6975 -0.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5407 -0.8305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5407 -0.8305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5955 -2.3624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5955 -2.3624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0431 -2.6846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0431 -2.6846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4642 -1.6255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4642 -1.6255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2408 1.6236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2408 1.6236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5662 1.2341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5662 1.2341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7802 1.9831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7802 1.9831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5662 2.7366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5662 2.7366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2408 3.1261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2408 3.1261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0268 2.3772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0268 2.3772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9741 3.6789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9741 3.6789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6473 3.3450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6473 3.3450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6308 2.8257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6308 2.8257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8299 1.5727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8299 1.5727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4415 3.7610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4415 3.7610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3088 2.3912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3088 2.3912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9491 2.8522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9491 2.8522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3048 1.7780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3048 1.7780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0441 -3.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0441 -3.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4707 -3.7610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4707 -3.7610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6005 -3.7558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6005 -3.7558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6494 -0.0867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6494 -0.0867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7317 -0.6165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7317 -0.6165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 32 1 1 0 0 0 | + | 37 32 1 1 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | 20 33 1 0 0 0 0 | + | 20 33 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 30 46 1 0 0 0 0 | + | 30 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 46 48 1 0 0 0 0 | + | 46 48 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 2 49 1 0 0 0 0 | + | 2 49 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 49 50 | + | M SAL 1 2 49 50 |
| − | M SBL 1 1 54 | + | M SBL 1 1 54 |
| − | M SMT 1 ^OCH3 | + | M SMT 1 ^OCH3 |
| − | M SBV 1 54 0.6050 0.3493 | + | M SBV 1 54 0.6050 0.3493 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FECGS0047 | + | ID FL5FECGS0047 |
| − | FORMULA C32H36O18 | + | FORMULA C32H36O18 |
| − | EXACTMASS 708.190164348 | + | EXACTMASS 708.190164348 |
| − | AVERAGEMASS 708.61744 | + | AVERAGEMASS 708.61744 |
| − | SMILES Oc(c5)c(cc(c5)C(=C1OC(O4)C(C(O)C(OC(C)=O)C4C)O)Oc(c2)c(c(O)c(OC)c2OC(C3O)OC(C)C(C3OC(C)=O)O)C1=O)O | + | SMILES Oc(c5)c(cc(c5)C(=C1OC(O4)C(C(O)C(OC(C)=O)C4C)O)Oc(c2)c(c(O)c(OC)c2OC(C3O)OC(C)C(C3OC(C)=O)O)C1=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
50 54 0 0 0 0 0 0 0 0999 V2000
-1.0444 1.0877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0444 0.2626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3299 -0.1498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3846 0.2626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3846 1.0877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3299 1.5003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0991 -0.1498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8136 0.2626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8136 1.0877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0991 1.5003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0991 -1.0365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7132 1.6590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4415 1.2386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1698 1.6590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1698 2.4999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4415 2.9203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7132 2.4999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3299 -0.9747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9491 2.9499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7539 1.4830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6186 -0.3113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4794 -1.5987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1671 -1.9957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9489 -1.2322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1671 -0.4641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4794 -0.0669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6975 -0.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5407 -0.8305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5955 -2.3624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0431 -2.6846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4642 -1.6255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2408 1.6236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5662 1.2341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7802 1.9831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5662 2.7366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2408 3.1261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0268 2.3772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9741 3.6789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6473 3.3450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6308 2.8257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8299 1.5727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4415 3.7610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3088 2.3912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9491 2.8522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3048 1.7780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0441 -3.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4707 -3.7610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6005 -3.7558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6494 -0.0867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7317 -0.6165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
26 21 1 0 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 32 1 1 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
37 40 1 0 0 0 0
32 41 1 0 0 0 0
20 33 1 0 0 0 0
16 42 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
40 43 1 0 0 0 0
30 46 1 0 0 0 0
46 47 2 0 0 0 0
46 48 1 0 0 0 0
49 50 1 0 0 0 0
2 49 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 49 50
M SBL 1 1 54
M SMT 1 ^OCH3
M SBV 1 54 0.6050 0.3493
S SKP 5
ID FL5FECGS0047
FORMULA C32H36O18
EXACTMASS 708.190164348
AVERAGEMASS 708.61744
SMILES Oc(c5)c(cc(c5)C(=C1OC(O4)C(C(O)C(OC(C)=O)C4C)O)Oc(c2)c(c(O)c(OC)c2OC(C3O)OC(C)C(C3OC(C)=O)O)C1=O)O
M END