Mol:FL5FECGS0023
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 37 40 0 0 0 0 0 0 0 0999 V2000 | + | 37 40 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.2844 -0.2743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2844 -0.2743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2844 -0.9167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2844 -0.9167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7281 -1.2379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7281 -1.2379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1718 -0.9167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1718 -0.9167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1718 -0.2743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1718 -0.2743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7281 0.0469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7281 0.0469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6155 -1.2379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6155 -1.2379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0592 -0.9167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0592 -0.9167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0592 -0.2743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0592 -0.2743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6155 0.0469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6155 0.0469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6155 -1.7387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6155 -1.7387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5031 0.0468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5031 0.0468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0638 -0.2806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0638 -0.2806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6308 0.0468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6308 0.0468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6308 0.7014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6308 0.7014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0638 1.0288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0638 1.0288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5031 0.7014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5031 0.7014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7281 -1.8800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7281 -1.8800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0638 1.6833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0638 1.6833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1128 1.2313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1128 1.2313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0612 1.6327 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.0612 1.6327 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.7604 1.1117 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.7604 1.1117 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.3388 1.2770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3388 1.2770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9208 1.1117 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.9208 1.1117 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.2217 1.6327 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.2217 1.6327 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.6432 1.4674 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.6432 1.4674 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.5025 1.4830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5025 1.4830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6847 1.9420 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6847 1.9420 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6557 1.3821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6557 1.3821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6417 0.3444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6417 0.3444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1418 1.2104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1418 1.2104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9722 -0.7025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9722 -0.7025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7941 -0.7746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7941 -0.7746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1932 -1.4167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1932 -1.4167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6728 -1.9167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6728 -1.9167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0913 0.7903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0913 0.7903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0913 -0.0348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0913 -0.0348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 20 22 1 0 0 0 0 | + | 20 22 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 2 32 1 0 0 0 0 | + | 2 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 8 34 1 0 0 0 0 | + | 8 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 24 36 1 0 0 0 0 | + | 24 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 36 37 | + | M SAL 4 2 36 37 |
| − | M SBL 4 1 39 | + | M SBL 4 1 39 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 39 3.2844 1.0986 | + | M SVB 4 39 3.2844 1.0986 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 34 35 | + | M SAL 3 2 34 35 |
| − | M SBL 3 1 37 | + | M SBL 3 1 37 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 37 -0.8604 -1.1229 | + | M SVB 3 37 -0.8604 -1.1229 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 32 33 | + | M SAL 2 2 32 33 |
| − | M SBL 2 1 35 | + | M SBL 2 1 35 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 35 -3.9989 -0.8252 | + | M SVB 2 35 -3.9989 -0.8252 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 30 31 | + | M SAL 1 2 30 31 |
| − | M SBL 1 1 33 | + | M SBL 1 1 33 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 33 -3.6417 0.3444 | + | M SVB 1 33 -3.6417 0.3444 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FECGS0023 | + | ID FL5FECGS0023 |
| − | KNApSAcK_ID C00005669 | + | KNApSAcK_ID C00005669 |
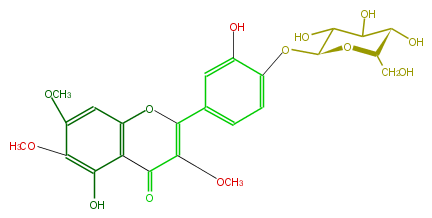
| − | NAME Chrysosplenoside D | + | NAME Chrysosplenoside D |
| − | CAS_RN 25321-00-0 | + | CAS_RN 25321-00-0 |
| − | FORMULA C24H26O13 | + | FORMULA C24H26O13 |
| − | EXACTMASS 522.137340918 | + | EXACTMASS 522.137340918 |
| − | AVERAGEMASS 522.45544 | + | AVERAGEMASS 522.45544 |
| − | SMILES O(C)c(c4OC)c(O)c(c3c4)C(C(OC)=C(O3)c(c1)ccc(O[C@@H](C2O)O[C@@H]([C@@H](C2O)O)CO)c1O)=O | + | SMILES O(C)c(c4OC)c(O)c(c3c4)C(C(OC)=C(O3)c(c1)ccc(O[C@@H](C2O)O[C@@H]([C@@H](C2O)O)CO)c1O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
37 40 0 0 0 0 0 0 0 0999 V2000
-3.2844 -0.2743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2844 -0.9167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7281 -1.2379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1718 -0.9167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1718 -0.2743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7281 0.0469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6155 -1.2379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0592 -0.9167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0592 -0.2743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6155 0.0469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6155 -1.7387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5031 0.0468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0638 -0.2806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6308 0.0468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6308 0.7014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0638 1.0288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5031 0.7014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7281 -1.8800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0638 1.6833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1128 1.2313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0612 1.6327 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.7604 1.1117 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.3388 1.2770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9208 1.1117 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.2217 1.6327 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.6432 1.4674 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.5025 1.4830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6847 1.9420 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6557 1.3821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6417 0.3444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1418 1.2104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9722 -0.7025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7941 -0.7746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1932 -1.4167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6728 -1.9167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0913 0.7903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0913 -0.0348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
16 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
20 22 1 0 0 0 0
1 30 1 0 0 0 0
30 31 1 0 0 0 0
2 32 1 0 0 0 0
32 33 1 0 0 0 0
8 34 1 0 0 0 0
34 35 1 0 0 0 0
24 36 1 0 0 0 0
36 37 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 36 37
M SBL 4 1 39
M SMT 4 CH2OH
M SVB 4 39 3.2844 1.0986
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 34 35
M SBL 3 1 37
M SMT 3 OCH3
M SVB 3 37 -0.8604 -1.1229
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 32 33
M SBL 2 1 35
M SMT 2 OCH3
M SVB 2 35 -3.9989 -0.8252
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 30 31
M SBL 1 1 33
M SMT 1 OCH3
M SVB 1 33 -3.6417 0.3444
S SKP 8
ID FL5FECGS0023
KNApSAcK_ID C00005669
NAME Chrysosplenoside D
CAS_RN 25321-00-0
FORMULA C24H26O13
EXACTMASS 522.137340918
AVERAGEMASS 522.45544
SMILES O(C)c(c4OC)c(O)c(c3c4)C(C(OC)=C(O3)c(c1)ccc(O[C@@H](C2O)O[C@@H]([C@@H](C2O)O)CO)c1O)=O
M END